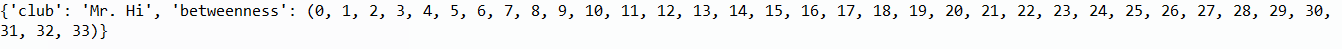Установка узлов в графе networkx как инфицированных и незараженных
У меня есть граф клуба карате, я хочу определить несколько случайных узлов внутри него как зараженные узлы, а остальные не инфицированы. Я пробовал использовать:
G = nx.karate_club_graph()
#pos = nx.spectral_layout(G)
bb = tuple(nx.betweenness_centrality(G))
nx.set_node_attributes(G, 'betweenness', bb)
G.nodes[1]['betweenness']
В качестве примера, чтобы увидеть, может ли это сработать. Хотя он возвращает ключевую ошибку «промежуточность» в последней строке. Какие существуют способы вручную или случайным образом выбрать несколько узлов на графе и присвоить им 1 для зараженных, в то время как остальные равны 0. Или есть лучший способ установить инфекции в этом графе?
1 ответ
просто следуйте правильному вызову функции для set_node_attributes
Правильный синтаксис:
set_node_attributes(G, values, name=None)
так что ваша переменная bb будет стоять перед промежуточностью имен
код:
import networkx as nx
G = nx.karate_club_graph()
bb = tuple(nx.betweenness_centrality(G))
# changed
nx.set_node_attributes(G, bb, 'betweenness')
print(G.nodes[1])
Результат:
Также, если вы удалите часть кортежа, вы получите результат:
import networkx as nx
G = nx.karate_club_graph()
bb = tuple(nx.betweenness_centrality(G))
# changed
bb = nx.betweenness_centrality(G)
nx.set_node_attributes(G, bb, 'betweenness')
print(G.nodes[1])
Результат: