Как извлечь фиксированные эффекты с помощью наблюдения?
У меня есть объект Ime, построенный на основе данных повторного измерения потребления питательных веществ (два 24-часовых периода потребления для каждого RespondentID):
Male.lme2 <- lmer(BoxCoxXY ~ -1 + AgeFactor + IntakeDay + (1|RespondentID),
data = Male.Data,
weights = SampleWeight)
и я могу успешно получить случайные эффекты с помощью RespondentID с помощью ranef(Male.lme1), Я также хотел бы собрать результат фиксированных эффектов RespondentID, coef(Male.lme1) не предоставляет именно то, что мне нужно, как я покажу ниже.
> summary(Male.lme1)
Linear mixed model fit by REML
Formula: BoxCoxXY ~ AgeFactor + IntakeDay + (1 | RespondentID)
Data: Male.Data
AIC BIC logLik deviance REMLdev
9994 10039 -4990 9952 9980
Random effects:
Groups Name Variance Std.Dev.
RespondentID (Intercept) 0.19408 0.44055
Residual 0.37491 0.61230
Number of obs: 4498, groups: RespondentID, 2249
Fixed effects:
Estimate Std. Error t value
(Intercept) 13.98016 0.03405 410.6
AgeFactor4to8 0.50572 0.04084 12.4
AgeFactor9to13 0.94329 0.04159 22.7
AgeFactor14to18 1.30654 0.04312 30.3
IntakeDayDay2Intake -0.13871 0.01809 -7.7
Correlation of Fixed Effects:
(Intr) AgFc48 AgF913 AF1418
AgeFactr4t8 -0.775
AgeFctr9t13 -0.761 0.634
AgFctr14t18 -0.734 0.612 0.601
IntkDyDy2In -0.266 0.000 0.000 0.000
Я добавил результаты к моим данным, head(Male.Data) шоу
NutrientID RespondentID Gender Age SampleWeight IntakeDay IntakeAmt AgeFactor BoxCoxXY lmefits
2 267 100020 1 12 0.4952835 Day1Intake 12145.852 9to13 15.61196 15.22633
7 267 100419 1 14 0.3632839 Day1Intake 9591.953 14to18 15.01444 15.31373
8 267 100459 1 11 0.4952835 Day1Intake 7838.713 9to13 14.51458 15.00062
12 267 101138 1 15 1.3258785 Day1Intake 11113.266 14to18 15.38541 15.75337
14 267 101214 1 6 2.1198688 Day1Intake 7150.133 4to8 14.29022 14.32658
18 267 101389 1 5 2.1198688 Day1Intake 5091.528 4to8 13.47928 14.58117
Первая пара строк из coef(Male.lme1) являются:
$RespondentID
(Intercept) AgeFactor4to8 AgeFactor9to13 AgeFactor14to18 IntakeDayDay2Intake
100020 14.28304 0.5057221 0.9432941 1.306542 -0.1387098
100419 14.00719 0.5057221 0.9432941 1.306542 -0.1387098
100459 14.05732 0.5057221 0.9432941 1.306542 -0.1387098
101138 14.44682 0.5057221 0.9432941 1.306542 -0.1387098
101214 13.82086 0.5057221 0.9432941 1.306542 -0.1387098
101389 14.07545 0.5057221 0.9432941 1.306542 -0.1387098
Чтобы продемонстрировать, как coef результаты относятся к подобранным оценкам в Male.Data (которые были получены с использованием Male.Data$lmefits <- fitted(Male.lme1)для первого RespondentID, который имеет уровень AgeFactor 9-13:
- установленное значение 15.22633, который равен - из коэффи- (Intercept) + (AgeFactor9-13) = 14.28304 + 0.9432941
Есть ли для меня умная команда для использования, которая автоматически захочет, что я хочу, то есть для извлечения фиксированной оценки эффекта для каждого субъекта, или я столкнулся с рядом if утверждения, пытающиеся применить правильный уровень AgeFactor к каждому субъекту, чтобы получить правильную оценку фиксированного эффекта, после вычета вклада случайного эффекта из Перехвата?
Обновление, извиняюсь, пытался сократить вывод, который я предоставлял, и забыл про str(). Выход:
>str(Male.Data)
'data.frame': 4498 obs. of 11 variables:
$ NutrientID : int 267 267 267 267 267 267 267 267 267 267 ...
$ RespondentID: Factor w/ 2249 levels "100020","100419",..: 1 2 3 4 5 6 7 8 9 10 ...
$ Gender : int 1 1 1 1 1 1 1 1 1 1 ...
$ Age : int 12 14 11 15 6 5 10 2 2 9 ...
$ BodyWeight : num 51.6 46.3 46.1 63.2 28.4 18 38.2 14.4 14.6 32.1 ...
$ SampleWeight: num 0.495 0.363 0.495 1.326 2.12 ...
$ IntakeDay : Factor w/ 2 levels "Day1Intake","Day2Intake": 1 1 1 1 1 1 1 1 1 1 ...
$ IntakeAmt : num 12146 9592 7839 11113 7150 ...
$ AgeFactor : Factor w/ 4 levels "1to3","4to8",..: 3 4 3 4 2 2 3 1 1 3 ...
$ BoxCoxXY : num 15.6 15 14.5 15.4 14.3 ...
$ lmefits : num 15.2 15.3 15 15.8 14.3 ...
BodyWeight и Gender не используются (это данные о мужчинах, поэтому все значения Gender одинаковы), и NutrientID аналогичным образом фиксируется для данных.
Я делал ужасные заявления ifelse, которые я опубликовал, поэтому немедленно опробую ваше предложение.:)
Обновление 2: это прекрасно работает с моими текущими данными и должно быть ориентировано на будущее для новых данных, благодаря DWin за дополнительную помощь в комментарии к этому.:)
AgeLevels <- length(unique(Male.Data$AgeFactor))
Temp <- as.data.frame(fixef(Male.lme1)['(Intercept)'] +
c(0,fixef(Male.lme1)[2:AgeLevels])[
match(Male.Data$AgeFactor, c("1to3", "4to8", "9to13","14to18", "19to30","31to50","51to70","71Plus") )] +
c(0,fixef(Male.lme1)[(AgeLevels+1)])[
match(Male.Data$IntakeDay, c("Day1Intake","Day2Intake") )])
names(Temp) <- c("FxdEffct")
2 ответа
Это будет что-то вроде этого (хотя вы действительно должны были дать нам результаты str(Male.Data), потому что выходные данные модели не сообщают нам уровни факторов для базовых значений:)
#First look at the coefficients
fixef(Male.lme2)
#Then do the calculations
fixef(Male.lme2)[`(Intercept)`] +
c(0,fixef(Male.lme2)[2:4])[
match(Male.Data$AgeFactor, c("1to3", "4to8", "9to13","14to18") )] +
c(0,fixef(Male.lme2)[5])[
match(Male.Data$IntakeDay, c("Day1Intake","Day2Intake") )]
Вы в основном запускаете исходные данные через match функция для выбора правильных коэффициентов для добавления в точку пересечения... которая будет равна 0, если данные являются базовым уровнем фактора (написание которого я предполагаю.)
РЕДАКТИРОВАТЬ: Я только что заметил, что вы положили "-1" в формулу, так что, возможно, все ваши термины AgeFactor перечислены в выходных данных, и вы можете указать 0 в векторе коэффициентов и изобретенный уровень AgeFactor в векторе таблицы соответствий.
Ниже я всегда находил, что проще всего извлечь отдельные фиксированные эффекты и компоненты случайных эффектов из пакета lme4. Это фактически извлекает соответствующую подгонку к каждому наблюдению. Предполагая, что у нас есть модель формы со смешанными эффектами:
y = Xb + Zu + e
где Xb - фиксированные эффекты, а Zu - случайные эффекты, мы можем извлечь компоненты (например, из режима сна lme4):
library(lme4)
fm1 <- lmer(Reaction ~ Days + (Days|Subject), sleepstudy)
# Xb
fix <- getME(fm1,'X') %*% fixef(fm1)
# Zu
ran <- t(as.matrix(getME(fm1,'Zt'))) %*% unlist(ranef(fm1))
# Xb + Zu
fixran <- fix + ran
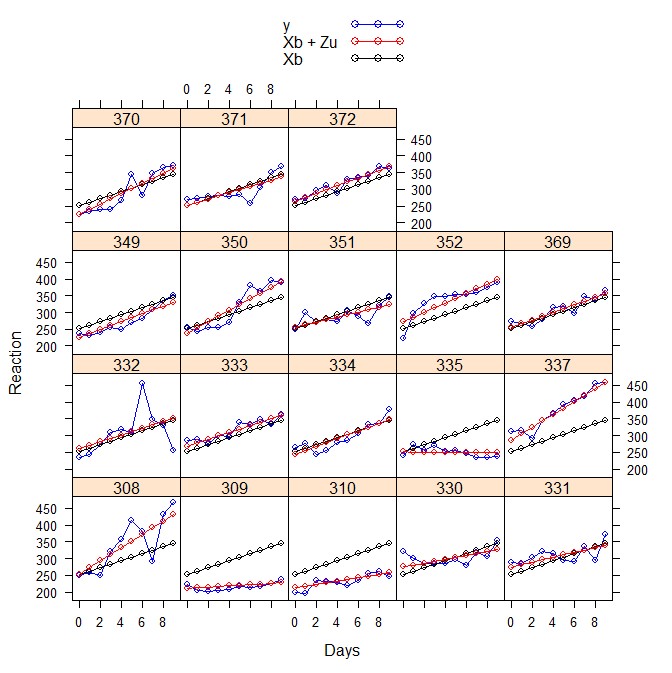
Я знаю, что это работает как обобщенный подход к выделению компонентов из линейных моделей смешанных эффектов. Для нелинейных моделей матрица модели X содержит повторы, и вам, возможно, придется немного адаптировать вышеуказанный код. Вот некоторые результаты проверки, а также визуализация с использованием решетки:
> head(cbind(fix, ran, fixran, fitted(fm1)))
[,1] [,2] [,3] [,4]
[1,] 251.4051 2.257187 253.6623 253.6623
[2,] 261.8724 11.456439 273.3288 273.3288
[3,] 272.3397 20.655691 292.9954 292.9954
[4,] 282.8070 29.854944 312.6619 312.6619
[5,] 293.2742 39.054196 332.3284 332.3284
[6,] 303.7415 48.253449 351.9950 351.9950
# Xb + Zu
> all(round((fixran),6) == round(fitted(fm1),6))
[1] TRUE
# e = y - (Xb + Zu)
> all(round(resid(fm1),6) == round(sleepstudy[,"Reaction"]-(fixran),6))
[1] TRUE
nobs <- 10 # 10 observations per subject
legend = list(text=list(c("y", "Xb + Zu", "Xb")), lines = list(col=c("blue", "red", "black"), pch=c(1,1,1), lwd=c(1,1,1), type=c("b","b","b")))
require(lattice)
xyplot(
Reaction ~ Days | Subject, data = sleepstudy,
panel = function(x, y, ...){
panel.points(x, y, type='b', col='blue')
panel.points(x, fix[(1+nobs*(panel.number()-1)):(nobs*(panel.number()))], type='b', col='black')
panel.points(x, fixran[(1+nobs*(panel.number()-1)):(nobs*(panel.number()))], type='b', col='red')
},
key = legend
)