Построение довольно сложной диаграммы, используя R и ось break()
Привет R пользователи и программисты, у меня есть набор данных, состоящий из 4563 аминокислот белка. При использовании трех разных обработок и двух разных окислителей аминокислоты в этом белке были окислены. Я хотел бы изобразить положение этих окислений в диаграмме, основанной на лечении. Различный размер линии будет представлять различные концентрации окислителя, а тип линии (пунктирная и сплошная) будет представлять различные типы окислителя. Я хотел бы разбить ось на каждую 1000 аминокислот. Я создал аналогичный шаблон с Excel и GIMP (что довольно много времени и, возможно, неуместно!). 0,33 в шаблоне это высота строки. http://dl.dropbox.com/u/58221687/Chakraborty_Figure1.png. Вот набор данных: http://dl.dropbox.com/u/58221687/AA-Position-template.xls
Заранее спасибо. Sourav
1 ответ
Я сделаю это в базовой графике, хотя я уверен, что другие могут сделать то же самое или лучше в решетке или ggplot2. Я думаю, что главное, что вам нужно сделать, чтобы легко создать такой график с вашими данными, это изменить форму и переосмыслить, в каком формате должны быть данные, чтобы их можно было наносить на график. Я сделал бы это, используя ваши данные, если бы 1) они были в длинном формате и 2) переменные, на которых вы основали цвет, тип линии, ширину и т. Д., Были доступны в качестве дополнительных столбцов. Если бы у вас были такие данные, то вы могли бы сократить их, включив в них только те аминокислоты, для которых нужно нарисовать отрезки. Я смоделировал набор данных, похожий на ваш. Вы должны быть в состоянии изменить этот код, чтобы он соответствовал вашему случаю. Сначала набор данных:
set.seed(1)
# make data.frame just with info for the lines you'll actually draw
# your data was mostly zeros, no need for those lines
position <- sort(sample(1:4563,45,replace = FALSE))
# but the x position needs to be shaved down!
# modulars are the real x positions on the plot:
xpos <- position%%600
# line direction appeared in your example but not in your text
posorneg <- sample(c(-1,1),45,replace = TRUE,prob=c(.05,.95))
# oxidant concentration for line width- just rescale the oxidant concentration
# values you have to fall between say .5 and 3, or whatever is nice and visible
oxconc <- (.5+runif(45))^2
# oxidant type determines line type- you mention 2
# just assign these types to lines types (integers in R)
oxitype <- sample(c(1,2),45,replace = TRUE)
# let's say there's another dimension you want to map color to
# in your example png, but not in your description.
color <- sample(c("green","black","blue"),45,replace=TRUE)
# and finally, which level does each segment need to belong to?
# you have 8 line levels in your example png. This works, might take
# some staring though:
level <- 0
for (i in 0:7){
level[position %in% ((i*600):(i*600+599))] <- 8-i
}
# now stick into data.drame:
AminoData <-data.frame(position = position, xpos = xpos, posorneg = posorneg,
oxconc = oxconc, oxitype = oxitype, level = level, color = color)
Итак, представьте, что вы можете сократить ваши данные до чего-то такого простого. Вашим основным инструментом при построении (в основании) будут сегменты (). Это векторизация, так что нет необходимости в зацикливании или фантазии:
# now we draw the base plot:
par(mar=c(3,3,3,3))
plot(NULL, type = "n", axes = FALSE, xlab = "", ylab = "",
ylim = c(0,9), xlim = c(-10,609))
# horizontal segments:
segments(0,1:8,599,1:8,gray(.5))
# some ticks: (also not pretty)
segments(rep(c((0:5)*100,599),8), rep(1:8,each=7)-.05, rep(c((0:5)*100,599),8),
rep(1:8,each=7)+.05, col=gray(.5))
# label endpoints:
text(rep(10,8)+.2,1:8-.2,(7:0)*600,pos=2,cex=.8)
text(rep(589,8)+.2,1:8-.2,(7:0)*600+599,pos=4,cex=.8)
# now the amino line segments, remember segments() is vectorized
segments(AminoData$xpos, AminoData$level, AminoData$xpos,
AminoData$level + .5 * AminoData$posorneg, lty = AminoData$oxitype,
lwd = AminoData$oxconc, col = as.character(AminoData$color))
title("mostly you just need to reshape and prepare\nyour data to do this easily in base")
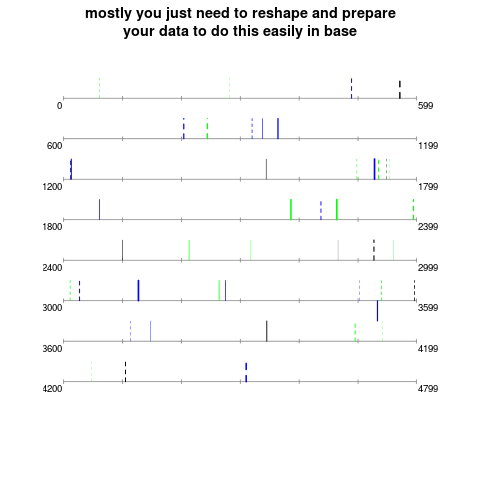
Это может быть слишком кустарным для вкуса некоторых, но это способ, которым я делаю специальные заговоры.