Расчетный Robustscaler в sklearn кажется неправильным
Я попробовал Robustscaler в sklearn и обнаружил, что результаты не совпадают с формулой.
Формула робустскалера в sklearn:
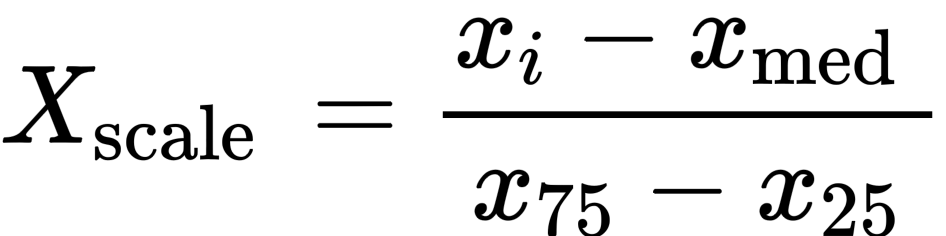
У меня есть матрица, показанная ниже:

Я тестирую первые данные в первой функции (первая строка и первый столбец). Масштабируемое значение должно быть
(1-3)/(5.5-1.5) = -0.5. Однако результат sklearn
-0.67. Кто-нибудь знает, где расчет не верный?
Код, использующий sklearn, выглядит следующим образом:
import numpy as np
from sklearn.preprocessing import RobustScaler
x=[[1,2,3,4],[4,5,6,7],[7,8,9,10],[2,1,1,1]]
scaler = RobustScaler(quantile_range=(25.0, 75.0),with_centering=True)
x_new = scaler.fit_transform(x)
print(x_new)
1 ответ
Из документации RobustScaler (выделено мной):
Центрирование и масштабирование происходят независимо для каждой функции путем вычисления соответствующей статистики по выборкам в обучающем наборе.
т.е. медиана и величины IQR рассчитываются для каждого столбца, а не для всего массива.
Выяснив это, давайте вычислим масштабированные значения для вашего первого столбца вручную:
import numpy as np
x1 = np.array([1, 4, 7, 2]) # your 1st column here
q75, q25 = np.percentile(x1, [75 ,25])
iqr = q75 - q25
x1_med = np.median(x1)
x1_scaled = (x1-x1_med)/iqr
x1_scaled
# array([-0.66666667, 0.33333333, 1.33333333, -0.33333333])
что то же самое с первым столбцом вашего собственного
x_new, по расчетам scikit-learn:
# your code verbatim:
from sklearn.preprocessing import RobustScaler
x=[[1,2,3,4],[4,5,6,7],[7,8,9,10],[2,1,1,1]]
scaler = RobustScaler(quantile_range=(25.0, 75.0),with_centering=True)
x_new = scaler.fit_transform(x)
print(x_new)
# result
[[-0.66666667 -0.375 -0.35294118 -0.33333333]
[ 0.33333333 0.375 0.35294118 0.33333333]
[ 1.33333333 1.125 1.05882353 1. ]
[-0.33333333 -0.625 -0.82352941 -1. ]]
np.all(x1_scaled == x_new[:,0])
# True
Аналогично для остальных столбцов (функций) - вам необходимо отдельно рассчитать медианные значения и значения IQR для каждого из них перед их масштабированием.
ОБНОВЛЕНИЕ (после комментария):
Как указано в статье Википедии о квартилях :
Для дискретных распределений нет единого мнения о выборе значений квартилей.
См. Также соответствующую ссылку, <em>Выборочные квантили в статистических пакетах</em> :
В статистических компьютерных пакетах для квантилей выборки используется большое количество различных определений.
Копаясь в документации используется здесь, вы увидите, что существует не менее пяти (5) различных методов интерполяции, и не все из них дают одинаковые результаты (см. также 4 разных метода, продемонстрированных в статье Википедии, ссылка на которую приведена выше); вот быстрая демонстрация этих методов и их результатов в
x1 данные, определенные выше:
np.percentile(x1, [75 ,25]) # interpolation='linear' by default
# array([4.75, 1.75])
np.percentile(x1, [75 ,25], interpolation='lower')
# array([4, 1])
np.percentile(x1, [75 ,25], interpolation='higher')
# array([7, 2])
np.percentile(x1, [75 ,25], interpolation='midpoint')
# array([5.5, 1.5])
np.percentile(x1, [75 ,25], interpolation='nearest')
# array([4, 2])
Помимо того факта, что не существует двух методов, дающих одинаковые результаты, также должно быть очевидно, что определение, которое вы используете в своих расчетах, соответствует
interpolation='midpoint', а метод Numpy по умолчанию -. И, как правильно указывает Бен Рейнигер в комментариях ниже, в исходном коде RobustScaler фактически используется
<tcode id="64861"></tcode> (вариант pf
np.percentile Я использовал здесь, что может обрабатывать
nan значения) со значением по умолчанию
interpolation='linear' настройка.