Преобразование 2D-массива numpy в файл STL
Я использую
[face-alignment][1]модуль для получения двумерных точек лица, очень похожий на детектор ориентиров лица DLIB, но на этот раз с Z-Index. Я хочу преобразовать или сохранить этот 2D-массив в файл STL. Я использую
numpy-stlоднако чтобы преобразовать это из их документации, всякий раз, когда я пытаюсь написать сетку, она всегда выдает ошибки. Ниже мой код и ошибка. Любые предложения помимо
numpy-stl также приветствуются.
import face_alignment
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from skimage import io
import collections
import numpy as np
from stl import mesh
# Run the 3D face alignment on a test image, without CUDA.
fa = face_alignment.FaceAlignment(face_alignment.LandmarksType._3D, device='cpu', flip_input=True)
try:
input_img = io.imread('/home/ubuntu/desOpencv/srkface.jpg')
except FileNotFoundError:
input_img = io.imread('test/assets/aflw-test.jpg')
preds = fa.get_landmarks(input_img)[-1]
vertices = np.array([\
[-1, -1, -1],
[+1, -1, -1],
[+1, +1, -1],
[-1, +1, -1],
[-1, -1, +1],
[+1, -1, +1],
[+1, +1, +1],
[-1, +1, +1]])
cube = mesh.Mesh(np.zeros(preds.shape[0], dtype=mesh.Mesh.dtype))
for i, f in enumerate(preds):
for j in range(3):
cube.vectors[i][j] = vertices[f[j],:]
# Write the mesh to file "cube.stl"
cube.save('cube.stl')
# 2D-Plot
plot_style = dict(marker='o',
markersize=4,
linestyle='-',
lw=2)
pred_type = collections.namedtuple('prediction_type', ['slice', 'color'])
pred_types = {'face': pred_type(slice(0, 17), (0.682, 0.780, 0.909, 0.5)),
'eyebrow1': pred_type(slice(17, 22), (1.0, 0.498, 0.055, 0.4)),
'eyebrow2': pred_type(slice(22, 27), (1.0, 0.498, 0.055, 0.4)),
'nose': pred_type(slice(27, 31), (0.345, 0.239, 0.443, 0.4)),
'nostril': pred_type(slice(31, 36), (0.345, 0.239, 0.443, 0.4)),
'eye1': pred_type(slice(36, 42), (0.596, 0.875, 0.541, 0.3)),
'eye2': pred_type(slice(42, 48), (0.596, 0.875, 0.541, 0.3)),
'lips': pred_type(slice(48, 60), (0.596, 0.875, 0.541, 0.3)),
'teeth': pred_type(slice(60, 68), (0.596, 0.875, 0.541, 0.4))
}
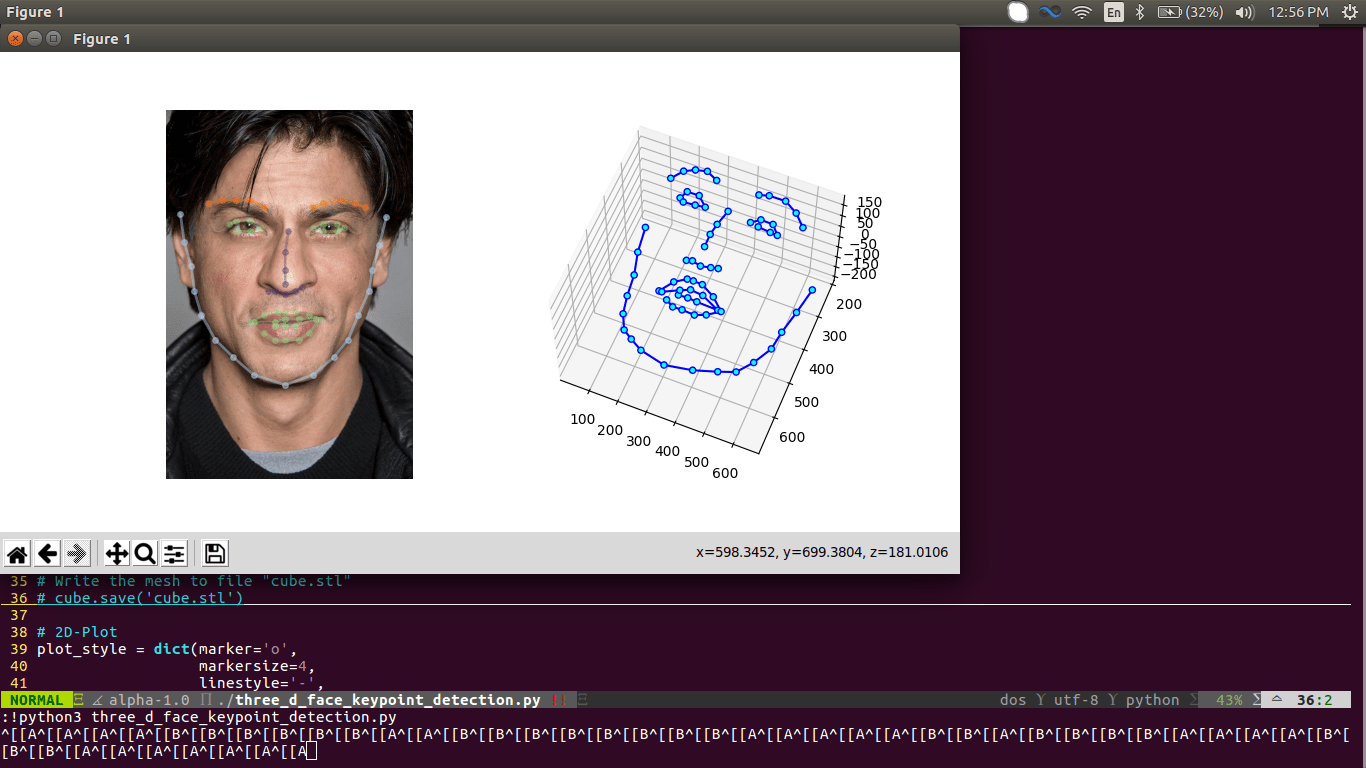
fig = plt.figure(figsize=plt.figaspect(.5))
ax = fig.add_subplot(1, 2, 1)
ax.imshow(input_img)
for pred_type in pred_types.values():
ax.plot(preds[pred_type.slice, 0],
preds[pred_type.slice, 1],
color=pred_type.color, **plot_style)
ax.axis('off')
# 3D-Plot
ax = fig.add_subplot(1, 2, 2, projection='3d')
surf = ax.scatter(preds[:, 0] * 1.2,
preds[:, 1],
preds[:, 2],
c='cyan',
alpha=1.0,
edgecolor='b')
for pred_type in pred_types.values():
ax.plot3D(preds[pred_type.slice, 0] * 1.2,
preds[pred_type.slice, 1],
preds[pred_type.slice, 2], color='blue')
ax.view_init(elev=90., azim=90.)
ax.set_xlim(ax.get_xlim()[::-1])
plt.show()
cube.vectors[i][j] = vertices[f[j],:]
Ошибка:
IndexError: only integers, slices (`:`), ellipsis (`...`), numpy.newaxis (`None`) and integer or boolean arrays are valid indices
Если я удалю
numpy-stl codeblock это вывод в matplotlib.