Как объединить диаграммы рассеяния, чтобы сформировать матрицу диаграммы рассеяния с общей осью X для различной оси Y?
Мой сюжет выглядит так
Это то, что я пробовал. Я делаю отдельные диаграммы рассеяния и объединяю их вместе с
grid.arrange.
data(methylmercurydata)
p1 <- ggplot(data=methylmercurydata,aes(x=MeHg, y=logTHg)) + geom_point()
p2 <- ggplot(data=methylmercurydata,aes(x=MeHg, y=OM)) + geom_point()
p3 <- ggplot(data=methylmercurydata,aes(x=MeHg, y=FeRB)) + geom_point()
grid.arrange(p1,p2,p3)
2 ответа
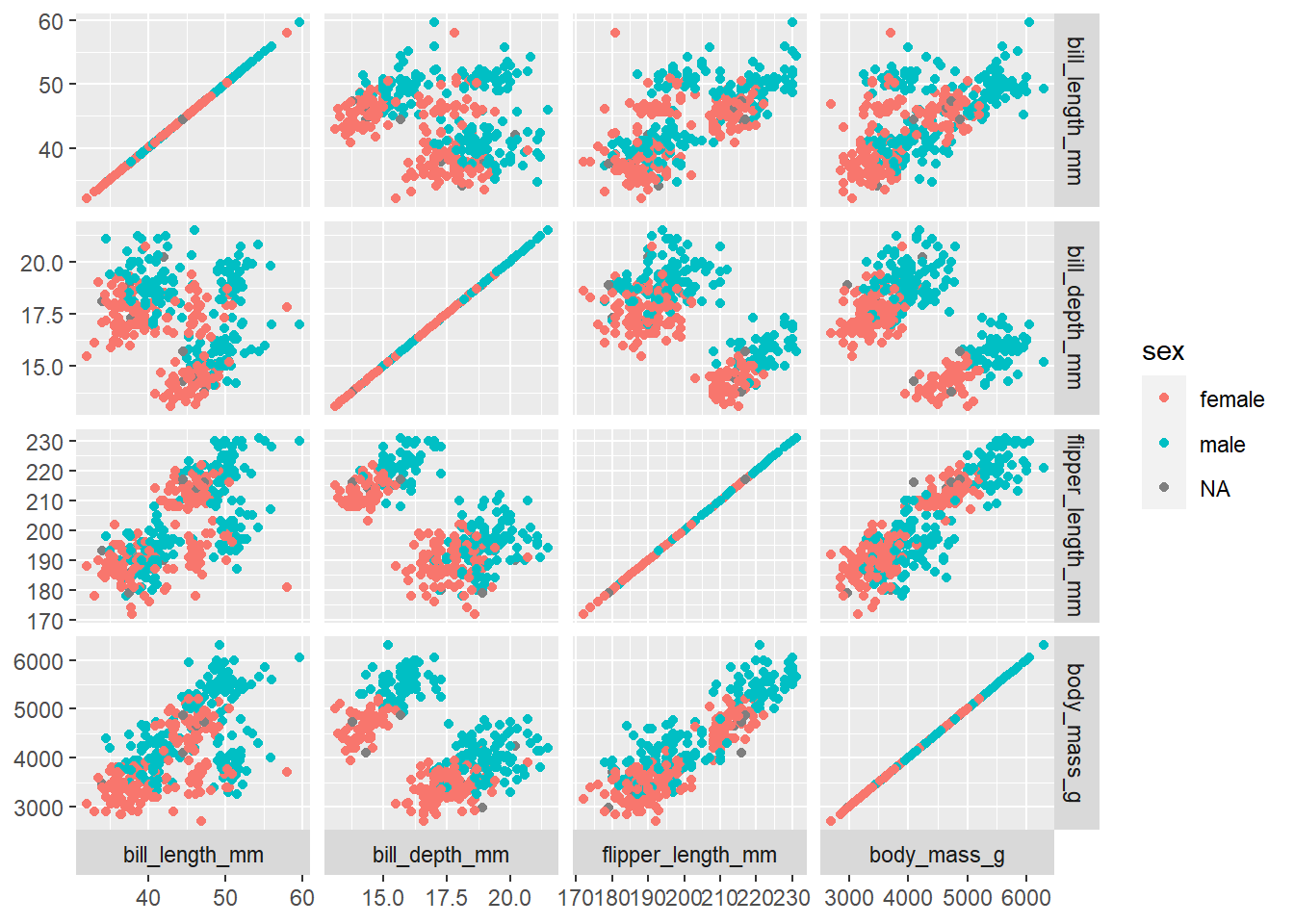
Мне не ясно, что вы имеете в виду под "матрицей диаграммы рассеяния", но если вы хотите создать матрицу корреляции, вы можете использовать ggforce (например, на https://ihaddadenfodil.com/post/it-s-a-bird-it-s-a-plane-it-s-a-ggforce-function/):
library(tidyverse)
library(ggforce)
library(palmerpenguins)
ggplot(penguins, aes(col = sex)) +
geom_autopoint(na.rm = TRUE) +
facet_matrix(rows = vars(bill_length_mm:body_mass_g),
switch = "x")
Самый простой способ сделать это, вероятно, будет использовать
dplyr::facet_wrap() после создания более длинной таблицы.
Что-то вроде:
library(tidyverse)
methylmercurydata %>%
pivot_longer(cols = c(logTHg, OM, FeRB), names_to = 'metric') %>%
ggplot() +
geom_point(aes(MeHG, value)) +
facet_wrap(~metric)
Изменить: r2evans делает хорошее замечание; если вам нужны отдельные шкалы по y, вы можете использовать
scales = 'free_y' в пределах
facet_wrapвызов. Точно так же
scales = 'free_x' предоставит разные оси x, и
scales = 'free'предоставит разные масштабы для каждой оси. Еще одна вещь, которую следует учитывать при воссоздании вышеуказанного графика, - это указать
ncol аргумент также, в этом случае
ncol = 1.