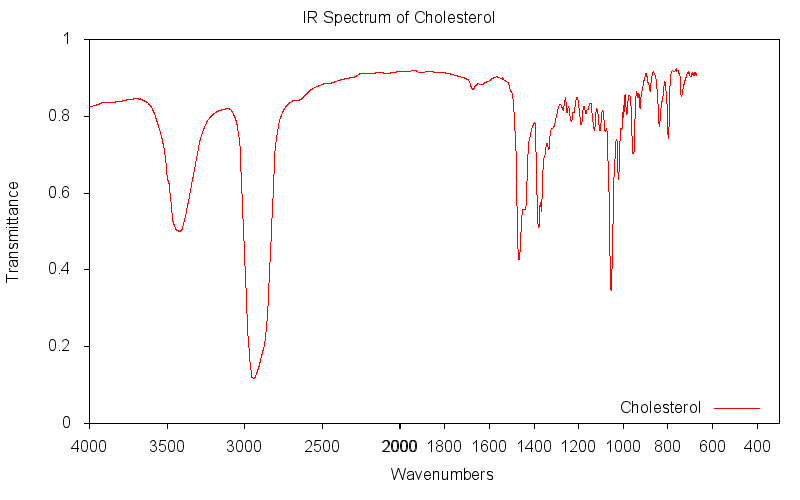
Построение ИК-спектра с помощью Gnuplot
У меня есть инфракрасный спектр для интересующего соединения, который я хотел бы построить, и у меня есть файл spectrum.dat со всеми точками данных. Это имеет форму:
# X Y
300 100
301 100
302 99
303 70
...
3999 98
4000 100
Я хотел бы построить это с использованием оси X, типичной для ИК-спектров, но у меня возникли проблемы с этим. Если вы незнакомы, вот как может выглядеть типичный ИК-спектр (кроме меток на самом графике). Обратите внимание, что ось X перевернута и что она резко удваивает масштабирование выше 2000 единиц (обратных сантиметров). Есть ли способ заставить Gnuplot составить таким образом мои данные? Мне до сих пор удалось придумать следующий скрипт:
# Make an SVG of size 800x500
set terminal svg size 800,500 fname 'CMU Sans Serif' fsize '10'
set output 'ir.svg'
# Color definitions
set border linewidth 1.5
set style line 1 lc rgb '#a0a0a0' lt 1 lw 2 pt 7 # gray
# Format graph
unset key
set xlabel 'Wavenumbers'
set ylabel 'Transmittance'
set xrange [4000:300]
# Plot data
plot 'spectrum.dat' with lines ls 1
Это прекрасно переворачивает ось X, но я не могу понять, как изменить масштабирование таким необычным способом.
2 ответа
Ответ Andyras хороший, это, возможно, более простое (более элегантное:-P) решение с точки зрения параметров макета. Это также должно быть более универсальным решением. Если тиков не так много (см. Рисунок ниже, если их слишком много), то это можно сделать, масштабируя саму кривую за пределами 2000, а затем добавляя все тики вручную. Поскольку у меня нет доступных данных ИК-спектра, я буду использовать фиктивный файл "+" и график log(x) от 4000 до 500:
xmax=4000 ; xmin = 500
pivot = 2000 ; rescfactor = 2.
rescale(x) = (x >= pivot ? x : pivot + rescfactor*(x-pivot))
set xrange [rescale(xmax):rescale(xmin)]
set xtics ("4000" 4000, "3000" 3000, "2000" 2000, \
"1500" rescale(1500), "1000" rescale(1000), "500" rescale(500))
plot "+" u (rescale($1)):(log($1)) w l
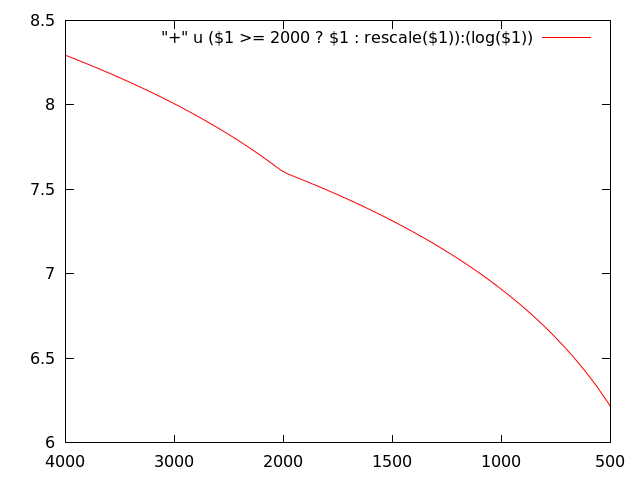
В вашем случае вы просто заменяете log($1) от 2 или что вы замышляете.
В более новых версиях gnuplot (начиная с 4.4) добавление тиков может выполняться автоматически с помощью цикла:
xmax = 4000 ; xmin = 500 ; step = 500
set xtics (sprintf("%i",xmax) rescale(xmax)) # Add the first tic by hand
set for [i=xmin:xmax-step:step] xtics add (sprintf("%i",i) rescale(i))
Начиная с gnuplot 4.6 также другой for строительство может быть сделано с использованием do for:
do for [i=xmin:xmax-step:step] {set xtics add (sprintf("%i",i) rescale(i))}
Как химик, я мотивирован, чтобы ответить...
Насколько я знаю, gnuplot не может легко разрешить произвольное масштабирование оси (если у кого-то нет ярких идей о том, как использовать set link). Моя стратегия в такой ситуации состоит в том, чтобы построить две половины по отдельности и объединить их без проблем:
#!/usr/bin/env gnuplot
set terminal png size 800,500
set output 'ir.png'
set xlabel 'Wavenumbers' offset 20
set ylabel 'Transmittance'
set tics out nomirror
set key bottom right
set bmargin 4
set yrange [0:1]
set multiplot layout 1,2 title 'IR Spectrum of Cholesterol'
# left half of plot
set xrange [4000:2000]
set rmargin 0
set border 7
plot 'cholesterol.txt' notitle
# right half of plot
set xrange [1999:300]
set lmargin 0
set rmargin 2
set border 13
unset xlabel
unset ylabel
unset ytics
plot 'cholesterol.txt' title 'Cholesterol'
unset multiplot
Мое единственное утешение в том, что 2000 написано дважды и выглядит смелее на моем экране, но я оставлю волнения с тиками для вас.