извлекать данные из файла netcdf, содержащегося в границах шейп-файла
У меня есть следующий шейп- файл и файл netcdf.
Я хотел бы извлечь данные из файла netcdf, которые содержатся в границах шейп-файла.
Есть ли у вас какие-нибудь предложения, как я могу этого добиться?
Шейп-файл соответствует региону SREX 11 Северная Европа (NEU), а файл netcdf является примером выходных данных климатической модели CMIP6 (переменная ua). Мой желаемый результат должен быть в формате netcdf.
Обновить
До сих пор я пытался создать маску netcdf, используя NCL и CDO, и применить эту маску к исходному набору данных netcdf. Вот шаги (и сценарии NCL):
#################
## remove plev dimension from netcdf file
cdo --reduce_dim -copy nc_file.nc nc_file2.nc
## convert longitude to -180, 180
cdo sellonlatbox,-180,180,-90,90 nc_file2.nc nc_file3.nc
## create mask
ncl create_nc_mask.ncl
## apply mask
cdo div nc_file3.nc shape1_mask.nc nc_file4.nc
#################
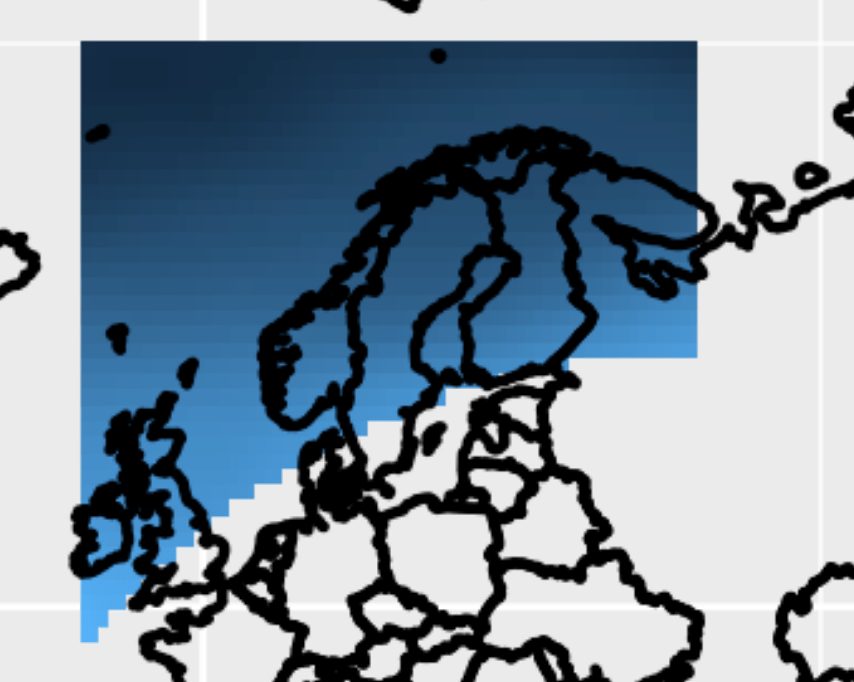
Вывод почти правильный. См. Картинку ниже. Но южные границы шейп-файла (SREX 11, NEU) не захватываются правильно. Итак, я полагаю, что что-то не так в сценарии NCL, который генерирует маску netcdf.
2 ответа
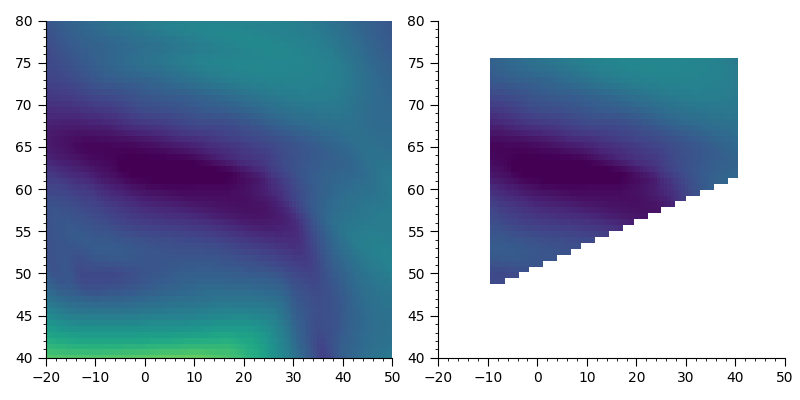
Повторно используя некоторые старые скрипты / код, я быстро придумал это для решения Python. По сути, он просто перебирает все точки сетки и проверяет, находится ли каждая точка сетки внутри или за пределами многоугольника из файла формы. Результатом является переменнаяmask (массив с True/False), который можно использовать для маскировки ваших переменных NetCDF.
Примечание: здесь используется Numba (все@jitlines) для ускорения кода, хотя в данном случае в этом нет необходимости. Вы можете просто прокомментировать их, если у вас нет Numba.
import matplotlib.pyplot as pl
import netCDF4 as nc4
import numpy as np
import fiona
from numba import jit
@jit(nopython=True, nogil=True)
def distance(x1, y1, x2, y2):
"""
Calculate distance from (x1,y1) to (x2,y2)
"""
return ((x1-x2)**2 + (y1-y2)**2)**0.5
@jit(nopython=True, nogil=True)
def point_is_on_line(x, y, x1, y1, x2, y2):
"""
Check whether point (x,y) is on line (x1,y1) to (x2,y2)
"""
d1 = distance(x, y, x1, y1)
d2 = distance(x, y, x2, y2)
d3 = distance(x1, y1, x2, y2)
eps = 1e-12
return np.abs((d1+d2)-d3) < eps
@jit(nopython=True, nogil=True)
def is_left(xp, yp, x0, y0, x1, y1):
"""
Check whether point (xp,yp) is left of line segment ((x0,y0) to (x1,y1))
returns: >0 if left of line, 0 if on line, <0 if right of line
"""
return (x1-x0) * (yp-y0) - (xp-x0) * (y1-y0)
@jit(nopython=True, nogil=True)
def is_inside(xp, yp, x_set, y_set, size):
"""
Given location (xp,yp) and set of line segments (x_set, y_set), determine
whether (xp,yp) is inside polygon.
"""
# First simple check on bounds
if (xp < x_set.min() or xp > x_set.max() or yp < y_set.min() or yp > y_set.max()):
return False
wn = 0
for i in range(size-1):
# Second check: see if point exactly on line segment:
if point_is_on_line(xp, yp, x_set[i], y_set[i], x_set[i+1], y_set[i+1]):
return False
# Calculate winding number
if (y_set[i] <= yp):
if (y_set[i+1] > yp):
if (is_left(xp, yp, x_set[i], y_set[i], x_set[i+1], y_set[i+1]) > 0):
wn += 1
else:
if (y_set[i+1] <= yp):
if (is_left(xp, yp, x_set[i], y_set[i], x_set[i+1], y_set[i+1]) < 0):
wn -= 1
if wn == 0:
return False
else:
return True
@jit(nopython=True, nogil=True)
def calc_mask(mask, lon, lat, shp_lon, shp_lat):
"""
Calculate mask of grid points which are inside `shp_lon, shp_lat`
"""
for j in range(lat.size):
for i in range(lon.size):
if is_inside(lon[i], lat[j], shp_lon, shp_lat, shp_lon.size):
mask[j,i] = True
if __name__ == '__main__':
# Selection of time and level:
time = 0
plev = 0
# Read NetCDF variables, shifting the longitudes
# from 0-360 to -180,180, like the shape file:
nc = nc4.Dataset('nc_file.nc')
nc_lon = nc.variables['lon'][:]-180.
nc_lat = nc.variables['lat'][:]
nc_ua = nc.variables['ua'][time,plev,:,:]
# Read shapefile and first feature
fc = fiona.open("shape1.shp")
feature = next(iter(fc))
# Extract array of lat/lon coordinates:
coords = feature['geometry']['coordinates'][0]
shp_lon = np.array(coords)[:,0]
shp_lat = np.array(coords)[:,1]
# Calculate mask
mask = np.zeros_like(nc_ua, dtype=bool)
calc_mask(mask, nc_lon, nc_lat, shp_lon, shp_lat)
# Mask the data array
nc_ua_masked = np.ma.masked_where(~mask, nc_ua)
# Plot!
pl.figure(figsize=(8,4))
pl.subplot(121)
pl.pcolormesh(nc_lon, nc_lat, nc_ua, vmin=-40, vmax=105)
pl.xlim(-20, 50)
pl.ylim(40, 80)
pl.subplot(122)
pl.pcolormesh(nc_lon, nc_lat, nc_ua_masked, vmin=-40, vmax=105)
pl.xlim(-20, 50)
pl.ylim(40, 80)
pl.tight_layout()
РЕДАКТИРОВАТЬ
Чтобы записать маску в NetCDF, можно использовать что-то вроде этого:
nc_out = nc4.Dataset('mask.nc', 'w')
nc_out.createDimension('lon', nc_lon.size)
nc_out.createDimension('lat', nc_lat.size)
nc_mask_out = nc_out.createVariable('mask', 'i2', ('lat','lon'))
nc_lon_out = nc_out.createVariable('lon', 'f8', ('lon'))
nc_lat_out = nc_out.createVariable('lat', 'f8', ('lat'))
nc_mask_out[:,:] = mask[:,:] # Or ~mask to reverse it
nc_lon_out[:] = nc_lon[:] # With +180 if needed
nc_lat_out[:] = nc_lat[:]
nc_out.close()
Пока что я придумал это (я знаю, что это не полное решение):
1) Чтобы открыть шейп-файл и файл NC, вам потребуется установить два пакета:
pip3 install pyshp
pip3 install netCDF4
2) Тогда вот как вы импортируете их в python:
import shapefile
from netCDF4 import Dataset
3) Прочитать данные из шейп-файла:
with shapefile.Reader("shape1.dbf") as dbf:
print(f'{dbf}\n')
print(f'bounding box: {dbf.bbox}')
shapes = dbf.shapes()
print(f'points: {shapes[0].points}')
print(f'parts: {shapes[0].parts}')
print(f'fields: {dbf.fields}')
records = dbf.records()
dct = records[0].as_dict()
print(f'record: {dct}')
Это дает вам результат:
shapefile Reader
1 shapes (type 'POLYGON')
1 records (4 fields)
bounding box: [-10.0, 48.0, 40.0, 75.0]
points: [(-10.0, 48.0), (-10.0, 75.0), (40.0, 75.0), (40.0, 61.3), (-10.0, 48.0)]
parts: [0]
fields: [('DeletionFlag', 'C', 1, 0), ['NAME', 'C', 40, 0], ['LAB', 'C', 40, 0], ['USAGE', 'C', 40, 0]]
record: {'NAME': 'North Europe [NEU:11]', 'LAB': 'NEU', 'USAGE': 'land'}
4) Прочтите файл NC:
nc_fdata = Dataset('nc_file.nc', 'r')
5) Используйте эту вспомогательную функцию, чтобы увидеть, что внутри:
def ncdump(nc_fid, verb=True):
def print_ncattr(key):
try:
print("\t\ttype:", repr(nc_fid.variables[key].dtype))
for ncattr in nc_fid.variables[key].ncattrs():
print('\t\t%s:' % ncattr, repr(nc_fid.variables[key].getncattr(ncattr)))
except KeyError:
print("\t\tWARNING: %s does not contain variable attributes" % key)
# NetCDF global attributes
nc_attrs = nc_fid.ncattrs()
if verb:
print("NetCDF Global Attributes:")
for nc_attr in nc_attrs:
print('\t%s:' % nc_attr, repr(nc_fid.getncattr(nc_attr)))
nc_dims = [dim for dim in nc_fid.dimensions] # list of nc dimensions
# Dimension shape information.
if verb:
print("NetCDF dimension information:")
for dim in nc_dims:
print("\tName:", dim)
print("\t\tsize:", len(nc_fid.dimensions[dim]))
print_ncattr(dim)
# Variable information.
nc_vars = [var for var in nc_fid.variables] # list of nc variables
if verb:
print("NetCDF variable information:")
for var in nc_vars:
if var not in nc_dims:
print('\tName:', var)
print("\t\tdimensions:", nc_fid.variables[var].dimensions)
print("\t\tsize:", nc_fid.variables[var].size)
print_ncattr(var)
return nc_attrs, nc_dims, nc_vars
nc_attrs, nc_dims, nc_vars = ncdump(nc_fdata)
Я предполагаю, что вам понадобится переменная с именем 'ua', потому что у нее, среди прочего, есть адреса долготы и широты.
Итак, чтобы построить маску, вам нужно будет извлечь все из ua, где долгота и широта находятся между значениями ограничивающего прямоугольника шейп-файла.