Обработка данных в Shiny: построение недавно обработанных данных после анализа кластеризации k-средних
Я пытаюсь создать панель инструментов для анализа данных, и я использую Shiny, в котором я относительно новичок. Одна из функций моей панели инструментов использует кластеризацию k-средних для пользовательских данных. Я могу заставить кластерный анализ работать нормально, но я хочу иметь возможность проводить исследовательский анализ данных по отдельным кластерам после завершения первоначального кластерного анализа. Кроме того, я хотел бы сделать это с реактивными фреймами данных в Shiny, чтобы, если пользователь изменяет значение на приборной панели, анализ обновляется, включая исследовательский материал после кластеризации.
Прежде всего, вот некоторые функции, которые я использую в коде сервера приборной панели и соответствующих библиотеках, поэтому сначала запустите их:
#libraries===================================================================
library(ids)
library(tidyverse)
library(dplyr)
library(shiny)
library(ggplot2)
library(shinydashboard)
library(shinyWidgets)
library(factoextra)
#functions required==========================================================
#scale https://stackru.com/questions/35775696/trying-to-use-dplyr-to-group-by-and-apply-scale
scale_this <- function(x){
(x - mean(x, na.rm=TRUE)) / sd(x, na.rm=TRUE)
}
#wss plot
wssplot <- function(data, nc = 15, seed = 1234) {
wss <- (nrow(data) - 1) * sum(apply(data, 2, var))
for (i in 2:nc) {
set.seed(seed)
wss[i] <- sum(kmeans(data, centers = i)$withinss)
}
plot(1:nc,
wss,
type = "b",
xlab = "Number of Clusters",
ylab = "Within groups sum of squares")
}
Вот код фиктивного фрейма данных для этого примера:-
#Create my mock data frame============================================
set.seed(123)
randomid<-random_id(333)#from 'ids' library
Duration<-c(floor(runif(10000, min=1, max=1000)))
mockdf<-cbind(randomid, Duration)
mockdf<-as.data.frame(mockdf)
mockdf$Duration<-as.numeric(mockdf$Duration)
Мой код пользовательского интерфейса:-
#UI============================================================================
ui<-fluidPage(
titlePanel('Minimal example'),
tabsetPanel(
#=============================================kmeans clustering==================================================
tabPanel("User Type Discovery",
sidebarLayout(
sidebarPanel(width = 4,numericInput('ksolution', 'Select k solution', 5),
pickerInput('userselect', 'Which users do you want to include:',
choices = unique(mockdf$randomid), options = list('actions-box'=TRUE),multiple = T)),
mainPanel(fluidRow(
column(12, plotOutput("elbowplot")),
column(12, plotOutput("clustplot")),
column(12, plotOutput("clust_dens")),
column(12, DT::dataTableOutput('Clusterdf'))))
)
)
)
)
И мой серверный код:-
#SERVER===========================================================
server<-function(input,output,session){
#create reactive dataframe
rval_df <-reactive({
mockdf
})
#=============================================kmeans clustering==================================================
rval_UserData<-reactive({
rval_df()%>%
filter(randomid %in% input$userselect)%>%
group_by(randomid)%>%
summarise(Count=n(),MeanDuration=mean(Duration),SDDuration=sd(Duration))%>%
mutate(SDDuration=if_else(is.na(SDDuration),0,SDDuration),
Cluster=as.factor(rval_kclust()$cluster))
})
#create a scaled dataset for the clustering
rval_cluster_df<-reactive({
rval_df()%>%
filter(randomid %in% input$userselect)%>%
group_by(randomid)%>%
summarise(Count=n(),MeanDuration=mean(Duration),SDDuration=sd(Duration))%>%
mutate(SDDuration=if_else(is.na(SDDuration),0,SDDuration),
Count=scale_this(Count),
MeanDuration=scale_this(MeanDuration),
SDDuration=scale_this(SDDuration))%>%
select(Count,MeanDuration,SDDuration)
})
#cluster algorithm
rval_kclust<-reactive({
kmeans(rval_cluster_df(), centers = input$ksolution)
})
output$clustplot<-renderPlot({
factoextra::fviz_cluster(rval_kclust(), data = rval_cluster_df())
})
output$elbowplot<-renderPlot({
wssplot(rval_cluster_df())
})
output$Clusterdf<- DT::renderDataTable({
rval_UserData()
})
}
shinyApp(ui, server)
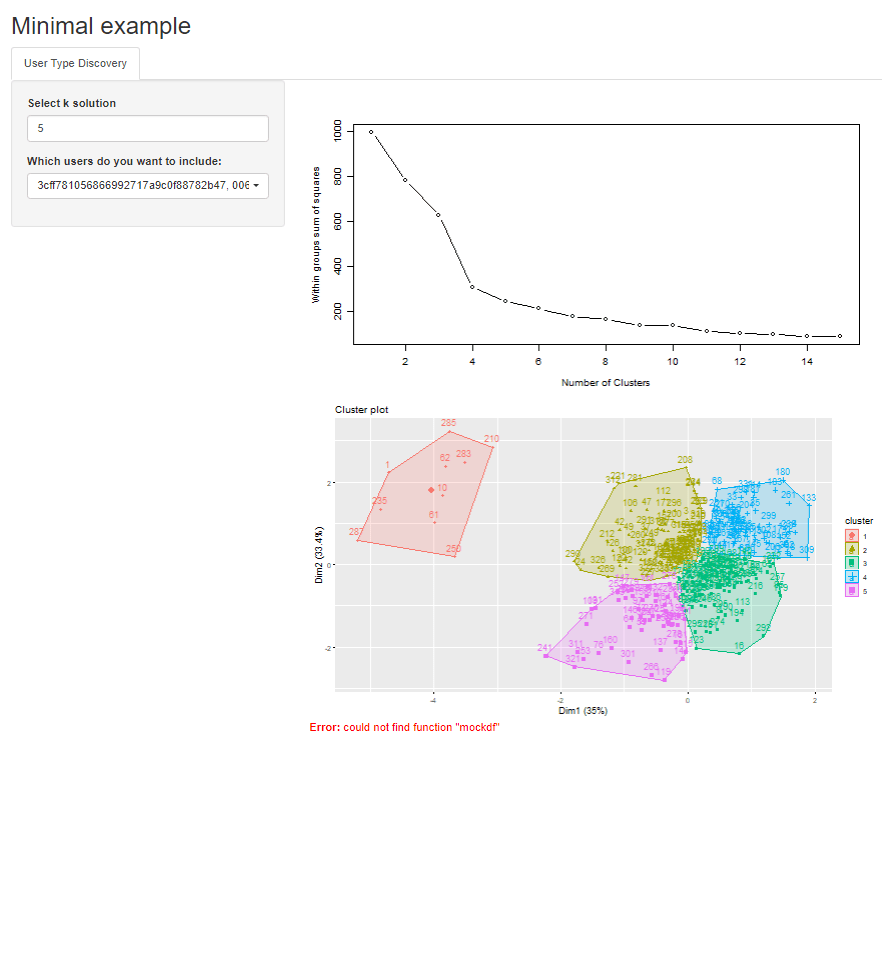
Когда ты бежишь shinyApp(ui,server)нажмите кнопку "Выбрать все" в раскрывающемся списке приложения, чтобы запустить кластеризацию.
Вот что я хочу сделать. Поскольку я снова присвоил номер кластераrval_UserData(), Я хочу иметь возможность объединить это присвоить номер кластера mockdf, поэтому я могу создавать графики, используя ggplot2 на Durationпеременная, а также создавать сводные таблицы на уровне кластера. Я предпочитаю иметь возможность делать это с помощью реактивных фреймов данных, поэтому графики будут обновляться в зависимости отksolution ввод в пользовательский интерфейс.
Вот некоторые из моих попыток объединить номер кластера обратно в mockdf, с последующей попыткой построить график плотности:-
rval_cluster_merged_df<-reactive({
merge(mockdf(), rval_UserData(), by="randomid")
#outside of shiny, this would be a quick way to paste the cluster number back onto the mock dataframe
})
output$clust_dens<-renderPlot({
dd<-rval_cluster_merged_df()
ggplot(dd,aes(x=Duration, colour=Cluster, group=Cluster))+
geom_density()+ggtitle("Cluster density plot")+scale_x_log10()
})
И вот что я получаю, см. Сообщение об ошибке:-
Вероятно, что-то очевидно, что я делаю неправильно, но любые указатели в правильном направлении будут оценены! Заранее спасибо:)
1 ответ
Вам нужно использовать req() для всех input$abc переменные и eval_tidyпоскольку они не являются стандартными переменными. Незначительное обновление функции вашего сервера, как показано ниже, решит вашу проблему.
server<-function(input,output,session){
#create reactive dataframe
rval_df <-reactive({
mockdf
})
#=============================================kmeans clustering==================================================
rval_UserData<-reactive({
req(input$userselect)
userselect <- eval_tidy(input$userselect)
rval_df()%>%
filter(randomid %in% userselect)%>%
group_by(randomid)%>%
summarise(Count=n(),MeanDuration=mean(Duration),SDDuration=sd(Duration))%>%
mutate(SDDuration=if_else(is.na(SDDuration),0,SDDuration),
Cluster=as.factor(rval_kclust()$cluster))
})
#create a scaled dataset for the clustering
rval_cluster_df<-reactive({
req(input$userselect)
userselect <- eval_tidy(input$userselect)
rval_df()%>%
filter(randomid %in% userselect)%>%
group_by(randomid)%>%
summarise(Count=n(),MeanDuration=mean(Duration),SDDuration=sd(Duration))%>%
mutate(SDDuration=if_else(is.na(SDDuration),0,SDDuration),
Count=scale_this(Count),
MeanDuration=scale_this(MeanDuration),
SDDuration=scale_this(SDDuration))%>%
select(Count,MeanDuration,SDDuration)
})
#cluster algorithm
rval_kclust<-reactive({
req(input$ksolution)
centers <- as.numeric(eval_tidy(input$ksolution))
kmeans(rval_cluster_df(), centers = centers)
})
output$clustplot<-renderPlot({
factoextra::fviz_cluster(rval_kclust(), data = rval_cluster_df())
})
output$elbowplot<-renderPlot({
wssplot(rval_cluster_df())
})
output$Clusterdf<- DT::renderDataTable({
rval_UserData()
})
rval_cluster_merged_df<-reactive({
merge(rval_df(), rval_UserData(), by="randomid")
})
output$clust_dens<-renderPlot({
dd<-rval_cluster_merged_df()
ggplot(dd,aes(x=Duration, colour=Cluster, group=Cluster))+
geom_density()+ggtitle("Cluster density plot")+scale_x_log10()
})
}
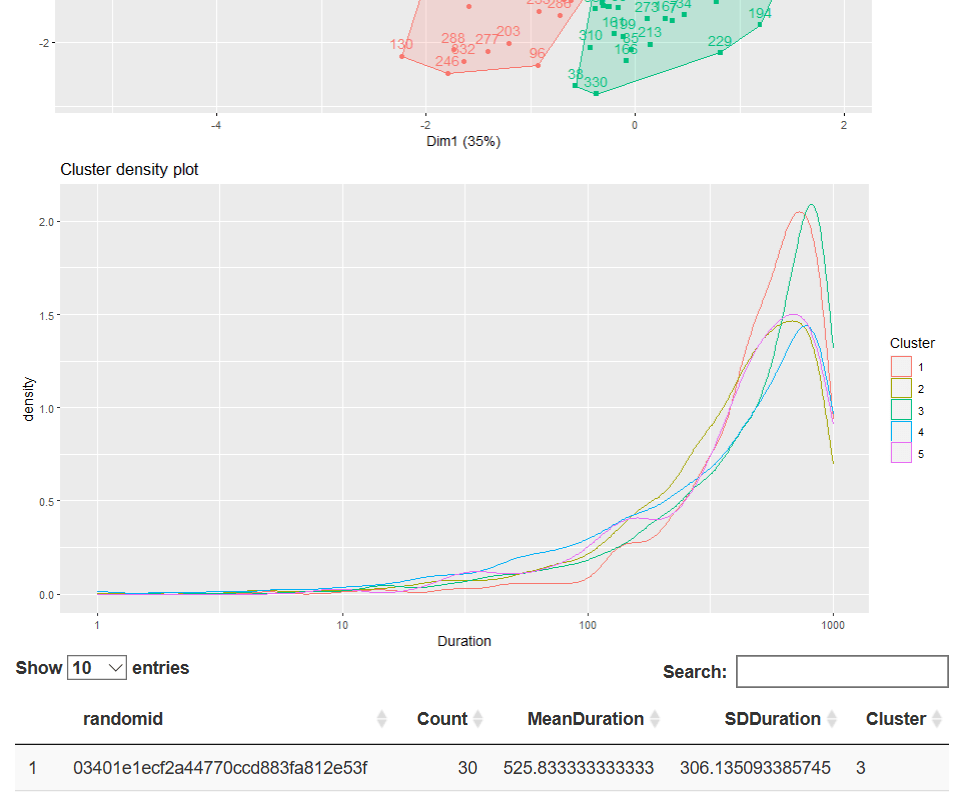
Окончательный результат будет: