Построение R2 каждого / определенного компонента PCA на длину волны с R
У меня есть некоторый опыт использования PCA, но я впервые пытаюсь использовать PCA для спектральных данных...
У меня есть большие данные со спектрами, где я использовал команду prcomp для расчета PCA для всего набора данных. Мои результаты показывают, что 3 компонента объясняют 99% дисперсии.
Я хотел бы представить вклад каждого из трех компонентов PCA на каждой длине волны (с шагом 4, 200-1000 нм), как пример графика 2, который я нашел на этом сайте: https://learnche.org/pid / латентное переменное-моделирование / принципал-компонентный анализ / PCA-пример-анализ-оф-спектрально-данных
У кого-нибудь есть код, как я мог бы сделать это в R?
Спасибо
1 ответ
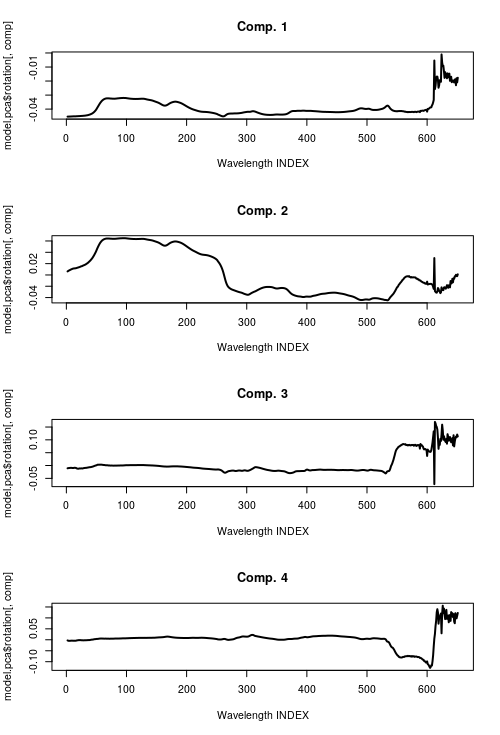
Я считаю, что матрица переменных нагрузок находится в model.pca$rotation см. документацию prcomp. Так что-то вроде этого должно быть сделано (используя пример на вашем сайте):
file <- 'http://openmv.net/file/tablet-spectra.csv'
spectra <- read.csv(file, header = FALSE)
n.comp <- 4
model.pca <- prcomp(spectra[,2:651],
center = TRUE,
scale =TRUE,
rank. = n.comp)
summary(model.pca)
par(mfrow=c(n.comp,1))
sapply(1:n.comp, function(comp){
plot(2:651, model.pca$rotation[,comp], type='l', lwd=2,
main=paste("Comp.", comp), xlab="Wavelength INDEX")
})
У меня нет значений длины волны, поэтому я использовал индексы массива здесь; вывод ниже.