Как сдвинуть ячейки на диаграмме роз, используя пакет "круговой" в R
Я создал диаграмму аспектов розы в градусах для данных о местоположении, используя "круговой" пакет в R и функцию rose.diag с основными аспектами N, NE, E и т. Д., В общей сложности 8 ячеек. Однако мусорные ведра не колеблются между аспектами. Другими словами, первая ячейка идет от 0-45, вторая - от 45 до 90 и т. Д., Что странным образом объединяет данные аспекта. Есть ли способ сместить ячейки так, чтобы 0, 45, 90 и т. Д. Были центрами корзин, а не краями?
rose.diag(Degrees$Degrees, bins=8,zero=pi/2, units = 'degrees', rotation='clock')
3 ответа
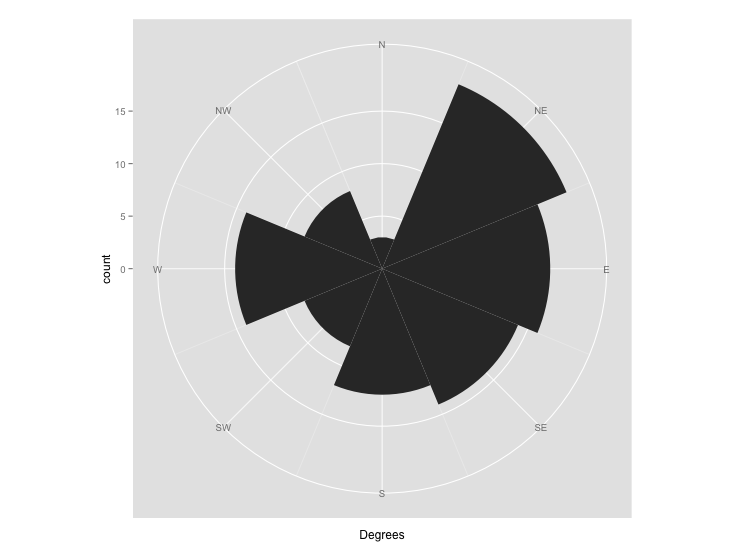
Я думаю, что Бен прав, что это не может быть легко сделано с rose.diagтак вот решение с использованием ggplot2:
library(ggplot2)
Degrees <- runif(100, 0, 360)
rose <- ggplot(mapping = aes(x = Degrees)) +
stat_bin(breaks = (0:8 - 0.5)/8 * 360) +
scale_x_continuous(
breaks = 0:7/8*360,
labels = c("N", "NE", "E", "SE", "S", "SW", "W", "NW")
) +
coord_polar(start=-pi/8)
rose
 Это не может быть идеальным, потому что не все функции в
Это не может быть идеальным, потому что не все функции в rose.diag иметь простые эквиваленты в ggplot2.
Вы можете получить что-то подобное, используя gridBase пакет. Мы продолжаем использовать rose.diag и мы hack сюжет, как только мы окажемся в пространстве хорошего смотрового окна.
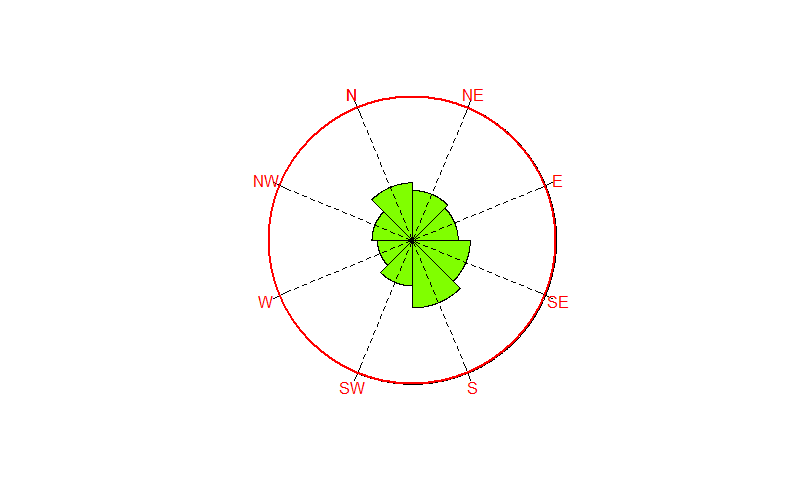
require(grid)
#grid.newpage()
##generate some data
x <- circular(runif(50, 0, 2*pi))
bins <- 8
rotation <- 'clock'
##tcl =0(no ticks), tcl.text=-2 to write away the ticks marks
rose.diag(x, bins=bins,zero=0, rotation='clock',
tcl=0,tcl.text=-2,col='#80FF00FF')
library(gridBase)
## I use the plot viewport
vp <- baseViewports()$plot
pushViewport(vp) ## here we go!
## radial transformation
at <- (0:bins - 0.5)/bins * 2 * pi
## ticks
grid.segments( x0 = .95*sin(at), y0 = 0.95*cos(at),
x1 = 1.05*sin(at), y1 = 1.05*cos(at),
default.units = "native")
## ticks labels
grid.text(x = 1.1*sin(at), default.units = "native",
y = 1.1*cos(at), gp=gpar(col='red'),
label = c("N", "NE", "E", "SE", "S", "SW", "W", "NW"))
Для визуального аспекта я добавлю некоторые настройки, но приведенный выше код уже отвечает на этот вопрос.
## dashed lines from the center for visual aspect
grid.segments( x0 = .95*sin(at), y0 = 0.95*cos(at),
x1 = 0, 0,
gp = gpar(lty="dashed"),
default.units = "native")
## circle just to get the same color of text
grid.circle(r=1,x=0,y=0,gp=gpar(col='red',fill=NA,lwd=2), default.units = "native")
## remove the viewport
popViewport(1)
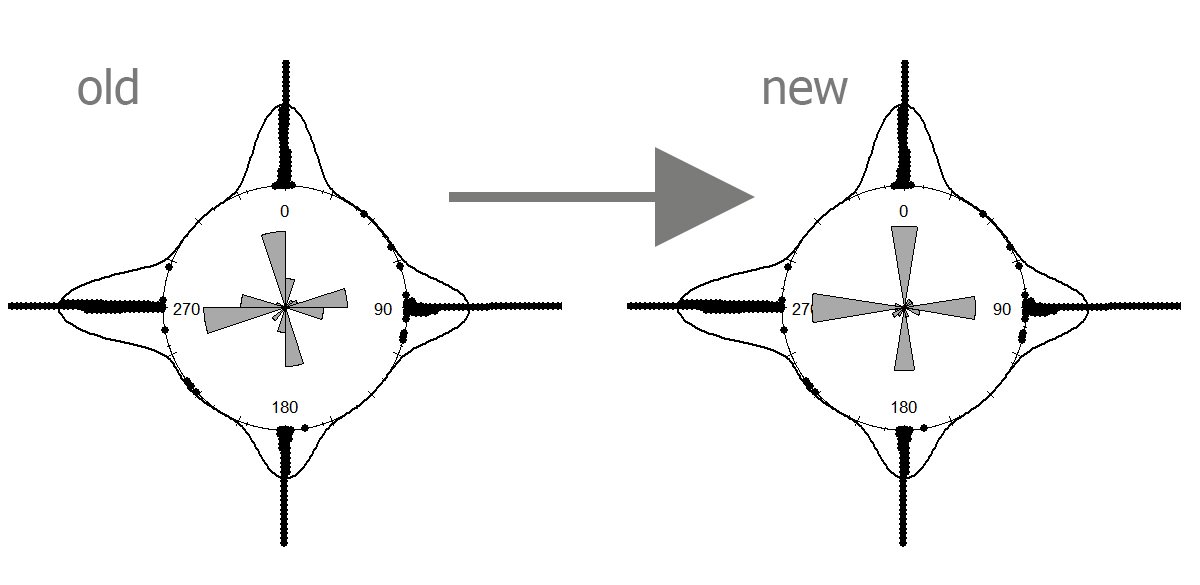
Почему бы не повернуть исходные данные? Nb ниже cdat указывается в градусах (ноль = пи / 2), а ноль в 2* пи
rose.diag(cdat - 10, bins = 20, col="darkgrey", prop=1.3, оси =FALSE, add=TRUE, ноль = pi/2-pi/20)
Скопируйте / вставьте то, над чем я работаю:
library(circular)
raw <-read.csv("C:\\Users\\Andy\\Desktop\\business\\research\\Oxford\\MichelDish\\r.csv", header=T)
raw <-na.omit(raw)
cdat <- circular(raw [, c ("kandUnknown")],type="angles",units="degrees", rotation="clock", zero=pi/2)
plot(cdat, cex=1.1, bin=720, stack=TRUE, sep=0.035, shrink=1.8, tcl.text=.2)
ticks.circular(circular(seq(0,2*pi,pi/8)), zero=pi/2, rotation='clock', tcl=0.075)
rose.diag(cdat - 10, bins = 20, col="darkgrey", prop=1.3, axes=FALSE, add=TRUE, zero = pi/2 - pi/20)
lines(density.circular(cdat, bw=40), lwd=2, lty=1)
Приведенный ниже код дает вам старую цифру (вверху слева):
rose.diag(cdat, bins = 20, col="darkgrey", prop=1.3, axes=FALSE, add=TRUE)
PS для любопытных, мы используем такую статистику, например, http://www.sciencedirect.com/science/article/pii/S0950329315001068