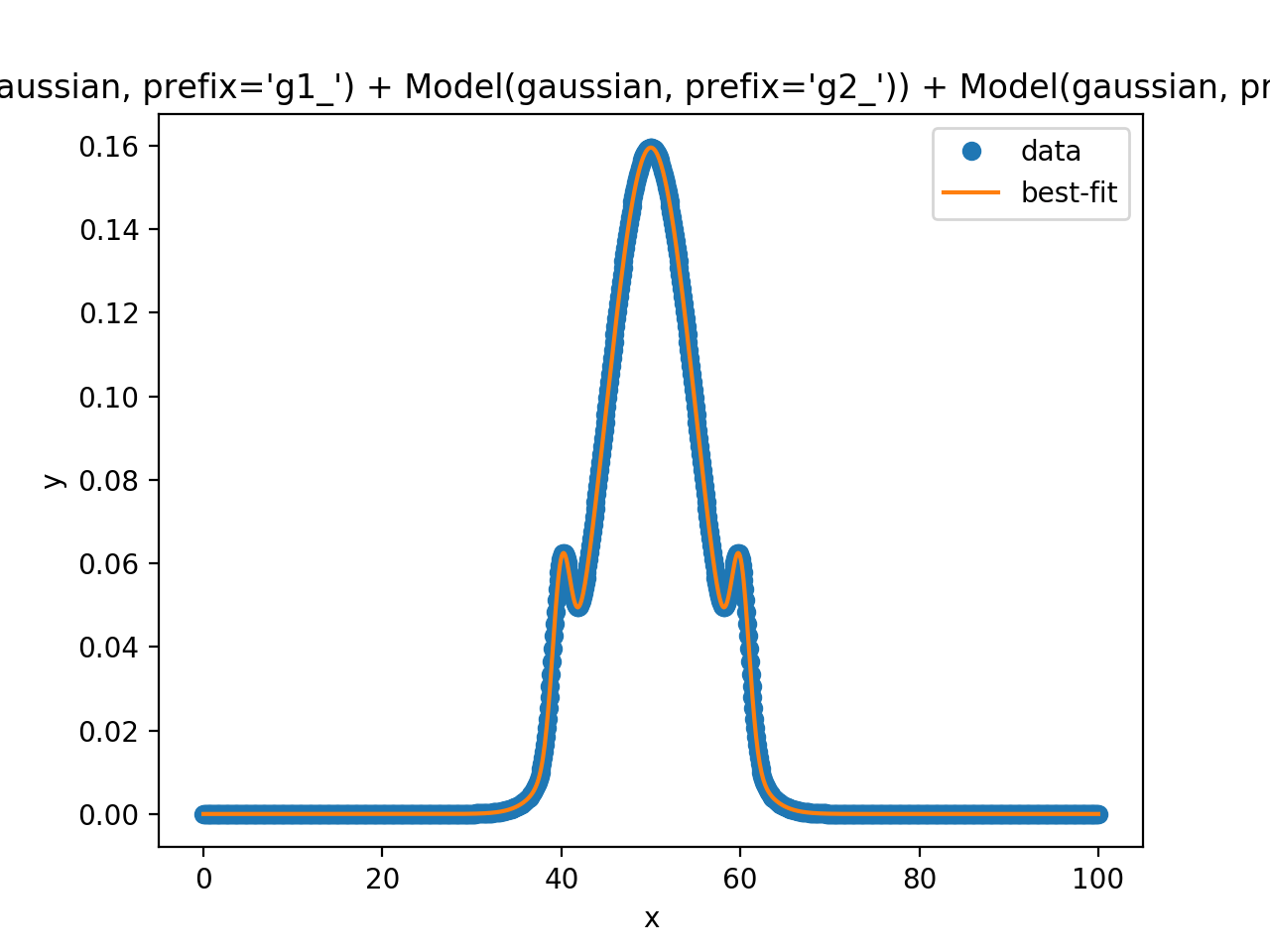
Используйте Python lmfit с переменным количеством параметров в функции
Я пытаюсь преобразовать сложные сигналы газовой хроматограммы в отдельные сигналы Гаусса. Вот пример, где пунктирная линия представляет сигнал, который я пытаюсь деконвольвировать.
 Я смог написать код для этого, используя scipy.optimize.curve_fit; однако после применения к реальным данным результаты оказались ненадежными. Я считаю, что возможность установить границы для моих параметров улучшит мои результаты, поэтому я пытаюсь использовать lmfit, что позволяет это. У меня проблема с тем, чтобы lmfit работал с переменным числом параметров. Сигналы, с которыми я работаю, могут иметь произвольное количество базовых гауссовых компонентов, поэтому количество необходимых мне параметров будет различным. Я нашел некоторые подсказки здесь, но все еще не могу понять это...
Я смог написать код для этого, используя scipy.optimize.curve_fit; однако после применения к реальным данным результаты оказались ненадежными. Я считаю, что возможность установить границы для моих параметров улучшит мои результаты, поэтому я пытаюсь использовать lmfit, что позволяет это. У меня проблема с тем, чтобы lmfit работал с переменным числом параметров. Сигналы, с которыми я работаю, могут иметь произвольное количество базовых гауссовых компонентов, поэтому количество необходимых мне параметров будет различным. Я нашел некоторые подсказки здесь, но все еще не могу понять это...
Создание модели Python lmfit с произвольным количеством параметров
Вот код, с которым я сейчас работаю. Код будет выполняться, но оценки параметров не изменятся, когда модель будет соответствовать. Кто-нибудь знает, как мне заставить мою модель работать?
import numpy as np
from collections import OrderedDict
from scipy.stats import norm
from lmfit import Parameters, Model
def add_peaks(x_range, *pars):
y = np.zeros(len(x_range))
for i in np.arange(0, len(pars), 3):
curve = norm.pdf(x_range, pars[i], pars[i+1]) * pars[i+2]
y = y + curve
return(y)
# generate some fake data
x_range = np.linspace(0, 100, 1000)
peaks = [50., 40., 60.]
a = norm.pdf(x_range, peaks[0], 5) * 2
b = norm.pdf(x_range, peaks[1], 1) * 0.1
c = norm.pdf(x_range, peaks[2], 1) * 0.1
fake = a + b + c
param_dict = OrderedDict()
for i in range(0, len(peaks)):
param_dict['pk' + str(i)] = peaks[i]
param_dict['wid' + str(i)] = 1.
param_dict['mult' + str(i)] = 1.
# In case, you'd like to see the plot of fake data
#y = add_peaks(x_range, *param_dict.values())
#plt.plot(x_range, y)
#plt.show()
# Initialize the model and fit
pmodel = Model(add_peaks)
params = pmodel.make_params()
for i in param_dict.keys():
params.add(i, value=param_dict[i])
result = pmodel.fit(fake, params=params, x_range=x_range)
print(result.fit_report())
2 ответа
Я думаю, что вам лучше использовать lmfitумение строить составную модель.
То есть с одним пиком, определенным с
from scipy.stats import norm
def peak(x, amp, center, sigma):
return amp * norm.pdf(x, center, sigma)
(смотрите также lmfit.models.GaussianModel), вы можете построить модель со многими вершинами:
npeaks = 3
model = Model(peak, prefix='p1_')
for i in range(1, npeaks):
model = model + Model(peak, prefix='p%d_' % (i+1))
params = model.make_params()
Сейчас model будет сумма 3 гауссовских функций, а params созданный для этой модели будет иметь имена, такие как p1_amp, p1_center, p2_amp,..., который вы можете добавить разумные начальные значения и / или границы и / или ограничения.
Учитывая данные вашего примера, вы можете передать начальные значения make_params лайк
params = model.make_params(p1_amp=2.0, p1_center=50., p1_sigma=2,
p2_amp=0.2, p2_center=40., p2_sigma=2,
p3_amp=0.2, p3_center=60., p3_sigma=2)
result = model.fit(fake, params, x=x_range)
Я смог найти решение здесь:
https://lmfit.github.io/lmfit-py/builtin_models.html
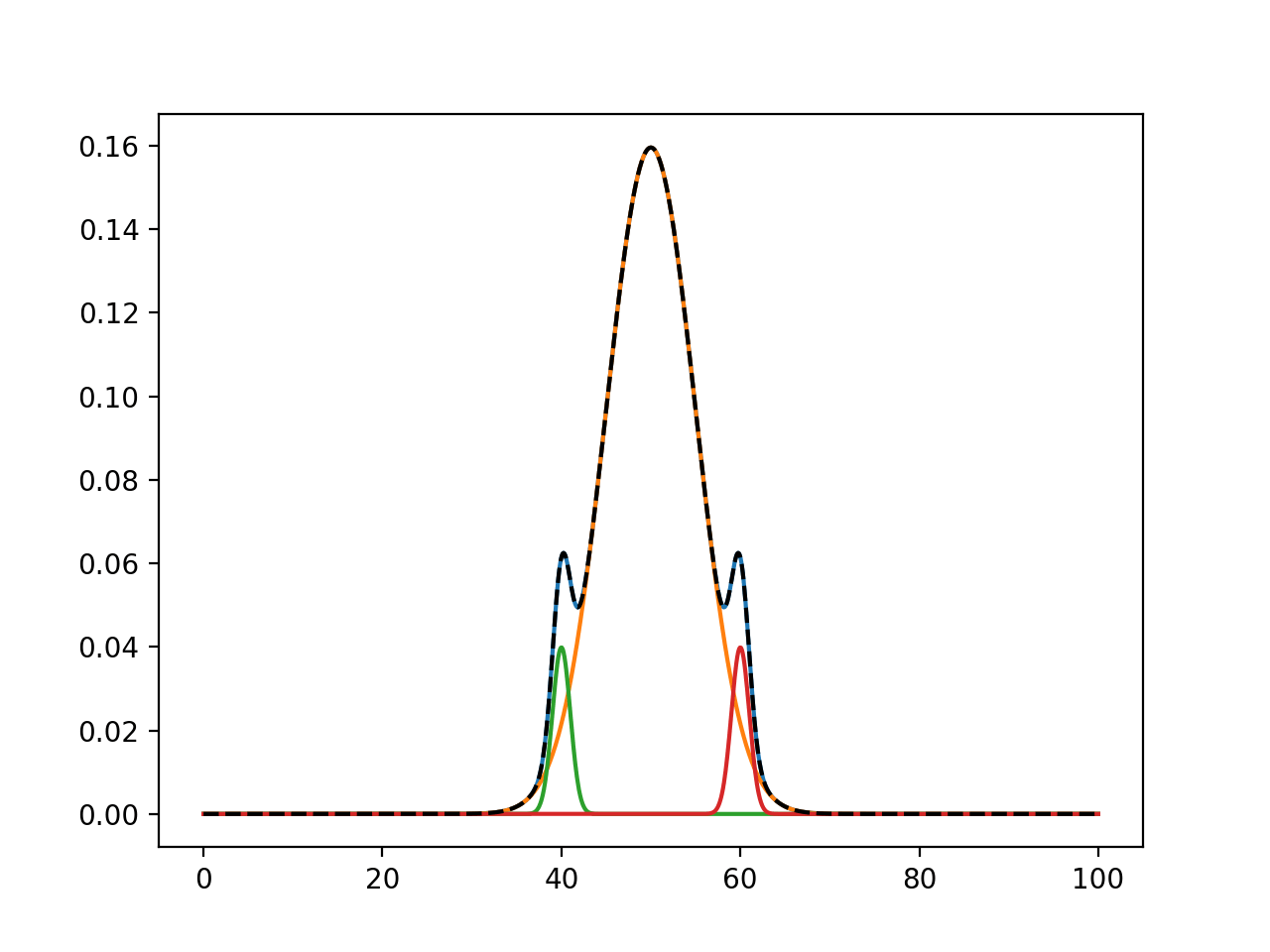
Основываясь на приведенном выше коде, следующее выполняет то, что я пытался сделать...
from lmfit.models import GaussianModel
gauss1 = GaussianModel(prefix='g1_')
gauss2 = GaussianModel(prefix='g2_')
gauss3 = GaussianModel(prefix='g3_')
gauss4 = GaussianModel(prefix='g4_')
gauss5 = GaussianModel(prefix='g5_')
gauss = [gauss1, gauss2, gauss3, gauss4, gauss5]
prefixes = ['g1_', 'g2_', 'g3_', 'g4_', 'g5_']
mod = np.sum(gauss[0:len(peaks)])
pars = mod.make_params()
for i, prefix in zip(range(0, len(peaks)), prefixes[0:len(peaks)]):
pars[prefix + 'center'].set(peaks[i])
init = mod.eval(pars, x=x_range)
out = mod.fit(fake, pars, x=x_range)
print(out.fit_report(min_correl=0.5))
out.plot_fit()
plt.show()