Подсчет количества клеток и ядра с помощью водораздела, скикит изображение
Я пытаюсь количественно оценить большой набор данных изображения. Каждое изображение имеет клетки и ядра. То, что должно быть сделано, представлено в схематической форме как "то, что мне нужно":
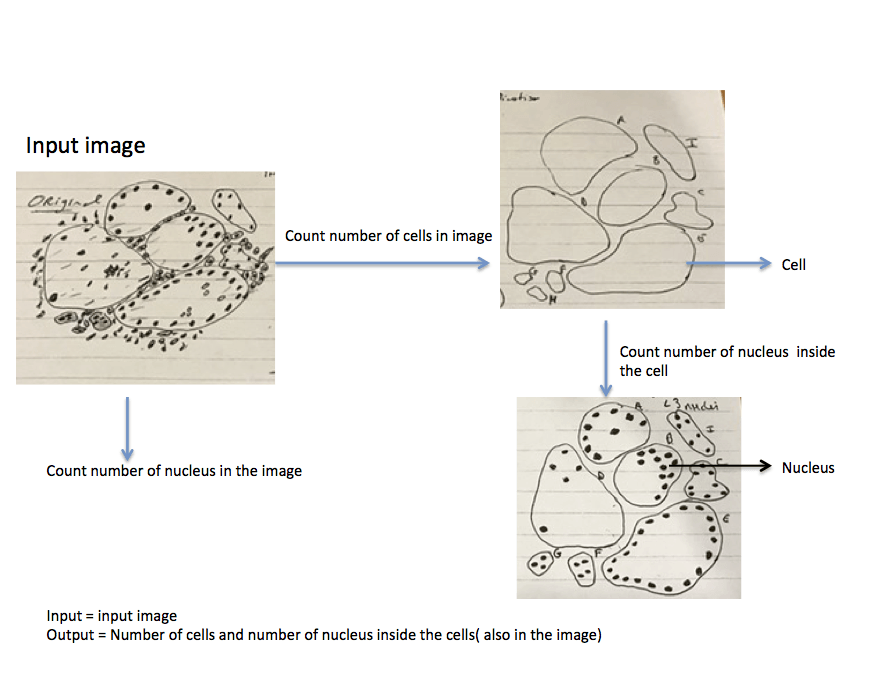
Пример изображения показан в "сыром изображении":

Я нашел программу алгоритма водораздела онлайн для подсчета клеток, но не смог подсчитать количество ядер внутри (и снаружи) клеток.
Вот программа, которую я использовал для подсчета ячеек из необработанного изображения
#import packages
import numpy as np
import matplotlib.pyplot as plt
from skimage import io, color, filters as filters
from scipy import ndimage
from skimage.morphology import watershed
from skimage.feature import peak_local_max
from skimage.measure import regionprops, label
import numpy as np
from scipy.ndimage import gaussian_filter
import matplotlib.pyplot as plt
from skimage import data
from skimage import img_as_float
from skimage.morphology import reconstruction
import skimage
from skimage import segmentation
%matplotlib inline
import matplotlib
#import image
from IPython.core.display import Image
Image(filename=('/Users/sasi/Desktop/image1.jpeg'))
# Find number of cells
image = color.rgb2gray(io.imread('/Users/sasi/Desktop/image1.jpeg'))
image = image < filters.threshold_otsu(image)
distance = ndimage.distance_transform_edt(image)
local_maxi = peak_local_max(distance, indices=False, footprint=np.ones((3, 3)), labels=image)
markers, num_features = ndimage.label(local_maxi)
labels = watershed(-distance, markers, mask=image)
regions = regionprops(labels)
regions = [r for r in regions if r.area > 60]
print('Number of cells:', len(regions) - 1)
Как мне подсчитать ядро внутри клетки, а также количество ядер во всем изображении? Кроме того, если у вас есть другая лучшая программа для подсчета клеток, пожалуйста, дайте мне знать.
1 ответ
import cv2
import numpy as np
from skimage.feature import peak_local_max
from skimage.morphology import watershed
from scipy import ndimage
image=cv2.imread("Da0003.jpg")
gray_image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)
sigma=2
gauss_img = cv2.GaussianBlur(gray_image,(0,0),1.6*sigma,0)
main_img = cv2.GaussianBlur(gray_image,(0,0),sigma,0)
ret,threshold_img = cv2.threshold(main_img,127,255,cv2.THRESH_BINARY)
threshold=cv2.bitwise_not(threshold_img)
D = ndimage.distance_transform_edt(threshold)
localMax = peak_local_max(D, indices=False, min_distance=10,
labels=threshold)
markers = ndimage.label(localMax, structure=np.ones((3, 3)))[0]
labels = watershed(-D, markers, mask=threshold)
for label in np.unique(labels):
# if the label is zero, we are examining the 'background'
# so simply ignore it
if label == 0:
continue
# otherwise, allocate memory for the label region and draw
# it on the mask
x,y,_=image.shape
image_copy=image
mask = np.zeros((x,y), dtype="uint8")
mask[labels == label] = 255
cnts = cv2.findContours(mask.copy(), cv2.RETR_EXTERNAL,
cv2.CHAIN_APPROX_SIMPLE)[-2]
c = max(cnts, key=cv2.contourArea)
cv2.drawContours(image_copy, [c], -1, (0, 255, 255), 1)
#print(label)
cv2.imwrite("_watershead"+".jpg", image_copy)
print("count:"+str(label))