Параллельная обработка в R doParallel foreach
Я написал скрипт, который работает нормально, но он не выполняет параллельную обработку. Я попытался изменить ядра с 3 на 16, но скорость, с которой генерировались данные, не изменилась. Может кто-нибудь сообщить мне, что я делаю неправильно и как я могу заставить это работать?
setwd("E:/Infections")
if (!require("pacman")) install.packages("pacman")
pacman::p_load(lakemorpho,rgdal,maptools,sp,doParallel,foreach,
doParallel)
cl <- makeCluster(5, outfile="E:/Infections/debug.txt")
registerDoParallel(cl)
x<-readOGR("E:/Infections/ByHUC6","Kodiak")
x_lake_length<-vector("numeric",length = nrow(x))
for(i in 1:nrow(x)){
tmp<-lakeMorphoClass(x[i,],NULL,NULL,NULL)
x_lake_length[i]<-lakeMaxLength(tmp,200)
print(i)
Sys.sleep(0.1)
}
df_Kodiak <- data.frame(x_lake_length)
write.table(df_Kodiak,file="E:/Infections/ByHUC6/Kodiak.csv",row.names=TRUE,col.names=TRUE, sep=",")
1 ответ
Хорошо, я думаю, что я получил это, вызвав foreach а также %dopar%:
# Libraries ---------------------------------------------------------------
if (!require("pacman")) install.packages("pacman")
pacman::p_load(lakemorpho,rgdal,maptools,sp,doParallel,foreach,
doParallel)
# Data --------------------------------------------------------------------
ogrDrivers()
dsn <- system.file("vectors", package = "rgdal")[1]
ogrListLayers(dsn)
ogrInfo(dsn=dsn, layer="trin_inca_pl03")
owd <- getwd()
setwd(dsn)
ogrInfo(dsn="trin_inca_pl03.shp", layer="trin_inca_pl03")
setwd(owd)
x <- readOGR(dsn=dsn, layer="trin_inca_pl03")
summary(x)
# HPC ---------------------------------------------------------------------
cores_2_use <- detectCores() - 4
cl <- makeCluster(cores_2_use, useXDR = F)
clusterSetRNGStream(cl, 9956)
registerDoParallel(cl, cores_2_use)
# Analysis ----------------------------------------------------------------
myfun <- function(x,i){tmp<-lakeMorphoClass(x[i,],NULL,NULL,NULL)
x_lake_length<-vector("numeric",length = nrow(x))
x_lake_length[i]<-lakeMaxLength(tmp,200)
print(i)
Sys.sleep(0.1)}
foreach(i = 1:nrow(x),.combine=cbind,.packages=c("lakemorpho","rgdal")) %dopar% (
myfun(x,i)
)
df_Kodiak <- data.frame(x_lake_length)
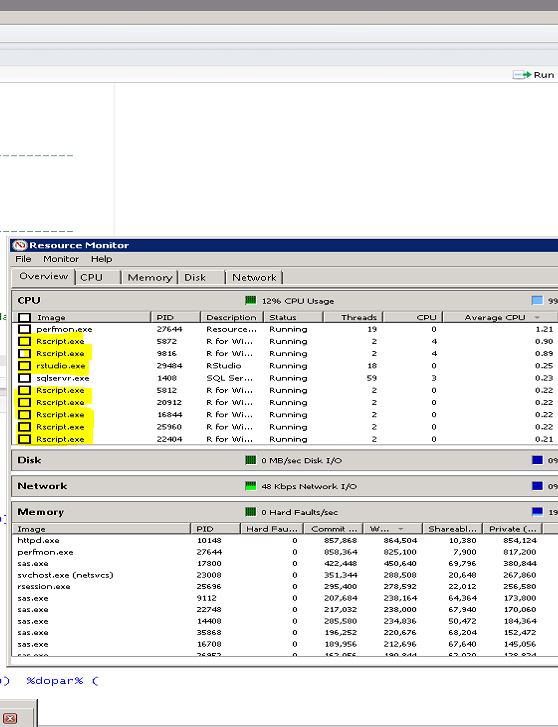
Как вы можете видеть на скриншоте ниже, это создает армию процессов Rscript.exe, использующих 20 из 24 процессорных ядер. Конечно, примеры данных, которые я использовал, маленькие, поэтому им не нужны все эти ядра, но они должны служить подтверждением концепции.
Я никогда не превышаю это соотношение, потому что если вы используете 100% всех процессорных ядер, иногда случаются плохие вещи, и другие пользователи сервера могут вас не устраивать.