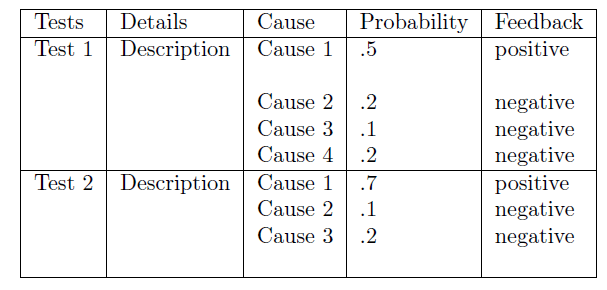
Таблица в г с несколькими подстроками и записи в PDF
Можно ли создать таблицу в R с несколькими строками, соответствующими одной строке? И записать таблицу результатов в PDF. Пример таблицы приведен ниже. 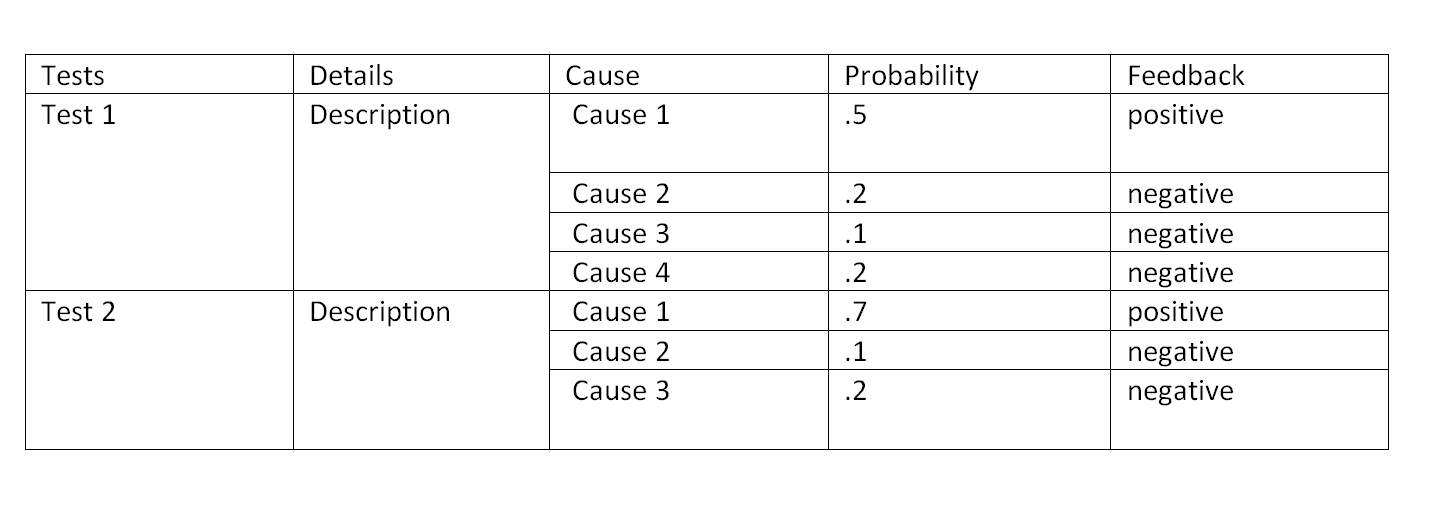
Можно ли объединить две отдельные таблицы так, как показано на рисунке. Пример кода для двух таблиц приведен ниже
tab1="Test Description
1 test1 description
2 test2 description"
table1 <-read.table(text = tab1,header = TRUE)
tab21="Cause Probability Feedback
1 cause1 .5 positive
2 Cause2 .2 negative
3 Cause3 .1 negative
4 Cause4 .2 negative"
table2 <-read.table(text = tab21,header = TRUE)
tab22="Cause Probability Feedback
1 cause1 .7 positive
2 Cause2 .2 negative
3 Cause3 .1 negative"
table3 <-read.table(text = tab22,header = TRUE)
2 ответа
Это немного сложно, но я бы воспользовался тем фактом, что ячейки с NA печатаются как пустые функцией print.xtable. Ячейки на самом деле не "объединены", но похоже, что содержимое выровнено по верху.
В основном шаги:
- Создайте подходящий data.frame в R
- Распечатайте это как.tex-совместимую таблицу, используя print.xtable из пакета 'xtable'
- Используйте Sweave/knitr/etc для создания подходящего.tex
- tools:: texi2pdf преобразует ваш.tex в подходящий.pdf
Вот файлы, которые вам нужны только для запуска RunSweave.R в вашем терминале R (и убедитесь, что у вас установлен LaTeX вместе с необходимыми пакетами, то есть 'xtable и файлы в одной папке; это было выполнено в Windows),
Файл StackExampleCode.R:
# StackExampleCode.R
library(xtable)
# A work-around by setting rows in the multi-row to NA after the initial top-justified line
header <- data.frame(test = "Tests", det = "Details", cause = "Cause", prob = "Probability", fb = "Feedback")
# Filling the fields for these is something you'd probably want to do in R in a more sophisticated manner
test1 <- data.frame(
test = c("Test 1", NA, NA, NA, NA),
det = c("Description", NA, NA, NA, NA),
cause = c("Cause 1", NA, paste("Cause", 2:4)),
prob = c(".5", NA, ".2", ".1", ".2"),
fb = c("positive", NA, "negative", "negative", "negative")
)
test2 <- data.frame(
test = c("Test 2", NA, NA, NA),
det = c("Description", NA, NA, NA),
cause = c(paste("Cause", 1:3), NA),
prob = c(".7", ".1", ".2", NA),
fb = c("positive", "negative", "negative", NA)
)
# Bind it all together, you probably want something similar if it's automatic data you're generating
tab <- rbind(header, test1, test2)
Файл StackExampleRnw.Rnw:
% StackExampleRnw.Rnw
% Note the different comment char, writing .tex here
\documentclass{article}
\begin{document}
<<echo=FALSE, results=tex>>=
# Printing the table
print(
xtable(tab,
align = "|l|l|l|l|l|l|" # Create the desired vertical lines and text alignments ala LaTeX; left align with vertical lines in-between each column)
),
add.to.row = list( # Add horizontal lines to correct spots, should be adjusted according to the desired data
pos = list(-1, 1, 6, nrow(tab)),
command = c("\\hline \n", "\\hline \n", "\\hline \n", "\\hline \n") # Horizontal lines and a neater formatting of output using a linechange
),
include.rownames = FALSE, # Don't include the rownames (which would be just numbers)
include.colnames = FALSE, # Don't include the rownames, these were already included as if it was an ordinary table row
hline.after = NULL # Suppress the empty horizontal line that is intended for an automated caption
)
@
\end{document}
Файл RunSweave.R:
# RunSweave.R
# Run the code
source("StackExampleCode.R")
# Bundle R code with LaTeX
Sweave("StackExampleRnw.Rnw")
# .tex -> .pdf
tools::texi2pdf("StackExampleRnw.tex")
Вот как это выглядит для меня в StackExampleRnw.pdf:
Кроме того, вы можете напрямую обратиться к таблице в.tex в файле StackExampleRnw.tex и выполнить дополнительное форматирование, если вам это удобно. Выше не требуется никаких дополнительных действий в.tex, но вы должны убедиться, что вы поместили горизонтальные линии и NA в правильные места.
Если вам не нравится.tex, у функции print.xtable есть много параметров, доступных для дальнейшего форматирования. Если частичные горизонтальные линии действительно важны для вас в трех столбцах справа, я бы, вероятно, разделил их на две таблицы, а затем просто склеил их по горизонтали и получил бы правильную горизонтальную линию в каждой строке.
Я хотел бы сделать это в pixiedust путем объединения некоторых ячеек, но у меня есть pixiedust это не учитывает вертикальные границы на объединенных строках. Обходной путь использует подход Teemu по настройке ячеек, которые мы не хотим просматривать NA и направление их для печати в виде пустых символов.
library(dplyr)
library(pixiedust)
table2$Test <- "test1"
table3$Test <- "test2"
bind_rows(
right_join(table1, table2),
right_join(table1, table3)
) %>%
mutate(Description = as.character(Description)) %>%
group_by(Test) %>%
mutate(Description = ifelse(duplicated(Description), NA, Description)) %>%
ungroup() %>%
mutate(Test = ifelse(duplicated(Test), NA, Test))%>%
dust(float = FALSE) %>%
sprinkle(cols = 1:2,
rows = c(4, 7),
border = "bottom") %>%
sprinkle(cols = 1:2,
rows = 1,
border = "top") %>%
sprinkle(cols = 1:2,
border = "left",
na.string = "") %>%
medley_all_borders(cols = 3:5) %>%
medley_all_borders(part = "head") %>%
sprinkle_print_method("latex")
Полный рабочий файл RMD:
---
title: "Untitled"
output: pdf_document
header-includes:
- \usepackage{amssymb}
- \usepackage{arydshln}
- \usepackage{caption}
- \usepackage{graphicx}
- \usepackage{hhline}
- \usepackage{longtable}
- \usepackage{multirow}
- \usepackage[dvipsnames,table]{xcolor}
---
```{r, echo = FALSE, message = FALSE, warning = FALSE}
library(dplyr)
library(pixiedust)
tab1="Test Description
1 test1 description
2 test2 description"
table1 <-read.table(text = tab1,header = TRUE)
tab21="Cause Probability Feedback
1 cause1 .5 positive
2 Cause2 .2 negative
3 Cause3 .1 negative
4 Cause4 .2 negative"
table2 <-read.table(text = tab21,header = TRUE)
tab22="Cause Probability Feedback
1 cause1 .7 positive
2 Cause2 .2 negative
3 Cause3 .1 negative"
table3 <-read.table(text = tab22,header = TRUE)
```
```{r, echo = FALSE, message = FALSE, warning = FALSE}
table2$Test <- "test1"
table3$Test <- "test2"
bind_rows(
right_join(table1, table2),
right_join(table1, table3)
) %>%
mutate(Description = as.character(Description)) %>%
group_by(Test) %>%
mutate(Description = ifelse(duplicated(Description), NA, Description)) %>%
ungroup() %>%
mutate(Test = ifelse(duplicated(Test), NA, Test))%>%
dust(float = FALSE) %>%
sprinkle(cols = 1:2,
rows = c(4, 7),
border = "bottom") %>%
sprinkle(cols = 1:2,
rows = 1,
border = "top") %>%
sprinkle(cols = 1:2,
border = "left",
na.string = "") %>%
medley_all_borders(cols = 3:5) %>%
medley_all_borders(part = "head")
```