R: замена отрицательных значений на ноль
Мы хотим установить все значения в массиве ноль, которые являются отрицательными. Я перепробовал много вещей, но еще не достиг рабочего решения. Я думал о цикле for с условием, но это, похоже, не работает.
#pred_precipitation is our array
pred_precipitation <-rnorm(25,2,4)
for (i in nrow(pred_precipitation))
{
if (pred_precipitation[i]<0) {pred_precipitation[i] = 0}
else{pred_precipitation[i] = pred_precipitation[i]}
}
Спасибо за ваши подсказки!
5 ответов
Спасибо за воспроизводимый пример. Это довольно простой R материал. Вы можете назначить выбранным элементам вектора (обратите внимание, что массив имеет размеры, а вы дали вектор, а не массив):
> pred_precipitation[pred_precipitation<0] <- 0
> pred_precipitation
[1] 1.2091281 0.0000000 7.7665555 0.0000000 0.0000000 0.0000000 0.5151504 0.0000000 1.8281251
[10] 0.5098688 2.8370263 0.4895606 1.5152191 4.1740177 7.1527742 2.8992215 4.5322934 6.7180530
[19] 0.0000000 1.1914052 3.6152333 0.0000000 0.3778717 0.0000000 1.4940469
Контрольные войны!
@James нашел еще более быстрый метод и оставил его в комментарии. Я проголосовал за него хотя бы потому, что знаю, что его победа будет недолгой.
Сначала я пытаюсь скомпилировать, но это никому не помогает:
p <- rnorm(10000)
gsk3 <- function(x) { x[x<0] <- 0; x }
jmsigner <- function(x) ifelse(x<0, 0, x)
joshua <- function(x) pmin(x,0)
james <- function(x) (abs(x)+x)/2
library(compiler)
gsk3.c <- cmpfun(gsk3)
jmsigner.c <- cmpfun(jmsigner)
joshua.c <- cmpfun(joshua)
james.c <- cmpfun(james)
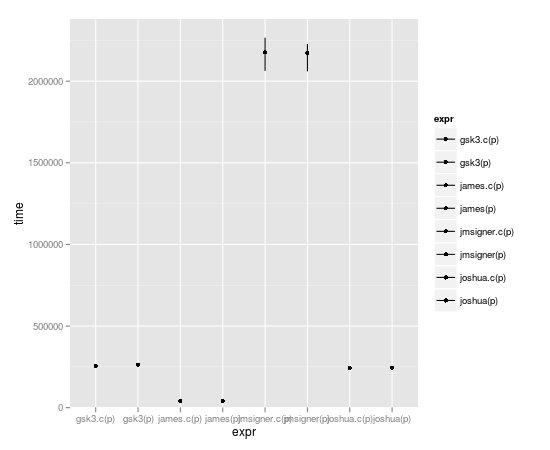
microbenchmark(joshua(p),joshua.c(p),gsk3(p),gsk3.c(p),jmsigner(p),james(p),jmsigner.c(p),james.c(p))
expr min lq median uq max
1 gsk3.c(p) 251.782 255.0515 266.8685 269.5205 457.998
2 gsk3(p) 256.262 261.6105 270.7340 281.3560 2940.486
3 james.c(p) 38.418 41.3770 43.3020 45.6160 132.342
4 james(p) 38.934 42.1965 43.5700 47.2085 4524.303
5 jmsigner.c(p) 2047.739 2145.9915 2198.6170 2291.8475 4879.418
6 jmsigner(p) 2047.502 2169.9555 2258.6225 2405.0730 5064.334
7 joshua.c(p) 237.008 244.3570 251.7375 265.2545 376.684
8 joshua(p) 237.545 244.8635 255.1690 271.9910 430.566
Но ждать! Дирк написал эту вещь Rcpp. Может ли полный C++ некомпетентный прочитать его документ JSS, адаптировать его пример и написать самую быструю функцию из них всех? Оставайтесь с нами, дорогие слушатели.
library(inline)
cpp_if_src <- '
Rcpp::NumericVector xa(a);
int n_xa = xa.size();
for(int i=0; i < n_xa; i++) {
if(xa[i]<0) xa[i] = 0;
}
return xa;
'
cpp_if <- cxxfunction(signature(a="numeric"), cpp_if_src, plugin="Rcpp")
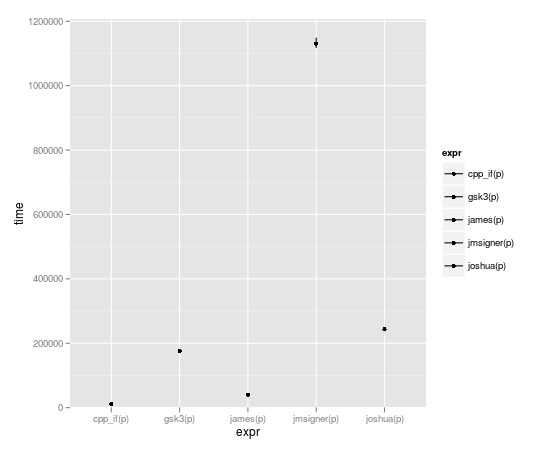
microbenchmark(joshua(p),joshua.c(p),gsk3(p),gsk3.c(p),jmsigner(p),james(p),jmsigner.c(p),james.c(p), cpp_if(p))
expr min lq median uq max
1 cpp_if(p) 8.233 10.4865 11.6000 12.4090 69.512
2 gsk3(p) 170.572 172.7975 175.0515 182.4035 2515.870
3 james(p) 37.074 39.6955 40.5720 42.1965 2396.758
4 jmsigner(p) 1110.313 1118.9445 1133.4725 1164.2305 65942.680
5 joshua(p) 237.135 240.1655 243.3990 250.3660 2597.429
Это утвердительно, капитан.
Это изменяет ввод p даже если вы не назначаете на это. Если вы хотите избежать такого поведения, вы должны клонировать:
cpp_ifclone_src <- '
Rcpp::NumericVector xa(Rcpp::clone(a));
int n_xa = xa.size();
for(int i=0; i < n_xa; i++) {
if(xa[i]<0) xa[i] = 0;
}
return xa;
'
cpp_ifclone <- cxxfunction(signature(a="numeric"), cpp_ifclone_src, plugin="Rcpp")
Что, к сожалению, убивает преимущество в скорости.
Я хотел бы использовать pmax так как ifelse иногда может быть немного медленным, и замена подмножества создает дополнительный вектор (который может быть проблемой с большими наборами данных).
set.seed(21)
pred_precipitation <- rnorm(25,2,4)
p <- pmax(pred_precipitation,0)
Замена подмножества, безусловно, самая быстрая, хотя:
library(rbenchmark)
gsk3 <- function(x) { x[x<0] <- 0; x }
jmsigner <- function(x) ifelse(x<0, 0, x)
joshua <- function(x) pmin(x,0)
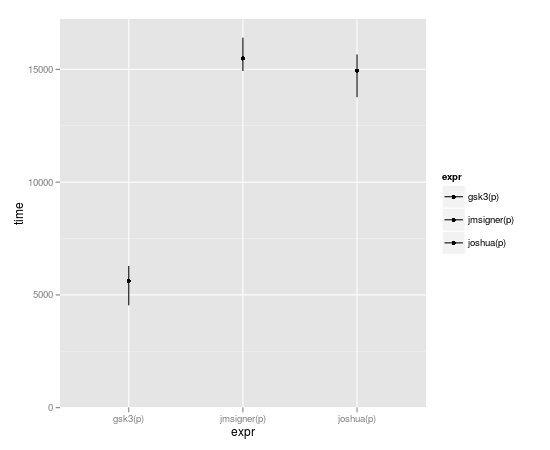
benchmark(joshua(p), gsk3(p), jmsigner(p), replications=10000, order="relative")
test replications elapsed relative user.self sys.self
2 gsk3(p) 10000 0.215 1.000000 0.216 0.000
1 joshua(p) 10000 0.444 2.065116 0.416 0.016
3 jmsigner(p) 10000 0.656 3.051163 0.652 0.000
В качестве альтернативы вы также можете использовать ifelse:
ifelse(pred_precipitation < 0, 0, pred_precipitation)
Если ваш основной объект - это тиббл или фрейм данных, вы также можете использовать пакет tidy. По сравнению с заменой, предложенной Ари Б. Фридманом, замену можно было бы написать "на лету" и скомбинировать с другими мутациями.
Пример использования dplyr и %>% трубы будут выглядеть так:
df %>% mutate(varA = if_else(varA < 0, 0, varA))
Вы можете добавить дополнительные мутации (т. Е. Новые переменные) в mutate()заявление. Преимущество, которое я вижу в этом типе кодирования, заключается в том, что вы не рискуете пропустить или повторно выполнить отдельный шаг преобразования, поскольку все они сгруппированы в одном операторе. Например, добавив%>% View()в RStudio вы уже можете предварительно просмотреть результат. Однако результат пока нигде не сохраняется ("на лету"). Таким образом вы сохраняете чистоту пространства имен / среды при изменении кода.
Чтобы ответить @ah bon, если у нас есть несколько столбцов, т. Е. И varA, и varB должны заменить свои отрицательные значения на 0, мы можем использовать mutate(across()), чтобы избежать повторения оператора ifelse.
Если varA и varB соседние:
df %>%
mutate(across(varA:varB, ~ ifelse(.x < 0, 0, .x)))
Если это не так:
df %>%
mutate(across(c(varA, varB), ~ ifelse(.x < 0, 0, .x)))