Подгонять эмпирическое распределение к теоретическому с помощью Scipy (Python)?
ВВЕДЕНИЕ: У меня есть список из более чем 30 000 значений в диапазоне от 0 до 47, например [0,0,0,0,..,1,1,1,1,...,2,2,2,2,..., 47 и т. Д.], Который является непрерывным распределением.
ПРОБЛЕМА. Исходя из моего распределения, я хотел бы рассчитать значение p (вероятность увидеть большие значения) для любого заданного значения. Например, как вы можете видеть, значение p для 0 будет приближаться к 1, а значение p для больших чисел будет стремиться к 0.
Я не знаю, прав ли я, но для определения вероятностей я думаю, что мне нужно согласовать мои данные с теоретическим распределением, наиболее подходящим для описания моих данных. Я предполагаю, что для определения наилучшей модели необходим какой-то тест на пригодность.
Есть ли способ реализовать такой анализ в Python (Scipy или Numpy)? Не могли бы вы привести примеры?
Спасибо!
13 ответов
Распределительная арматура с ошибкой суммы квадратов (SSE)
Это обновление и модификация ответа Саулло, который использует полный список текущих scipy.stats распределения и возвращает распределение с наименьшим SSE между гистограммой распределения и гистограммой данных.
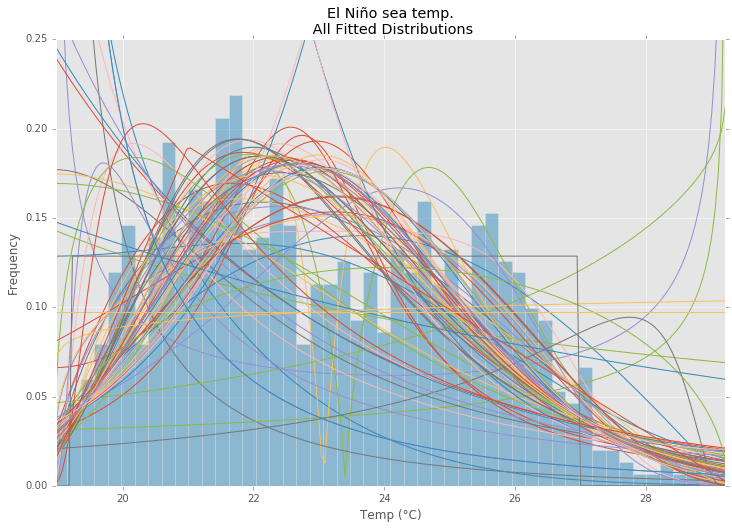
Пример примерки
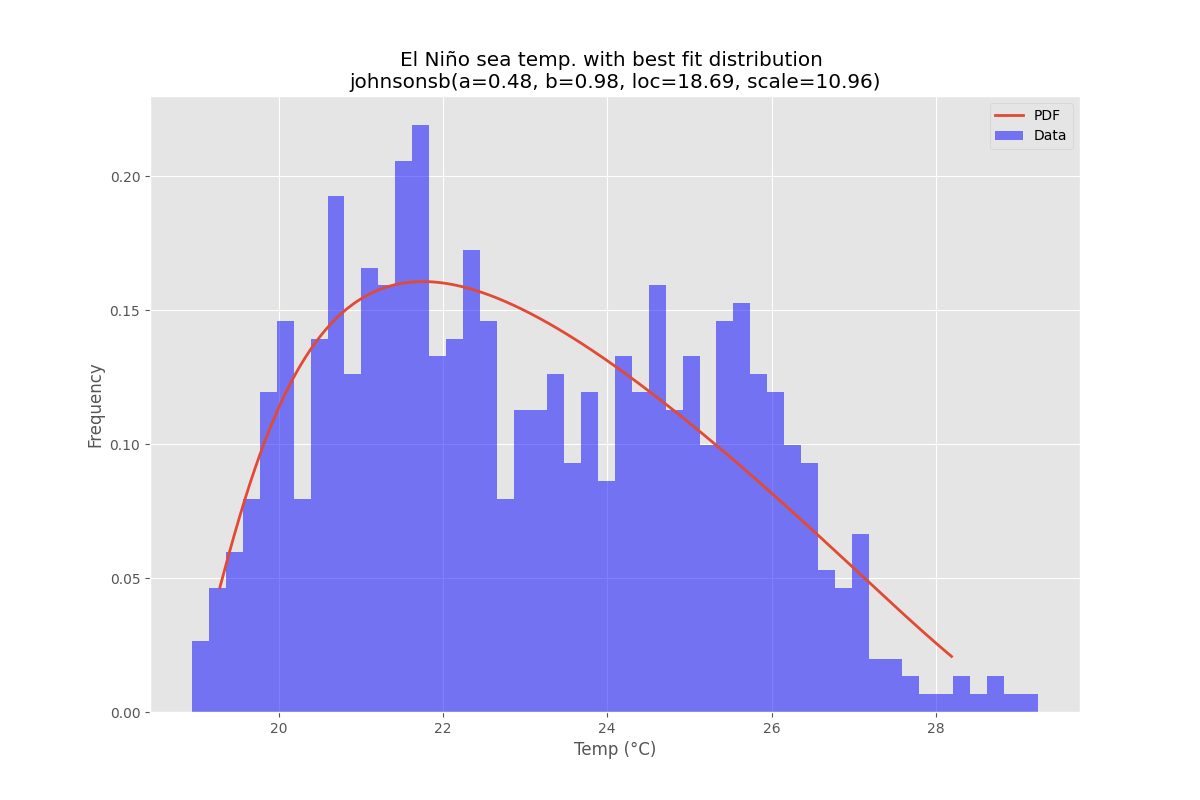
Использование набора данных Эль-Ниньо изstatsmodels, распределения соответствуют, и ошибка определена. Распределение с наименьшей ошибкой возвращается.
Все Распределения
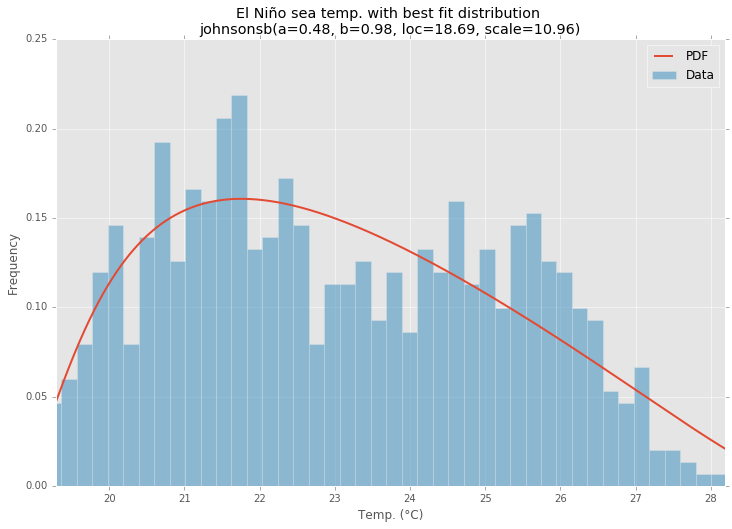
Best Fit Distribution
Пример кода
%matplotlib inline
import warnings
import numpy as np
import pandas as pd
import scipy.stats as st
import statsmodels as sm
import matplotlib
import matplotlib.pyplot as plt
matplotlib.rcParams['figure.figsize'] = (16.0, 12.0)
matplotlib.style.use('ggplot')
# Create models from data
def best_fit_distribution(data, bins=200, ax=None):
"""Model data by finding best fit distribution to data"""
# Get histogram of original data
y, x = np.histogram(data, bins=bins, density=True)
x = (x + np.roll(x, -1))[:-1] / 2.0
# Distributions to check
DISTRIBUTIONS = [
st.alpha,st.anglit,st.arcsine,st.beta,st.betaprime,st.bradford,st.burr,st.cauchy,st.chi,st.chi2,st.cosine,
st.dgamma,st.dweibull,st.erlang,st.expon,st.exponnorm,st.exponweib,st.exponpow,st.f,st.fatiguelife,st.fisk,
st.foldcauchy,st.foldnorm,st.frechet_r,st.frechet_l,st.genlogistic,st.genpareto,st.gennorm,st.genexpon,
st.genextreme,st.gausshyper,st.gamma,st.gengamma,st.genhalflogistic,st.gilbrat,st.gompertz,st.gumbel_r,
st.gumbel_l,st.halfcauchy,st.halflogistic,st.halfnorm,st.halfgennorm,st.hypsecant,st.invgamma,st.invgauss,
st.invweibull,st.johnsonsb,st.johnsonsu,st.ksone,st.kstwobign,st.laplace,st.levy,st.levy_l,st.levy_stable,
st.logistic,st.loggamma,st.loglaplace,st.lognorm,st.lomax,st.maxwell,st.mielke,st.nakagami,st.ncx2,st.ncf,
st.nct,st.norm,st.pareto,st.pearson3,st.powerlaw,st.powerlognorm,st.powernorm,st.rdist,st.reciprocal,
st.rayleigh,st.rice,st.recipinvgauss,st.semicircular,st.t,st.triang,st.truncexpon,st.truncnorm,st.tukeylambda,
st.uniform,st.vonmises,st.vonmises_line,st.wald,st.weibull_min,st.weibull_max,st.wrapcauchy
]
# Best holders
best_distribution = st.norm
best_params = (0.0, 1.0)
best_sse = np.inf
# Estimate distribution parameters from data
for distribution in DISTRIBUTIONS:
# Try to fit the distribution
try:
# Ignore warnings from data that can't be fit
with warnings.catch_warnings():
warnings.filterwarnings('ignore')
# fit dist to data
params = distribution.fit(data)
# Separate parts of parameters
arg = params[:-2]
loc = params[-2]
scale = params[-1]
# Calculate fitted PDF and error with fit in distribution
pdf = distribution.pdf(x, loc=loc, scale=scale, *arg)
sse = np.sum(np.power(y - pdf, 2.0))
# if axis pass in add to plot
try:
if ax:
pd.Series(pdf, x).plot(ax=ax)
end
except Exception:
pass
# identify if this distribution is better
if best_sse > sse > 0:
best_distribution = distribution
best_params = params
best_sse = sse
except Exception:
pass
return (best_distribution.name, best_params)
def make_pdf(dist, params, size=10000):
"""Generate distributions's Probability Distribution Function """
# Separate parts of parameters
arg = params[:-2]
loc = params[-2]
scale = params[-1]
# Get sane start and end points of distribution
start = dist.ppf(0.01, *arg, loc=loc, scale=scale) if arg else dist.ppf(0.01, loc=loc, scale=scale)
end = dist.ppf(0.99, *arg, loc=loc, scale=scale) if arg else dist.ppf(0.99, loc=loc, scale=scale)
# Build PDF and turn into pandas Series
x = np.linspace(start, end, size)
y = dist.pdf(x, loc=loc, scale=scale, *arg)
pdf = pd.Series(y, x)
return pdf
# Load data from statsmodels datasets
data = pd.Series(sm.datasets.elnino.load_pandas().data.set_index('YEAR').values.ravel())
# Plot for comparison
plt.figure(figsize=(12,8))
ax = data.plot(kind='hist', bins=50, normed=True, alpha=0.5, color=plt.rcParams['axes.color_cycle'][1])
# Save plot limits
dataYLim = ax.get_ylim()
# Find best fit distribution
best_fit_name, best_fit_params = best_fit_distribution(data, 200, ax)
best_dist = getattr(st, best_fit_name)
# Update plots
ax.set_ylim(dataYLim)
ax.set_title(u'El Niño sea temp.\n All Fitted Distributions')
ax.set_xlabel(u'Temp (°C)')
ax.set_ylabel('Frequency')
# Make PDF with best params
pdf = make_pdf(best_dist, best_fit_params)
# Display
plt.figure(figsize=(12,8))
ax = pdf.plot(lw=2, label='PDF', legend=True)
data.plot(kind='hist', bins=50, normed=True, alpha=0.5, label='Data', legend=True, ax=ax)
param_names = (best_dist.shapes + ', loc, scale').split(', ') if best_dist.shapes else ['loc', 'scale']
param_str = ', '.join(['{}={:0.2f}'.format(k,v) for k,v in zip(param_names, best_fit_params)])
dist_str = '{}({})'.format(best_fit_name, param_str)
ax.set_title(u'El Niño sea temp. with best fit distribution \n' + dist_str)
ax.set_xlabel(u'Temp. (°C)')
ax.set_ylabel('Frequency')
В SciPy 0.12.0 реализовано 82 функции распределения. Вы можете проверить, как некоторые из них соответствуют вашим данным, используя их fit() метод. Проверьте код ниже для более подробной информации:
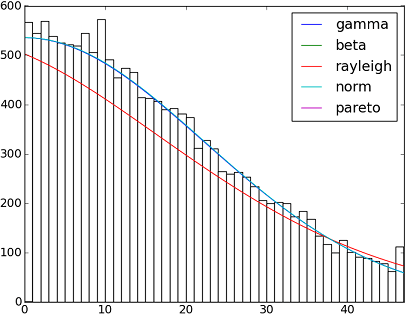
import matplotlib.pyplot as plt
import scipy
import scipy.stats
size = 30000
x = scipy.arange(size)
y = scipy.int_(scipy.round_(scipy.stats.vonmises.rvs(5,size=size)*47))
h = plt.hist(y, bins=range(48), color='w')
dist_names = ['gamma', 'beta', 'rayleigh', 'norm', 'pareto']
for dist_name in dist_names:
dist = getattr(scipy.stats, dist_name)
param = dist.fit(y)
pdf_fitted = dist.pdf(x, *param[:-2], loc=param[-2], scale=param[-1]) * size
plt.plot(pdf_fitted, label=dist_name)
plt.xlim(0,47)
plt.legend(loc='upper right')
plt.show()
Рекомендации:
- Распределение фитингов, качество посадки, значение p. Возможно ли это сделать с помощью Scipy (Python)?
- Распределительная арматура со Scipy
А вот список с именами всех функций распределения, доступных в Scipy 0.12.0 (VI):
dist_names = [ 'alpha', 'anglit', 'arcsine', 'beta', 'betaprime', 'bradford', 'burr', 'cauchy', 'chi', 'chi2', 'cosine', 'dgamma', 'dweibull', 'erlang', 'expon', 'exponweib', 'exponpow', 'f', 'fatiguelife', 'fisk', 'foldcauchy', 'foldnorm', 'frechet_r', 'frechet_l', 'genlogistic', 'genpareto', 'genexpon', 'genextreme', 'gausshyper', 'gamma', 'gengamma', 'genhalflogistic', 'gilbrat', 'gompertz', 'gumbel_r', 'gumbel_l', 'halfcauchy', 'halflogistic', 'halfnorm', 'hypsecant', 'invgamma', 'invgauss', 'invweibull', 'johnsonsb', 'johnsonsu', 'ksone', 'kstwobign', 'laplace', 'logistic', 'loggamma', 'loglaplace', 'lognorm', 'lomax', 'maxwell', 'mielke', 'nakagami', 'ncx2', 'ncf', 'nct', 'norm', 'pareto', 'pearson3', 'powerlaw', 'powerlognorm', 'powernorm', 'rdist', 'reciprocal', 'rayleigh', 'rice', 'recipinvgauss', 'semicircular', 't', 'triang', 'truncexpon', 'truncnorm', 'tukeylambda', 'uniform', 'vonmises', 'wald', 'weibull_min', 'weibull_max', 'wrapcauchy']
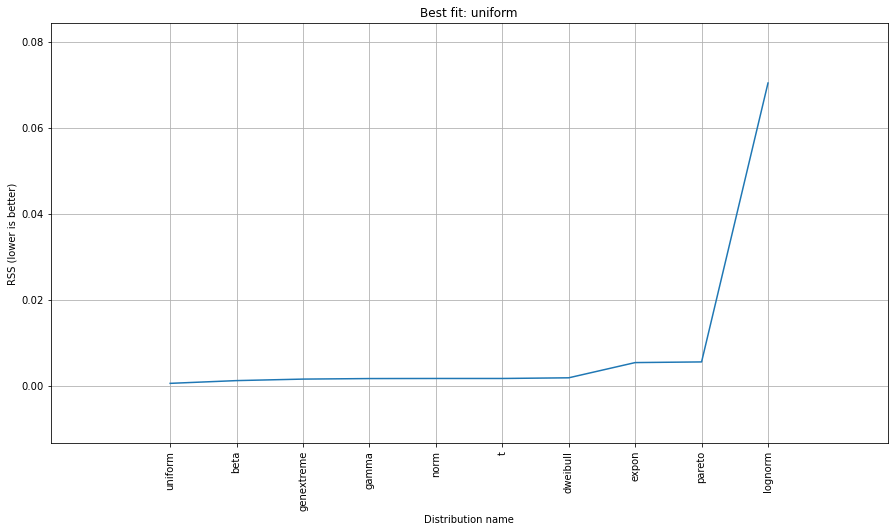
Попробуйте distfit библиотека.
pip установить distfit
# Create 1000 random integers, value between [0-50]
X = np.random.randint(0, 50,1000)
# Retrieve P-value for y
y = [0,10,45,55,100]
# From the distfit library import the class distfit
from distfit import distfit
# Initialize.
# Set any properties here, such as alpha.
# The smoothing can be of use when working with integers. Otherwise your histogram
# may be jumping up-and-down, and getting the correct fit may be harder.
dist = distfit(alpha=0.05, smooth=10)
# Search for best theoretical fit on your empirical data
dist.fit_transform(X)
> [distfit] >fit..
> [distfit] >transform..
> [distfit] >[norm ] [RSS: 0.0037894] [loc=23.535 scale=14.450]
> [distfit] >[expon ] [RSS: 0.0055534] [loc=0.000 scale=23.535]
> [distfit] >[pareto ] [RSS: 0.0056828] [loc=-384473077.778 scale=384473077.778]
> [distfit] >[dweibull ] [RSS: 0.0038202] [loc=24.535 scale=13.936]
> [distfit] >[t ] [RSS: 0.0037896] [loc=23.535 scale=14.450]
> [distfit] >[genextreme] [RSS: 0.0036185] [loc=18.890 scale=14.506]
> [distfit] >[gamma ] [RSS: 0.0037600] [loc=-175.505 scale=1.044]
> [distfit] >[lognorm ] [RSS: 0.0642364] [loc=-0.000 scale=1.802]
> [distfit] >[beta ] [RSS: 0.0021885] [loc=-3.981 scale=52.981]
> [distfit] >[uniform ] [RSS: 0.0012349] [loc=0.000 scale=49.000]
# Best fitted model
best_distr = dist.model
print(best_distr)
# Uniform shows best fit, with 95% CII (confidence intervals), and all other parameters
> {'distr': <scipy.stats._continuous_distns.uniform_gen at 0x16de3a53160>,
> 'params': (0.0, 49.0),
> 'name': 'uniform',
> 'RSS': 0.0012349021241149533,
> 'loc': 0.0,
> 'scale': 49.0,
> 'arg': (),
> 'CII_min_alpha': 2.45,
> 'CII_max_alpha': 46.55}
# Ranking distributions
dist.summary
# Plot the summary of fitted distributions
dist.plot_summary()
# Make prediction on new datapoints based on the fit
dist.predict(y)
# Retrieve your pvalues with
dist.y_pred
# array(['down', 'none', 'none', 'up', 'up'], dtype='<U4')
dist.y_proba
array([0.02040816, 0.02040816, 0.02040816, 0. , 0. ])
# Or in one dataframe
dist.df
# The plot function will now also include the predictions of y
dist.plot()
Обратите внимание, что в этом случае все точки будут значимы из-за равномерного распределения. При необходимости вы можете фильтровать с помощью dist.y_pred.
fit() Метод, упомянутый @Saullo Castro, обеспечивает оценку максимального правдоподобия (MLE). Лучшее распределение ваших данных - это то, которое дает вам самое высокое, может быть определено несколькими различными способами: например,
1, тот, который дает вам наибольшую вероятность журнала.
2, тот, который дает вам наименьшие значения AIC, BIC или BICc (см. Вики: http://en.wikipedia.org/wiki/Akaike_information_criterion, в основном можно рассматривать как логарифмическую вероятность, скорректированную на количество параметров, как распределение с большим количеством параметров. Ожидается, что параметры будут соответствовать лучше)
3, тот, который максимизирует байесовскую апостериорную вероятность. (см. вики: http://en.wikipedia.org/wiki/Posterior_probability)
Конечно, если у вас уже есть дистрибутив, который должен описывать ваши данные (основанные на теориях в вашей конкретной области) и вы хотите придерживаться этого, вы пропустите этап определения наиболее подходящего распределения.
scipy не поставляется с функцией для вычисления правдоподобия журнала (хотя предусмотрен метод MLE), но жесткий код один прост: см. Медленнее ли встроенные функции плотности вероятности `scipy.stat.distributions`, чем предоставленные пользователем?
AFAICU, ваше распределение дискретно (и ничего, кроме дискретного). Поэтому достаточно просто подсчитать частоты разных значений и нормализовать их. Итак, пример, чтобы продемонстрировать это:
In []: values= [0, 0, 0, 0, 0, 1, 1, 1, 1, 2, 2, 2, 3, 3, 4]
In []: counts= asarray(bincount(values), dtype= float)
In []: cdf= counts.cumsum()/ counts.sum()
Таким образом, вероятность увидеть значения выше, чем 1 это просто (согласно дополнительной кумулятивной функции распределения (ccdf):
In []: 1- cdf[1]
Out[]: 0.40000000000000002
Обратите внимание, что ccdf тесно связан с функцией выживания (sf), но он также определяется с помощью дискретных распределений, тогда как sf определяется только для смежных распределений.
Следующий код является версией общего ответа, но с исправлениями и ясностью.
import numpy as np
import pandas as pd
import scipy.stats as st
import statsmodels.api as sm
import matplotlib as mpl
import matplotlib.pyplot as plt
import math
import random
mpl.style.use("ggplot")
def danoes_formula(data):
"""
DANOE'S FORMULA
https://en.wikipedia.org/wiki/Histogram#Doane's_formula
"""
N = len(data)
skewness = st.skew(data)
sigma_g1 = math.sqrt((6*(N-2))/((N+1)*(N+3)))
num_bins = 1 + math.log(N,2) + math.log(1+abs(skewness)/sigma_g1,2)
num_bins = round(num_bins)
return num_bins
def plot_histogram(data, results, n):
## n first distribution of the ranking
N_DISTRIBUTIONS = {k: results[k] for k in list(results)[:n]}
## Histogram of data
plt.figure(figsize=(10, 5))
plt.hist(data, density=True, ec='white', color=(63/235, 149/235, 170/235))
plt.title('HISTOGRAM')
plt.xlabel('Values')
plt.ylabel('Frequencies')
## Plot n distributions
for i, distribution in N_DISTRIBUTIONS:
sse = results[distribution][0]
arg = results[distribution][1]
loc = results[distribution][2]
scale = results[distribution][3]
x_plot = np.linspace(min(data), max(data), 1000)
y_plot = distribution.pdf(x_plot, loc=loc, scale=scale, *arg)
plt.plot(x_plot, y_plot, label=str(distribution)[32:-34] + ": " + str(sse)[0:6], color=(random.uniform(0, 1), random.uniform(0, 1), random.uniform(0, 1)))
plt.legend(title='DISTRIBUTIONS', bbox_to_anchor=(1.05, 1), loc='upper left')
plt.show()
def fit_data(data):
## st.frechet_r,st.frechet_l: are disbled in current SciPy version
## st.levy_stable: a lot of time of estimation parameters
DISTRIBUTIONS = [
st.alpha,st.anglit,st.arcsine,st.beta,st.betaprime,st.bradford,st.burr,st.cauchy,st.chi,st.chi2,st.cosine,
st.dgamma,st.dweibull,st.erlang,st.expon,st.exponnorm,st.exponweib,st.exponpow,st.f,st.fatiguelife,st.fisk,
st.foldcauchy,st.foldnorm, st.genlogistic,st.genpareto,st.gennorm,st.genexpon,
st.genextreme,st.gausshyper,st.gamma,st.gengamma,st.genhalflogistic,st.gilbrat,st.gompertz,st.gumbel_r,
st.gumbel_l,st.halfcauchy,st.halflogistic,st.halfnorm,st.halfgennorm,st.hypsecant,st.invgamma,st.invgauss,
st.invweibull,st.johnsonsb,st.johnsonsu,st.ksone,st.kstwobign,st.laplace,st.levy,st.levy_l,
st.logistic,st.loggamma,st.loglaplace,st.lognorm,st.lomax,st.maxwell,st.mielke,st.nakagami,st.ncx2,st.ncf,
st.nct,st.norm,st.pareto,st.pearson3,st.powerlaw,st.powerlognorm,st.powernorm,st.rdist,st.reciprocal,
st.rayleigh,st.rice,st.recipinvgauss,st.semicircular,st.t,st.triang,st.truncexpon,st.truncnorm,st.tukeylambda,
st.uniform,st.vonmises,st.vonmises_line,st.wald,st.weibull_min,st.weibull_max,st.wrapcauchy
]
## Calculae Histogram
num_bins = danoes_formula(data)
frequencies, bin_edges = np.histogram(data, num_bins, density=True)
central_values = [(bin_edges[i] + bin_edges[i+1])/2 for i in range(len(bin_edges)-1)]
results = {}
for distribution in DISTRIBUTIONS:
## Get parameters of distribution
params = distribution.fit(data)
## Separate parts of parameters
arg = params[:-2]
loc = params[-2]
scale = params[-1]
## Calculate fitted PDF and error with fit in distribution
pdf_values = [distribution.pdf(c, loc=loc, scale=scale, *arg) for c in central_values]
## Calculate SSE (sum of squared estimate of errors)
sse = np.sum(np.power(frequencies - pdf_values, 2.0))
## Build results and sort by sse
results[distribution] = [sse, arg, loc, scale]
results = {k: results[k] for k in sorted(results, key=results.get)}
return results
def main():
## Import data
data = pd.Series(sm.datasets.elnino.load_pandas().data.set_index('YEAR').values.ravel())
results = fit_data(data)
plot_histogram(data, results, 10)
if __name__ == "__main__":
main()
Хотя многие из приведенных выше ответов полностью верны, никто, похоже, не ответит на ваш вопрос полностью, особенно в части:
Я не знаю, прав ли я, но для определения вероятностей, я думаю, мне нужно подогнать мои данные к теоретическому распределению, которое наиболее подходит для описания моих данных. Я предполагаю, что для определения лучшей модели необходим какой-то тест согласия.
Параметрический подход
Это описываемый вами процесс использования некоторого теоретического распределения и подгонки параметров к вашим данным, и есть отличные ответы, как это сделать.
Непараметрический подход
Однако также можно использовать непараметрический подход к вашей проблеме, что означает, что вы вообще не предполагаете никакого базового распределения.
Используя так называемую эмпирическую функцию распределения, которая равна:Fn(x)= SUM(I [X<= x]) / n. Итак, пропорция значений ниже x.
Как было указано в одном из приведенных выше ответов, вас интересует обратный CDF (кумулятивная функция распределения), который равен 1-F(x)
Можно показать, что эмпирическая функция распределения сходится к любой "истинной" CDF, которая сгенерировала ваши данные.
Кроме того, легко построить доверительный интервал 1 альфа:
L(X) = max{Fn(x)-en, 0}
U(X) = min{Fn(x)+en, 0}
en = sqrt( (1/2n)*log(2/alpha)
Тогда P( L(X) <= F(X) <= U(X)) >= 1-альфа для всех x.
Я очень удивлен, что ответ PierrOz получил 0 голосов, в то время как это полностью действительный ответ на вопрос с использованием непараметрического подхода к оценке F(x).
Обратите внимание, что проблема, которую вы упоминаете о P(X>=x)=0 для любого x>47, является просто личным предпочтением, которое может привести вас к выбору параметрического подхода над непараметрическим подходом. Однако оба подхода - вполне допустимые решения вашей проблемы.
Для получения более подробной информации и доказательств приведенных выше утверждений я бы рекомендовал взглянуть на "Вся статистика: Краткий курс статистических выводов Ларри А. Вассермана". Отличная книга как по параметрическому, так и по непараметрическому выводу.
РЕДАКТИРОВАТЬ: Поскольку вы специально попросили несколько примеров Python, это можно сделать с помощью numpy:
import numpy as np
def empirical_cdf(data, x):
return np.sum(x<=data)/len(data)
def p_value(data, x):
return 1-empirical_cdf(data, x)
# Generate some data for demonstration purposes
data = np.floor(np.random.uniform(low=0, high=48, size=30000))
print(empirical_cdf(data, 20))
print(p_value(data, 20)) # This is the value you're interested in
Самый простой способ, который я нашел, - использовать модуль установки, и вы можете просто
pip install fitter. Все, что вам нужно сделать, это импортировать набор данных с помощью pandas. Он имеет встроенную функцию для поиска всех 80 дистрибутивов из scipy и получения наилучшего соответствия данным различными методами. Пример:
f = Fitter(height, distributions=['gamma','lognorm', "beta","burr","norm"])
f.fit()
f.summary()
Здесь автор предоставил список дистрибутивов, поскольку сканирование всех 80 может занять много времени.
f.get_best(method = 'sumsquare_error')
Это даст вам 5 лучших дистрибутивов с их критериями соответствия:
sumsquare_error aic bic kl_div
chi2 0.000010 1716.234916 -1945.821606 inf
gamma 0.000010 1716.234909 -1945.821606 inf
rayleigh 0.000010 1711.807360 -1945.526026 inf
norm 0.000011 1758.797036 -1934.865211 inf
cauchy 0.000011 1762.735606 -1934.803414 inf
У вас также есть
distributions=get_common_distributions()атрибут, который имеет около 10 наиболее часто используемых дистрибутивов, подбирает и проверяет их для вас.
Он также имеет множество других функций, таких как построение гистограмм, и всю полную документацию можно найти здесь.
Это серьезно недооцененный модуль для ученых, инженеров и вообще.
Извините, если я не понимаю вашу потребность, но как насчет хранения ваших данных в словаре, где ключами будут числа от 0 до 47 и значения количества вхождений их связанных ключей в вашем исходном списке?
Таким образом, ваша вероятность p(x) будет суммой всех значений для ключей больше x, деленной на 30000.
Это звучит как проблема оценки плотности вероятности для меня.
from scipy.stats import gaussian_kde
occurences = [0,0,0,0,..,1,1,1,1,...,2,2,2,2,...,47]
values = range(0,48)
kde = gaussian_kde(map(float, occurences))
p = kde(values)
p = p/sum(p)
print "P(x>=1) = %f" % sum(p[1:])
Также см. http://jpktd.blogspot.com/2009/03/using-gaussian-kernel-density.html.
С OpenTURNS я бы использовал критерии BIC, чтобы выбрать лучшее распределение, которое соответствует таким данным. Это связано с тем, что этот критерий не дает слишком большого преимущества распределениям с большим количеством параметров. В самом деле, если распределение имеет больше параметров, подобранному распределению легче быть ближе к данным. Более того, в этом случае метод Колмогорова-Смирнова может не иметь смысла, поскольку небольшая ошибка в измеренных значениях будет иметь огромное влияние на значение p.
Чтобы проиллюстрировать процесс, я загружаю данные Эль-Ниньо, которые содержат 732 ежемесячных измерения температуры с 1950 по 2010 год:
import statsmodels.api as sm
dta = sm.datasets.elnino.load_pandas().data
dta['YEAR'] = dta.YEAR.astype(int).astype(str)
dta = dta.set_index('YEAR').T.unstack()
data = dta.values
Легко получить 30 встроенных одномерных фабрик распределений с GetContinuousUniVariateFactoriesстатический метод. После этогоBestModelBIC статический метод возвращает лучшую модель и соответствующий балл BIC.
sample = ot.Sample(data, 1)
tested_factories = ot.DistributionFactory.GetContinuousUniVariateFactories()
best_model, best_bic = ot.FittingTest.BestModelBIC(sample,
tested_factories)
print("Best=",best_model)
который печатает:
Best= Beta(alpha = 1.64258, beta = 2.4348, a = 18.936, b = 29.254)
Чтобы графически сравнить соответствие с гистограммой, я использую drawPDF методы наилучшего распространения.
import openturns.viewer as otv
graph = ot.HistogramFactory().build(sample).drawPDF()
bestPDF = best_model.drawPDF()
bestPDF.setColors(["blue"])
graph.add(bestPDF)
graph.setTitle("Best BIC fit")
name = best_model.getImplementation().getClassName()
graph.setLegends(["Histogram",name])
graph.setXTitle("Temperature (°C)")
otv.View(graph)
Это производит:
Более подробная информация по этой теме представлена в документе BestModelBIC. Можно было бы включить дистрибутив Scipy в SciPyDistribution или даже в дистрибутивы ChaosPy с ChaosPyDistribution, но я предполагаю, что текущий скрипт выполняет большинство практических задач.
Я перерабатываю функцию распределения из первого ответа, где я включил параметр выбора для выбора одного из тестов на соответствие, который сужает функцию распределения, которая соответствует данным:
import numpy as np
import pandas as pd
import scipy.stats as st
import matplotlib.pyplot as plt
import pylab
def make_hist(data):
#### General code:
bins_formulas = ['auto', 'fd', 'scott', 'rice', 'sturges', 'doane', 'sqrt']
bins = np.histogram_bin_edges(a=data, bins='fd', range=(min(data), max(data)))
# print('Bin value = ', bins)
# Obtaining the histogram of data:
Hist, bin_edges = histogram(a=data, bins=bins, range=(min(data), max(data)), density=True)
bin_mid = (bin_edges + np.roll(bin_edges, -1))[:-1] / 2.0 # go from bin edges to bin middles
return Hist, bin_mid
def make_pdf(dist, params, size):
"""Generate distributions's Probability Distribution Function """
# Separate parts of parameters
arg = params[:-2]
loc = params[-2]
scale = params[-1]
# Get sane start and end points of distribution
start = dist.ppf(0.01, *arg, loc=loc, scale=scale) if arg else dist.ppf(0.01, loc=loc, scale=scale)
end = dist.ppf(0.99, *arg, loc=loc, scale=scale) if arg else dist.ppf(0.99, loc=loc, scale=scale)
# Build PDF and turn into pandas Series
x = np.linspace(start, end, size)
y = dist.pdf(x, loc=loc, scale=scale, *arg)
pdf = pd.Series(y, x)
return pdf, x, y
def compute_r2_test(y_true, y_predicted):
sse = sum((y_true - y_predicted)**2)
tse = (len(y_true) - 1) * np.var(y_true, ddof=1)
r2_score = 1 - (sse / tse)
return r2_score, sse, tse
def get_best_distribution_2(data, method, plot=False):
dist_names = ['alpha', 'anglit', 'arcsine', 'beta', 'betaprime', 'bradford', 'burr', 'cauchy', 'chi', 'chi2', 'cosine', 'dgamma', 'dweibull', 'erlang', 'expon', 'exponweib', 'exponpow', 'f', 'fatiguelife', 'fisk', 'foldcauchy', 'foldnorm', 'frechet_r', 'frechet_l', 'genlogistic', 'genpareto', 'genexpon', 'genextreme', 'gausshyper', 'gamma', 'gengamma', 'genhalflogistic', 'gilbrat', 'gompertz', 'gumbel_r', 'gumbel_l', 'halfcauchy', 'halflogistic', 'halfnorm', 'hypsecant', 'invgamma', 'invgauss', 'invweibull', 'johnsonsb', 'johnsonsu', 'ksone', 'kstwobign', 'laplace', 'logistic', 'loggamma', 'loglaplace', 'lognorm', 'lomax', 'maxwell', 'mielke', 'moyal', 'nakagami', 'ncx2', 'ncf', 'nct', 'norm', 'pareto', 'pearson3', 'powerlaw', 'powerlognorm', 'powernorm', 'rdist', 'reciprocal', 'rayleigh', 'rice', 'recipinvgauss', 'semicircular', 't', 'triang', 'truncexpon', 'truncnorm', 'tukeylambda', 'uniform', 'vonmises', 'wald', 'weibull_min', 'weibull_max', 'wrapcauchy']
# Applying the Goodness-to-fit tests to select the best distribution that fits the data:
dist_results = []
dist_IC_results = []
params = {}
params_IC = {}
params_SSE = {}
if method == 'sse':
########################################################################################################################
######################################## Sum of Square Error (SSE) test ################################################
########################################################################################################################
# Best holders
best_distribution = st.norm
best_params = (0.0, 1.0)
best_sse = np.inf
for dist_name in dist_names:
dist = getattr(st, dist_name)
param = dist.fit(data)
params[dist_name] = param
N_len = len(list(data))
# Obtaining the histogram:
Hist_data, bin_data = make_hist(data=data)
# fit dist to data
params_dist = dist.fit(data)
# Separate parts of parameters
arg = params_dist[:-2]
loc = params_dist[-2]
scale = params_dist[-1]
# Calculate fitted PDF and error with fit in distribution
pdf = dist.pdf(bin_data, loc=loc, scale=scale, *arg)
sse = np.sum(np.power(Hist_data - pdf, 2.0))
# identify if this distribution is better
if best_sse > sse > 0:
best_distribution = dist
best_params = params_dist
best_stat_test_val = sse
print('\n################################ Sum of Square Error test parameters #####################################')
best_dist = best_distribution
print("Best fitting distribution (SSE test) :" + str(best_dist))
print("Best SSE value (SSE test) :" + str(best_stat_test_val))
print("Parameters for the best fit (SSE test) :" + str(params[best_dist]))
print('###########################################################################################################\n')
########################################################################################################################
########################################################################################################################
########################################################################################################################
if method == 'r2':
########################################################################################################################
##################################################### R Square (R^2) test ##############################################
########################################################################################################################
# Best holders
best_distribution = st.norm
best_params = (0.0, 1.0)
best_r2 = np.inf
for dist_name in dist_names:
dist = getattr(st, dist_name)
param = dist.fit(data)
params[dist_name] = param
N_len = len(list(data))
# Obtaining the histogram:
Hist_data, bin_data = make_hist(data=data)
# fit dist to data
params_dist = dist.fit(data)
# Separate parts of parameters
arg = params_dist[:-2]
loc = params_dist[-2]
scale = params_dist[-1]
# Calculate fitted PDF and error with fit in distribution
pdf = dist.pdf(bin_data, loc=loc, scale=scale, *arg)
r2 = compute_r2_test(y_true=Hist_data, y_predicted=pdf)
# identify if this distribution is better
if best_r2 > r2 > 0:
best_distribution = dist
best_params = params_dist
best_stat_test_val = r2
print('\n############################## R Square test parameters ###########################################')
best_dist = best_distribution
print("Best fitting distribution (R^2 test) :" + str(best_dist))
print("Best R^2 value (R^2 test) :" + str(best_stat_test_val))
print("Parameters for the best fit (R^2 test) :" + str(params[best_dist]))
print('#####################################################################################################\n')
########################################################################################################################
########################################################################################################################
########################################################################################################################
if method == 'ic':
########################################################################################################################
######################################## Information Criteria (IC) test ################################################
########################################################################################################################
for dist_name in dist_names:
dist = getattr(st, dist_name)
param = dist.fit(data)
params[dist_name] = param
N_len = len(list(data))
# Obtaining the histogram:
Hist_data, bin_data = make_hist(data=data)
# fit dist to data
params_dist = dist.fit(data)
# Separate parts of parameters
arg = params_dist[:-2]
loc = params_dist[-2]
scale = params_dist[-1]
# Calculate fitted PDF and error with fit in distribution
pdf = dist.pdf(bin_data, loc=loc, scale=scale, *arg)
sse = np.sum(np.power(Hist_data - pdf, 2.0))
# Obtaining the log of the pdf:
loglik = np.sum(dist.logpdf(bin_data, *params_dist))
k = len(params_dist[:])
n = len(data)
aic = 2 * k - 2 * loglik
bic = n * np.log(sse / n) + k * np.log(n)
dist_IC_results.append((dist_name, aic))
# dist_IC_results.append((dist_name, bic))
# select the best fitted distribution and store the name of the best fit and its IC value
best_dist, best_ic = (min(dist_IC_results, key=lambda item: item[1]))
print('\n############################ Information Criteria (IC) test parameters ##################################')
print("Best fitting distribution (IC test) :" + str(best_dist))
print("Best IC value (IC test) :" + str(best_ic))
print("Parameters for the best fit (IC test) :" + str(params[best_dist]))
print('###########################################################################################################\n')
########################################################################################################################
########################################################################################################################
########################################################################################################################
if method == 'chi':
########################################################################################################################
################################################ Chi-Square (Chi^2) test ###############################################
########################################################################################################################
# Set up 50 bins for chi-square test
# Observed data will be approximately evenly distrubuted aross all bins
percentile_bins = np.linspace(0,100,51)
percentile_cutoffs = np.percentile(data, percentile_bins)
observed_frequency, bins = (np.histogram(data, bins=percentile_cutoffs))
cum_observed_frequency = np.cumsum(observed_frequency)
chi_square = []
for dist_name in dist_names:
dist = getattr(st, dist_name)
param = dist.fit(data)
params[dist_name] = param
# Obtaining the histogram:
Hist_data, bin_data = make_hist(data=data)
# fit dist to data
params_dist = dist.fit(data)
# Separate parts of parameters
arg = params_dist[:-2]
loc = params_dist[-2]
scale = params_dist[-1]
# Calculate fitted PDF and error with fit in distribution
pdf = dist.pdf(bin_data, loc=loc, scale=scale, *arg)
# Get expected counts in percentile bins
# This is based on a 'cumulative distrubution function' (cdf)
cdf_fitted = dist.cdf(percentile_cutoffs, *arg, loc=loc, scale=scale)
expected_frequency = []
for bin in range(len(percentile_bins) - 1):
expected_cdf_area = cdf_fitted[bin + 1] - cdf_fitted[bin]
expected_frequency.append(expected_cdf_area)
# calculate chi-squared
expected_frequency = np.array(expected_frequency) * size
cum_expected_frequency = np.cumsum(expected_frequency)
ss = sum(((cum_expected_frequency - cum_observed_frequency) ** 2) / cum_observed_frequency)
chi_square.append(ss)
# Applying the Chi-Square test:
# D, p = scipy.stats.chisquare(f_obs=pdf, f_exp=Hist_data)
# print("Chi-Square test Statistics value for " + dist_name + " = " + str(D))
print("p value for " + dist_name + " = " + str(chi_square))
dist_results.append((dist_name, chi_square))
# select the best fitted distribution and store the name of the best fit and its p value
best_dist, best_stat_test_val = (min(dist_results, key=lambda item: item[1]))
print('\n#################################### Chi-Square test parameters #######################################')
print("Best fitting distribution (Chi^2 test) :" + str(best_dist))
print("Best p value (Chi^2 test) :" + str(best_stat_test_val))
print("Parameters for the best fit (Chi^2 test) :" + str(params[best_dist]))
print('#########################################################################################################\n')
########################################################################################################################
########################################################################################################################
########################################################################################################################
if method == 'ks':
########################################################################################################################
########################################## Kolmogorov-Smirnov (KS) test ################################################
########################################################################################################################
for dist_name in dist_names:
dist = getattr(st, dist_name)
param = dist.fit(data)
params[dist_name] = param
# Applying the Kolmogorov-Smirnov test:
D, p = st.kstest(data, dist_name, args=param)
# D, p = st.kstest(data, dist_name, args=param, N=N_len, alternative='greater')
# print("Kolmogorov-Smirnov test Statistics value for " + dist_name + " = " + str(D))
print("p value for " + dist_name + " = " + str(p))
dist_results.append((dist_name, p))
# select the best fitted distribution and store the name of the best fit and its p value
best_dist, best_stat_test_val = (max(dist_results, key=lambda item: item[1]))
print('\n################################ Kolmogorov-Smirnov test parameters #####################################')
print("Best fitting distribution (KS test) :" + str(best_dist))
print("Best p value (KS test) :" + str(best_stat_test_val))
print("Parameters for the best fit (KS test) :" + str(params[best_dist]))
print('###########################################################################################################\n')
########################################################################################################################
########################################################################################################################
########################################################################################################################
# Collate results and sort by goodness of fit (best at top)
results = pd.DataFrame()
results['Distribution'] = dist_names
results['chi_square'] = chi_square
# results['p_value'] = p_values
results.sort_values(['chi_square'], inplace=True)
# Plotting the distribution with histogram:
if plot:
bins_val = np.histogram_bin_edges(a=data, bins='fd', range=(min(data), max(data)))
plt.hist(x=data, bins=bins_val, range=(min(data), max(data)), density=True)
# pylab.hist(x=data, bins=bins_val, range=(min(data), max(data)))
best_param = params[best_dist]
best_dist_p = getattr(st, best_dist)
pdf, x_axis_pdf, y_axis_pdf = make_pdf(dist=best_dist_p, params=best_param, size=len(data))
plt.plot(x_axis_pdf, y_axis_pdf, color='red', label='Best dist ={0}'.format(best_dist))
plt.legend()
plt.title('Histogram and Distribution plot of data')
# plt.show()
plt.show(block=False)
plt.pause(5) # Pauses the program for 5 seconds
plt.close('all')
return best_dist, best_stat_test_val, params[best_dist]
затем продолжите функцию make_pdf, чтобы получить выбранный дистрибутив на основе вашего критерия согласия.
Основываясь на ответе Тимоти Давенпорта , я реорганизовал код, чтобы его можно было использовать в качестве библиотеки, и сделал его доступным в виде проекта github и pypi, см.:
- https://github.com/WolfgangFahl/pyProbabilityDistributionFit
- https://github.com/WolfgangFahl/pyProbabilityDistributionFit/blob/main/pdffit/distfit.py
Одна из целей — сделать доступной опцию плотности и вывести результат в виде файлов. См. основную часть реализации:
bfd=BestFitDistribution(data)
for density in [True,False]:
suffix="density" if density else ""
bfd.analyze(title=u'El Niño sea temp.',x_label=u'Temp (°C)',y_label='Frequency',outputFilePrefix=f"/tmp/ElNinoPDF{suffix}",density=density)
# uncomment for interactive display
# plt.show()
Также есть модульные тесты для библиотеки, см., например , тест нормального распределения .
def testNormal(self):
'''
test the normal distribution
'''
# use euler constant as seed
np.random.seed(0)
# statistically relevant number of datapoints
datapoints=1000
a = np.random.normal(40, 10, datapoints)
df= pd.DataFrame({'nums':a})
outputFilePrefix="/tmp/normalDist"
bfd=BestFitDistribution(df,debug=True)
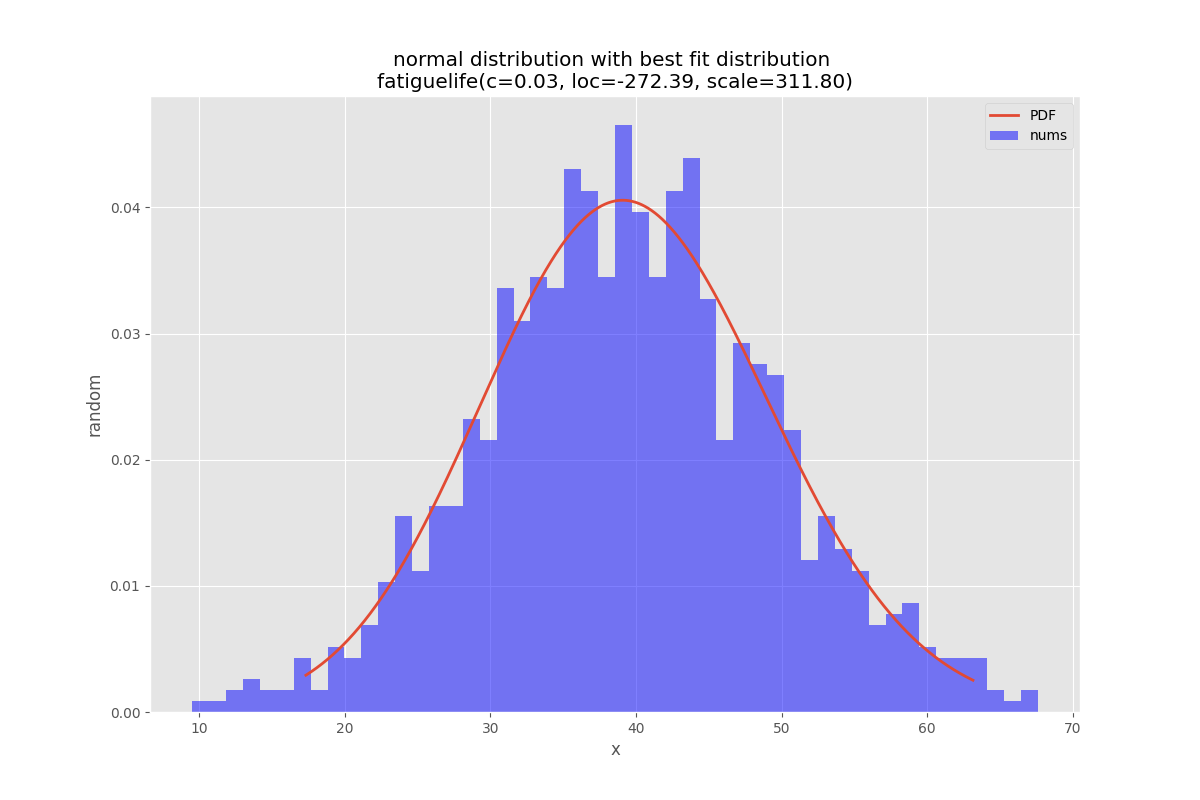
bfd.analyze(title="normal distribution",x_label="x",y_label="random",outputFilePrefix=outputFilePrefix)
 Пожалуйста , добавляйте вопросы к проекту , если вы видите проблемы или возможности для улучшения. Обсуждения также включены.
Пожалуйста , добавляйте вопросы к проекту , если вы видите проблемы или возможности для улучшения. Обсуждения также включены.
приведенный ниже код может быть устаревшим, используйте pypi или репозиторий github для получения самой последней версии.
'''
Created on 2022-05-17
see
https://stackoverflow.com/questions/6620471/fitting-empirical-distribution-to-theoretical-ones-with-scipy-python/37616966#37616966
@author: https://stackoverflow.com/users/832621/saullo-g-p-castro
see https://stackoverflow.com/a/37616966/1497139
@author: https://stackoverflow.com/users/2087463/tmthydvnprt
see https://stackoverflow.com/a/37616966/1497139
@author: https://stackoverflow.com/users/1497139/wolfgang-fahl
see
'''
import traceback
import sys
import warnings
import numpy as np
import pandas as pd
import scipy.stats as st
from scipy.stats._continuous_distns import _distn_names
import statsmodels.api as sm
import matplotlib
import matplotlib.pyplot as plt
class BestFitDistribution():
'''
Find the best Probability Distribution Function for the given data
'''
def __init__(self,data,distributionNames:list=None,debug:bool=False):
'''
constructor
Args:
data(dataFrame): the data to analyze
distributionNames(list): list of distributionNames to try
debug(bool): if True show debugging information
'''
self.debug=debug
self.matplotLibParams()
if distributionNames is None:
self.distributionNames=[d for d in _distn_names if not d in ['levy_stable', 'studentized_range']]
else:
self.distributionNames=distributionNames
self.data=data
def matplotLibParams(self):
'''
set matplotlib parameters
'''
matplotlib.rcParams['figure.figsize'] = (16.0, 12.0)
#matplotlib.style.use('ggplot')
matplotlib.use("WebAgg")
# Create models from data
def best_fit_distribution(self,bins:int=200, ax=None,density:bool=True):
"""
Model data by finding best fit distribution to data
"""
# Get histogram of original data
y, x = np.histogram(self.data, bins=bins, density=density)
x = (x + np.roll(x, -1))[:-1] / 2.0
# Best holders
best_distributions = []
distributionCount=len(self.distributionNames)
# Estimate distribution parameters from data
for ii, distributionName in enumerate(self.distributionNames):
print(f"{ii+1:>3} / {distributionCount:<3}: {distributionName}")
distribution = getattr(st, distributionName)
# Try to fit the distribution
try:
# Ignore warnings from data that can't be fit
with warnings.catch_warnings():
warnings.filterwarnings('ignore')
# fit dist to data
params = distribution.fit(self.data)
# Separate parts of parameters
arg = params[:-2]
loc = params[-2]
scale = params[-1]
# Calculate fitted PDF and error with fit in distribution
pdf = distribution.pdf(x, loc=loc, scale=scale, *arg)
sse = np.sum(np.power(y - pdf, 2.0))
# if axis pass in add to plot
try:
if ax:
pd.Series(pdf, x).plot(ax=ax)
except Exception:
pass
# identify if this distribution is better
best_distributions.append((distribution, params, sse))
except Exception as ex:
if self.debug:
trace=traceback.format_exc()
msg=f"fit for {distributionName} failed:{ex}\n{trace}"
print(msg,file=sys.stderr)
pass
return sorted(best_distributions, key=lambda x:x[2])
def make_pdf(self,dist, params:list, size=10000):
"""
Generate distributions's Probability Distribution Function
Args:
dist: Distribution
params(list): parameter
size(int): size
Returns:
dataframe: Power Distribution Function
"""
# Separate parts of parameters
arg = params[:-2]
loc = params[-2]
scale = params[-1]
# Get sane start and end points of distribution
start = dist.ppf(0.01, *arg, loc=loc, scale=scale) if arg else dist.ppf(0.01, loc=loc, scale=scale)
end = dist.ppf(0.99, *arg, loc=loc, scale=scale) if arg else dist.ppf(0.99, loc=loc, scale=scale)
# Build PDF and turn into pandas Series
x = np.linspace(start, end, size)
y = dist.pdf(x, loc=loc, scale=scale, *arg)
pdf = pd.Series(y, x)
return pdf
def analyze(self,title,x_label,y_label,outputFilePrefix=None,imageFormat:str='png',allBins:int=50,distBins:int=200,density:bool=True):
"""
analyze the Probabilty Distribution Function
Args:
data: Panda Dataframe or numpy array
title(str): the title to use
x_label(str): the label for the x-axis
y_label(str): the label for the y-axis
outputFilePrefix(str): the prefix of the outputFile
imageFormat(str): imageFormat e.g. png,svg
allBins(int): the number of bins for all
distBins(int): the number of bins for the distribution
density(bool): if True show relative density
"""
self.allBins=allBins
self.distBins=distBins
self.density=density
self.title=title
self.x_label=x_label
self.y_label=y_label
self.imageFormat=imageFormat
self.outputFilePrefix=outputFilePrefix
self.color=list(matplotlib.rcParams['axes.prop_cycle'])[1]['color']
self.best_dist=None
self.analyzeAll()
if outputFilePrefix is not None:
self.saveFig(f"{outputFilePrefix}All.{imageFormat}", imageFormat)
plt.close(self.figAll)
if self.best_dist:
self.analyzeBest()
if outputFilePrefix is not None:
self.saveFig(f"{outputFilePrefix}Best.{imageFormat}", imageFormat)
plt.close(self.figBest)
def analyzeAll(self):
'''
analyze the given data
'''
# Plot for comparison
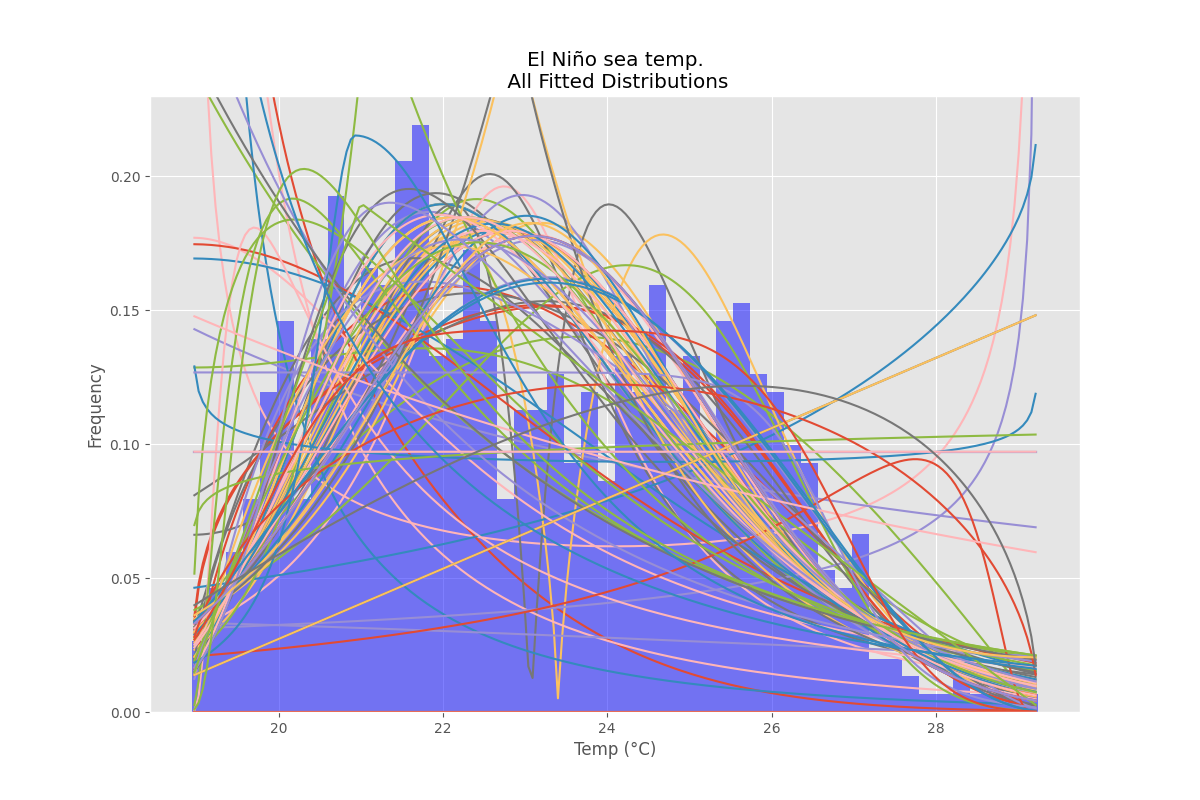
figTitle=f"{self.title}\n All Fitted Distributions"
self.figAll=plt.figure(figTitle,figsize=(12,8))
ax = self.data.plot(kind='hist', bins=self.allBins, density=self.density, alpha=0.5, color=self.color)
# Save plot limits
dataYLim = ax.get_ylim()
# Update plots
ax.set_ylim(dataYLim)
ax.set_title(figTitle)
ax.set_xlabel(self.x_label)
ax.set_ylabel(self.y_label)
# Find best fit distribution
best_distributions = self.best_fit_distribution(bins=self.distBins, ax=ax,density=self.density)
if len(best_distributions)>0:
self.best_dist = best_distributions[0]
# Make PDF with best params
self.pdf = self.make_pdf(self.best_dist[0], self.best_dist[1])
def analyzeBest(self):
'''
analyze the Best Property Distribution function
'''
# Display
figLabel="PDF"
self.figBest=plt.figure(figLabel,figsize=(12,8))
ax = self.pdf.plot(lw=2, label=figLabel, legend=True)
self.data.plot(kind='hist', bins=self.allBins, density=self.density, alpha=0.5, label='Data', legend=True, ax=ax,color=self.color)
param_names = (self.best_dist[0].shapes + ', loc, scale').split(', ') if self.best_dist[0].shapes else ['loc', 'scale']
param_str = ', '.join(['{}={:0.2f}'.format(k,v) for k,v in zip(param_names, self.best_dist[1])])
dist_str = '{}({})'.format(self.best_dist[0].name, param_str)
ax.set_title(f'{self.title} with best fit distribution \n' + dist_str)
ax.set_xlabel(self.x_label)
ax.set_ylabel(self.y_label)
def saveFig(self,outputFile:str=None,imageFormat='png'):
'''
save the current Figure to the given outputFile
Args:
outputFile(str): the outputFile to save to
imageFormat(str): the imageFormat to use e.g. png/svg
'''
plt.savefig(outputFile, format=imageFormat) # dpi
if __name__ == '__main__':
# Load data from statsmodels datasets
data = pd.Series(sm.datasets.elnino.load_pandas().data.set_index('YEAR').values.ravel())
bfd=BestFitDistribution(data)
for density in [True,False]:
suffix="density" if density else ""
bfd.analyze(title=u'El Niño sea temp.',x_label=u'Temp (°C)',y_label='Frequency',outputFilePrefix=f"/tmp/ElNinoPDF{suffix}",density=density)
# uncomment for interactive display
# plt.show()