Применить вращение к карте HEALPix в healpy
У меня есть карта HEALPix, которую я прочитал, используя healpy, однако она находится в галактических координатах, и мне она нужна в небесных / экваториальных координатах. Кто-нибудь знает простой способ конвертировать карту?
Я пытался использовать healpy.Rotator преобразовать из (l,b) в (фи, тета), а затем с помощью healpy.ang2pix изменить порядок пикселей, но карта все еще выглядит странно.
Было бы здорово, если бы была функция, аналогичная Rotator что вы могли бы назвать как: map = AnotherRotator(map,coord=['G','C']), Кто-нибудь знает о любой такой функции?
Спасибо,
Alex
3 ответа
Я нашел другое потенциальное решение после нескольких месяцев поиска. Я еще не очень много тестировал, поэтому, пожалуйста, будьте осторожны!
Решение Саула 2, выше, является ключом (отличное предложение!)
По сути, вы объединяете функциональность healpy.mollview (gnomview, cartview, а также orthview работать также) с этим из reproject_to_healpix функция в reproject пакет ( http://reproject.readthedocs.org/en/stable/).
Полученная карта подходит для моих угловых масштабов, но я не могу сказать, насколько точна трансформация по сравнению с другими методами.
----- Основные положения ----------
Шаг 1: Считайте карту и создайте прямоугольный массив с помощью cartview, Как Саул указал выше, это также один из способов сделать вращение. Если вы просто делаете стандартное вращение / преобразование координат, тогда все, что вам нужно, это coord ключевое слово. От небесных до галактических координат установите coord = ['C','G']
map_Gal = hp.cartview(map_Cel, coord=['C','G'], return_projected_map=True, xsize=desired_xsize, norm='hist',nest=False)
Шаг 2: Напишите шаблон заголовка FITS для всего неба (как в примере ниже). Я написал свой, чтобы иметь тот же средний пиксельный масштаб, что и моя желаемая карта HEALPix.
Шаг 3: Используйте reproject.transform_to_healpix
reproject включает в себя функцию для отображения "нормального" массива (или файла FITS) в проекцию HEALPix. Добавьте к этому возможность возвращать массив, созданный healpy.mollview/cartview/orthview/gnomview, и вы можете повернуть карту HEALPix одной системы координат (Небесная) в другую систему координат (Галактическая).
map_Gal_HP, footprint_Gal_HP = rp.reproject_to_healpix((map_Gal, target_header), coord_system_out= 'GALACTIC', nside=nside, nested=False)
Это сводится, по сути, к этим двум командам. Однако вам нужно будет создать заголовок шаблона, указав масштаб и размер в пикселях, соответствующие промежуточной карте всего неба, которую вы хотите создать.
----- Полный рабочий пример (формат записной книжки iPython + пример данных FITS) ------
https://github.com/aaroncnb/healpix_coordtrans_example/tree/master
Код должен работать очень быстро, но это потому, что карты сильно деградировали. Я сделал то же самое для своих карт NSIDE 1024 и 2048, и это заняло около часа.
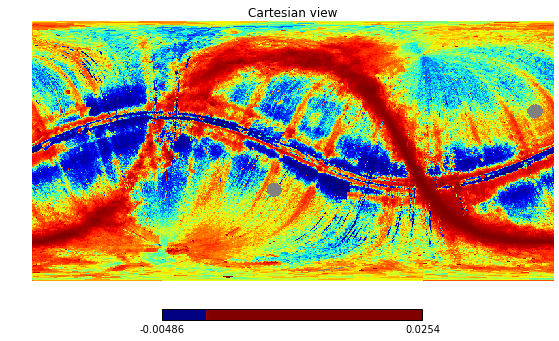
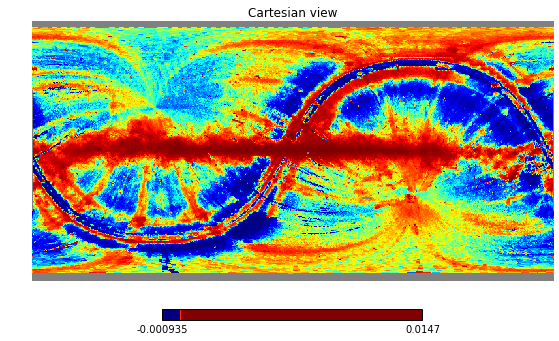
------ до и после изображения ------
Я понимаю, что об этом спрашивали много лет назад, но у меня была такая же проблема на этой неделе, и я нашел ваш пост. Я нашел пару потенциальных решений, поэтому я поделюсь, если кто-то еще придет к этому и сочтет это полезным.
Решение 1: Этот вид зависит от формата, в который поступают ваши данные. Мой поступил в виде сетки (тэта, фи).
import numpy as np
import healpy as H
map = <your original map>
nside = <your map resolution, mine=256>
npix = H.nside2npix(nside)
pix = N.arange(npix)
t,p = H.pix2ang(nside,pix) #theta, phi
r = H.Rotator(deg=True, rot=[<THETA ROTATION>, <PHI ROTATION>])
map_rot = np.zeros(npix)
for i in pix:
trot, prot = r(t[i],p[i])
tpix = int(trot*180./np.pi) #my data came in a theta, phi grid -- this finds its location there
ppix = int(prot*180./np.pi)
map_rot[i] = map[ppix,tpix] #this being the rright way round may need double-checking
Решение 2: еще не закончил тестировать это, а просто наткнулся на это ПОСЛЕ выполнения раздражающей работы выше...
map_rot = H.mollview(map,deg=True,rot=[<THETA>,<PHI>], return_projected_map=True)
который дает двумерный массив. Мне интересно знать, как преобразовать это обратно в карту healpix...
Эта функция, кажется, делает свое дело (довольно медленно, но должно быть лучше, чем цикл for):
def rotate_map(hmap, rot_theta, rot_phi):
"""
Take hmap (a healpix map array) and return another healpix map array
which is ordered such that it has been rotated in (theta, phi) by the
amounts given.
"""
nside = hp.npix2nside(len(hmap))
# Get theta, phi for non-rotated map
t,p = hp.pix2ang(nside, np.arange(hp.nside2npix(nside))) #theta, phi
# Define a rotator
r = hp.Rotator(deg=False, rot=[rot_phi,rot_theta])
# Get theta, phi under rotated co-ordinates
trot, prot = r(t,p)
# Interpolate map onto these co-ordinates
rot_map = hp.get_interp_val(hmap, trot, prot)
return rot_map
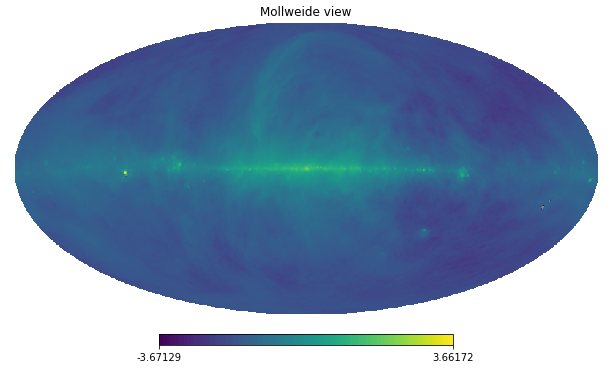
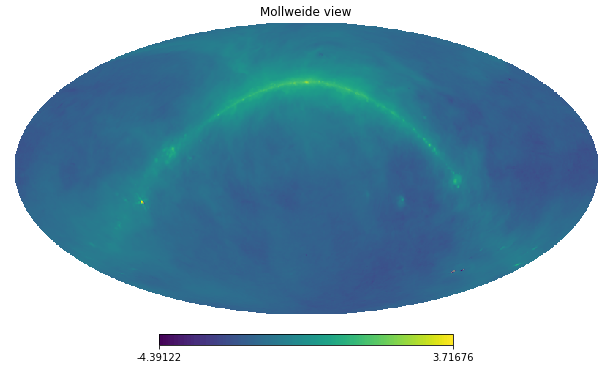
Используя это на данных из PyGSM дает следующее:
hp.mollview(np.log(rotate_map(gsm.generated_map_data, 0,0)))
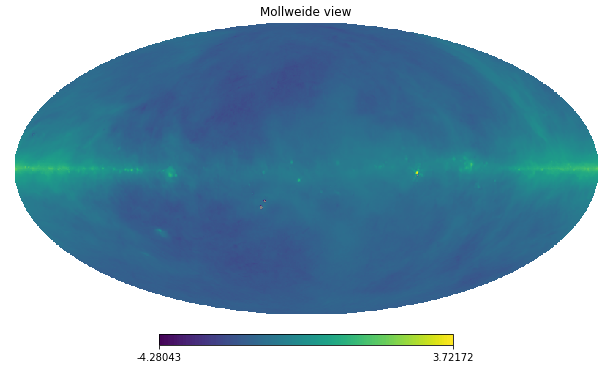
При вращении phi:
hp.mollview(np.log(rotate_map(gsm.generated_map_data, 0,np.pi)))
Или вращающийся theta:
hp.mollview(np.log(rotate_map(gsm.generated_map_data, np.pi/4,0)))