Несколько функций над списком столбцов и автоматически генерировать новые имена столбцов с помощью data.table
Как настроить манипулирование таблицей данных так, чтобы, помимо sum для категории из нескольких столбцов одновременно вычисляются и другие функции, такие как mean и считается (.N) и автоматически создавать имена столбцов: "сумма с1", "сумма с2", "сумма с4", "среднее значение с1", "среднее значение с2", "среднее значение с4" и предпочтительно также 1 столбец "рассчитывает"?
Моим старым решением было выписать
mean col1 = ....
mean col2 = ....
Etc, внутри команды data.table
Это сработало, но, по-моему, ужасно неэффективно, и больше не будет работать для его предварительного кодирования, если в новой версии приложения вычисления зависят от выбора пользователя в приложении R Shiny, что рассчитывать для каких столбцов.
Я пролистал кучу постов и статей в блогах, но не совсем понял, как лучше всего это сделать. Я читал, что в некоторых случаях манипуляции могут быть довольно медленными в больших таблицах данных в зависимости от того, какой подход вы используете (.sdcols, get, lapply и или by =). Поэтому я добавил "большой" набор фиктивных данных
Мои реальные данные - около 100 тыс. Строк на 100 столбцов и примерно 1-100 групп.
library(data.table)
n = 100000
dt = data.table(index=1:100000,
category = sample(letters[1:25], n, replace = T),
c1=rnorm(n,10000),
c2=rnorm(n,1000),
c3=rnorm(n,100),
c4 = rnorm(n,10)
)
# add more columns to test for big data tables
lapply(c(paste('c', 5:100, sep ='')),
function(addcol) dt[[addcol]] <<- rnorm(n,1000) )
# Simulate columns selected by shiny app user
Colchoice <- c("c1", "c4")
FunChoice <- c(".N", "mean", "sum")
# attempt which now does just one function and doesn't add names
dt[, lapply(.SD, sum, na.rm=TRUE), by=category, .SDcols=Colchoice ]
Ожидаемый результат - строка для каждой группы и столбец для каждой функции в каждом выбранном столбце.
Category Mean c1 Sum c1 Mean c4 ...
A
B
C
D
E
......
Возможно, дубликат, но я не нашел точного ответа, который мне нужен
5 ответов
Если я правильно понимаю, этот вопрос состоит из двух частей:
- Как группировать и объединять с несколькими функциями по списку столбцов и автоматически генерировать новые имена столбцов.
- Как передать имена функций как символьный вектор.
Для части 1 это почти дубликат Применить несколько функций к нескольким столбцам в data.table, но с дополнительным требованием, чтобы результаты были сгруппированы с использованием by =,
Поэтому ответ Эдди должен быть изменен путем добавления параметра recursive = FALSE в призыве к unlist():
my.summary = function(x) list(N = length(x), mean = mean(x), median = median(x))
dt[, unlist(lapply(.SD, my.summary), recursive = FALSE),
.SDcols = ColChoice, by = category]
category c1.N c1.mean c1.median c4.N c4.mean c4.median 1: f 3974 9999.987 9999.989 3974 9.994220 9.974125 2: w 4033 10000.008 9999.991 4033 10.004261 9.986771 3: n 4025 9999.981 10000.000 4025 10.003686 9.998259 4: x 3975 10000.035 10000.019 3975 10.010448 9.995268 5: k 3957 10000.019 10000.017 3957 9.991886 10.007873 6: j 4027 10000.026 10000.023 4027 10.015663 9.998103 ...
Для части 2 нам нужно создать my.summary() из символьного вектора имен функций. Это может быть достигнуто путем "программирования на языке", то есть путем сборки выражения в виде символьной строки и, наконец, его анализа и оценки:
my.summary <-
sapply(FunChoice, function(f) paste0(f, "(x)")) %>%
paste(collapse = ", ") %>%
sprintf("function(x) setNames(list(%s), FunChoice)", .) %>%
parse(text = .) %>%
eval()
my.summary
function(x) setNames(list(length(x), mean(x), sum(x)), FunChoice) <environment: 0xe376640>
В качестве альтернативы, мы можем перебрать категории и rbind() результаты потом:
library(magrittr) # used only to improve readability
lapply(dt[, unique(category)],
function(x) dt[category == x,
c(.(category = x), unlist(lapply(.SD, my.summary))),
.SDcols = ColChoice]) %>%
rbindlist()
эталонный тест
Пока что 4 data.table и один dplyr решения были размещены. По крайней мере, один из ответов утверждает, что он "сверхбыстрый". Итак, я хотел проверить по бенчмарку с разным количеством строк:
library(data.table)
library(magrittr)
bm <- bench::press(
n = 10L^(2:6),
{
set.seed(12212018)
dt <- data.table(
index = 1:n,
category = sample(letters[1:25], n, replace = T),
c1 = rnorm(n, 10000),
c2 = rnorm(n, 1000),
c3 = rnorm(n, 100),
c4 = rnorm(n, 10)
)
# use set() instead of <<- for appending additional columns
for (i in 5:100) set(dt, , paste0("c", i), rnorm(n, 1000))
tables()
ColChoice <- c("c1", "c4")
FunChoice <- c("length", "mean", "sum")
my.summary <- function(x) list(length = length(x), mean = mean(x), sum = sum(x))
bench::mark(
unlist = {
dt[, unlist(lapply(.SD, my.summary), recursive = FALSE),
.SDcols = ColChoice, by = category]
},
loop_category = {
lapply(dt[, unique(category)],
function(x) dt[category == x,
c(.(category = x), unlist(lapply(.SD, my.summary))),
.SDcols = ColChoice]) %>%
rbindlist()
},
dcast = {
dcast(dt, category ~ 1, fun = list(length, mean, sum), value.var = ColChoice)
},
loop_col = {
lapply(ColChoice, function(col)
dt[, setNames(lapply(FunChoice, function(f) get(f)(get(col))),
paste0(col, "_", FunChoice)),
by=category]
) %>%
Reduce(function(x, y) merge(x, y, by="category"), .)
},
dplyr = {
dt %>%
dplyr::group_by(category) %>%
dplyr::summarise_at(dplyr::vars(ColChoice), .funs = setNames(FunChoice, FunChoice))
},
check = function(x, y)
all.equal(setDT(x)[order(category)],
setDT(y)[order(category)] %>%
setnames(stringr::str_replace(names(.), "_", ".")),
ignore.col.order = TRUE,
check.attributes = FALSE
)
)
}
)
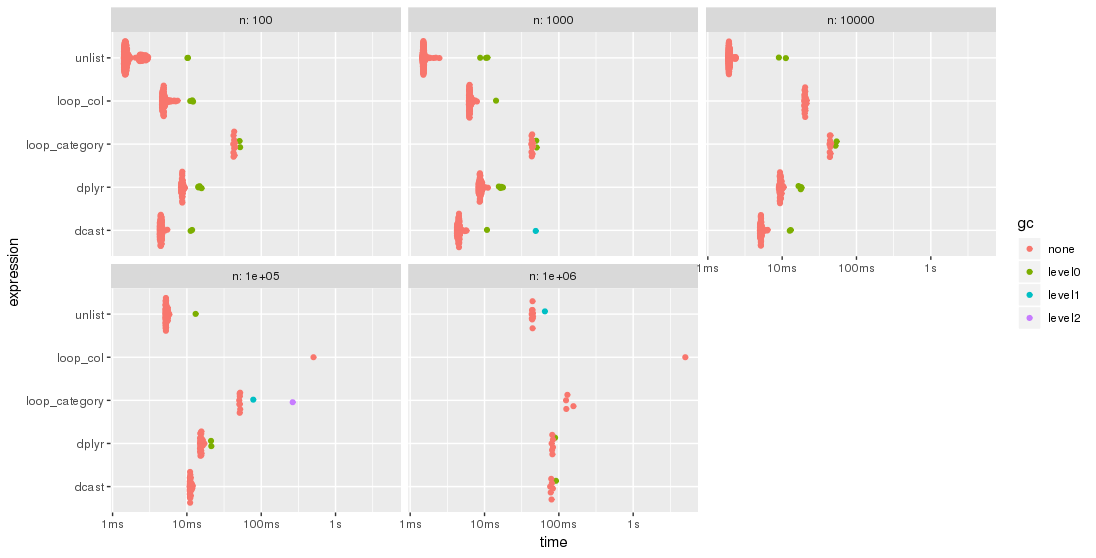
Результаты легче сравнивать при построении графика:
library(ggplot2)
autoplot(bm)
Пожалуйста, обратите внимание на логарифмическую шкалу времени.
Для этого тестового случая методunlist всегда самый быстрый, за которым следуетdcast.dplyr догоняет большие проблемыn, Оба подхода lapply / loop менее производительны. В частности, подход Parfait для обхода столбцов и последующего слияния подрезультатов кажется довольно чувствительным к размеру проблемы.n,
Изменить: второй тест
По предложению jangorecki, я повторил тест с гораздо большим количеством строк, а также с различным количеством групп. Из-за ограничений памяти наибольший размер проблемы составляет 10 М строк на 102 столбца, что занимает 7,7 ГБ памяти.
Итак, первая часть кода тестов изменена на
bm <- bench::press(
n_grp = 10^(1:3),
n_row = 10L^seq(3, 7, by = 2),
{
set.seed(12212018)
dt <- data.table(
index = 1:n_row,
category = sample(n_grp, n_row, replace = TRUE),
c1 = rnorm(n_row),
c2 = rnorm(n_row),
c3 = rnorm(n_row),
c4 = rnorm(n_row, 10)
)
for (i in 5:100) set(dt, , paste0("c", i), rnorm(n_row, 1000))
tables()
...
Как и ожидал Джангорецкий, некоторые решения более чувствительны к количеству групп, чем другие. В частности, производительность loop_category значительно снижается с увеличением количества групп, в то время как dcast, похоже, менее подвержен влиянию. Для меньшего количества групп подход с использованием списков всегда быстрее, чем dcast, в то время как для многих групп dcast быстрее. Тем не менее, для больших размеров проблемы unlist, кажется, опережает dcast.
Вот ответ data.table:
funs_list <- lapply(FunChoice, as.symbol)
dcast(dt, category~1, fun=eval(funs_list), value.var = Colchoice)
Это супер быстро и делает то, что вы хотите.
Подумайте о создании списка таблиц данных, в котором вы просматриваете каждый ColChoice и применяете каждую функцию FuncChoice (соответственно устанавливая имена). Затем, чтобы объединить все таблицы данных вместе, запустите merge в Reduce вызов. Также используйте get извлекать объекты среды (функции / столбцы).
Примечание: ColChoice был переименован для случая верблюда и length функция заменяет .N для функциональной формы для подсчета:
set.seed(12212018) # RUN BEFORE data.table() BUILD TO REPRODUCE OUTPUT
...
ColChoice <- c("c1", "c4")
FunChoice <- c("length", "mean", "sum")
output <- lapply(ColChoice, function(col)
dt[, setNames(lapply(FunChoice, function(f) get(f)(get(col))),
paste0(col, "_", FunChoice)),
by=category]
)
final_dt <- Reduce(function(x, y) merge(x, y, by="category"), output)
head(final_dt)
# category c1_length c1_mean c1_sum c4_length c4_mean c4_sum
# 1: a 3893 10000.001 38930003 3893 9.990517 38893.08
# 2: b 4021 10000.028 40210113 4021 9.977178 40118.23
# 3: c 3931 10000.008 39310030 3931 9.996538 39296.39
# 4: d 3954 10000.010 39540038 3954 10.004578 39558.10
# 5: e 4016 9999.998 40159992 4016 10.002131 40168.56
# 6: f 3974 9999.987 39739947 3974 9.994220 39717.03
Кажется, что нет простого ответа с использованием data.table, так как никто еще не ответил на это. Поэтому я предложу ответ на основе dplyr, который должен делать то, что вы хотите. Я использую встроенный набор данных радужной оболочки для примера:
library(dplyr)
iris %>%
group_by(Species) %>%
summarise_at(vars(Sepal.Length, Sepal.Width), .funs = c(sum=sum,mean= mean), na.rm=TRUE)
## A tibble: 3 x 5
# Species Sepal.Length_sum Sepal.Width_sum Sepal.Length_mean Sepal.Width_mean
# <fct> <dbl> <dbl> <dbl> <dbl>
#1 setosa 245. 171. 5.00 3.43
#2 versicolor 297. 138. 5.94 2.77
#3 virginica 323. 149. 6.60 2.97
или используя ввод символов для столбцов и функций:
Colchoice <- c("Sepal.Length", "Sepal.Width")
FunChoice <- c("mean", "sum")
iris %>%
group_by(Species) %>%
summarise_at(vars(Colchoice), .funs = setNames(FunChoice, FunChoice), na.rm=TRUE)
## A tibble: 3 x 5
# Species Sepal.Length_mean Sepal.Width_mean Sepal.Length_sum Sepal.Width_sum
# <fct> <dbl> <dbl> <dbl> <dbl>
#1 setosa 5.00 3.43 245. 171.
#2 versicolor 5.94 2.77 297. 138.
#3 virginica 6.60 2.97 323. 149.
Если итоговую статистику вам нужно вычислить, mean, .N, и, возможно) median, который data.table Оптимизируя код C, вы можете получить более высокую производительность, если преобразуете таблицу в длинную форму, чтобы вы могли выполнять вычисления так, чтобы таблица данных могла их оптимизировать:
> library(data.table)
> n = 100000
> dt = data.table(index=1:100000,
category = sample(letters[1:25], n, replace = T),
c1=rnorm(n,10000),
c2=rnorm(n,1000),
c3=rnorm(n,100),
c4 = rnorm(n,10)
)
> {lapply(c(paste('c', 5:100, sep ='')), function(addcol) dt[[addcol]] <<- rnorm(n,1000) ); dt}
> Colchoice <- c("c1", "c4")
> dt[, .SD
][, c('index', 'category', Colchoice), with=F
][, melt(.SD, id.vars=c('index', 'category'))
][, mean := mean(value), .(category, variable)
][, median := median(value), .(category, variable)
][, N := .N, .(category, variable)
][, value := NULL
][, index := NULL
][, unique(.SD)
][, dcast(.SD, category ~ variable, value.var=c('mean', 'median', 'N')
]
category mean_c1 mean_c4 median_c1 median_c4 N_c1 N_c4
1: a 10000 10.021 10000 10.041 4128 4128
2: b 10000 10.012 10000 10.003 3942 3942
3: c 10000 10.005 10000 9.999 3926 3926
4: d 10000 10.002 10000 10.007 4046 4046
5: e 10000 9.974 10000 9.993 4037 4037
6: f 10000 10.025 10000 10.015 4009 4009
7: g 10000 9.994 10000 9.998 4012 4012
8: h 10000 10.007 10000 9.986 3950 3950
...