R: Найти кратчайший геодезический путь между 2 точками двумерного облака точек
Я создал следующий график с помощью двух функций, написанных Винсентом Зоонекендом (вы можете найти их здесь) (мой код приведен в конце поста).
Чтобы можно было объяснить, что такое граф окрестностей и этот параметр "k", который использует сопоставление изометрических объектов. "k" указывает, сколько точек каждая точка напрямую связана с. Их расстояние - это просто евклидово расстояние друг от друга. Расстояние между любой точкой и ее (k + 1) ближайшей точкой (или любой точкой, находящейся дальше) называется "геодезической" и представляет собой наименьшую сумму всех длин ребер, необходимых для ее достижения. Иногда это намного больше, чем евклидово расстояние. Это касается точек A и B на моей фигуре.
Теперь я хочу добавить черную линию, показывающую геодезическое расстояние от точки A до точки B. Я знаю о команде segments(), который, вероятно, будет лучшим для добавления линии, и я знаю, что один алгоритм, чтобы найти кратчайший путь (геодезическое расстояние), является алгоритмом Дейкстры и что он реализован в пакете igraph, Тем не менее, я не могу igraph не интерпретировать мой график и не найти точки (вершины), которые нужно пройти (и их координаты) самостоятельно.
Кстати, если k = 18, т. Е. Если каждая точка напрямую связана с 18 ближайшими точками, геодезическое расстояние между A и B будет просто евклидовым расстоянием.
isomap.incidence.matrix <- function (d, eps=NA, k=NA) {
stopifnot(xor( is.na(eps), is.na(k) ))
d <- as.matrix(d)
if(!is.na(eps)) {
im <- d <= eps
} else {
im <- apply(d,1,rank) <= k+1
diag(im) <- FALSE
}
im | t(im)
}
plot.graph <- function (im,x,y=NULL, ...) {
if(is.null(y)) {
y <- x[,2]
x <- x[,1]
}
plot(x,y, ...)
k <- which( as.vector(im) )
i <- as.vector(col(im))[ k ]
j <- as.vector(row(im))[ k ]
segments( x[i], y[i], x[j], y[j], col = "grey")
}
z <- seq(1.1,3.7,length=140)*pi
set.seed(4)
zz <- rnorm(1:length(z))+z*sin(z)
zz <- cbind(zz,z*cos(z)*seq(3,1,length=length(z)))
dist.grafik <- dist(zz)
pca.grafik <- princomp(zz)
x11(8, 8)
par(mar=c(0,0,0,0))
plot.graph(isomap.incidence.matrix(dist.grafik, k=3), pca.grafik$scores[,1], pca.grafik$scores[,2],
xaxt = "n", yaxt = "n", xlab = "", ylab = "", cex = 1.3)
legend("topright", inset = 0.02, legend = "k = 3", col = "grey", lty = 1, cex = 1.3)
segments(x0 = -8.57, y0 = -1.11, x1 = -10.83, y1 = -5.6, col = "black", lwd = 2, lty = "dashed")
text(x = -8.2, y = -1.4, labels = "A", font = 2, cex = 1.2)
text(x = -11, y = -5.1, labels = "B", font = 2, cex = 1.2)
1 ответ
Следующий код может вам помочь, он использует ваши данные для создания объекта igraph с весом, который в вашем случае равен евклидову расстоянию между узлами. Затем вы найдете взвешенный кратчайший путь, который возвращается sp$vpath[[1]], В следующем примере это кратчайший путь между узлами № 5 и 66. Я отредактировал код с решением для построения из mattu
isomap.incidence.matrix <- function (d, eps=NA, k=NA) {
stopifnot(xor( is.na(eps), is.na(k) ))
d <- as.matrix(d)
if(!is.na(eps)) {
im <- d <= eps
} else {
im <- apply(d,1,rank) <= k+1
diag(im) <- FALSE
}
im | t(im)
}
plot.graph <- function (im,x,y=NULL, ...) {
if(is.null(y)) {
y <- x[,2]
x <- x[,1]
}
plot(x,y, ...)
k <- which( as.vector(im) )
i <- as.vector(col(im))[ k ]
j <- as.vector(row(im))[ k ]
segments( x[i], y[i], x[j], y[j], col = "grey")
}
z <- seq(1.1,3.7,length=100)*pi
set.seed(4)
zz <- rnorm(1:length(z))+z*sin(z)
zz <- cbind(zz,z*cos(z)*seq(3,1,length=length(z)))
dist.grafik <- as.matrix(dist(zz))
pca.grafik <- princomp(zz)
isomap.resul <- function (d, eps=NA, k=NA) {
a <- isomap.incidence.matrix(d, eps, k)
b <- dist.grafik
res <- a * b
return(res)
}
a <- graph_from_adjacency_matrix(isomap.resul(dist.grafik, k=3),
mode = c("undirected"), weight = TRUE)
sp <- shortest_paths(a, 5, to = 66, mode = c("out", "all", "in"),
weights = NULL, output = c("vpath", "epath", "both"),
predecessors = FALSE, inbound.edges = FALSE)
path <- sp$vpath[[1]]
x11(8, 8)
par(mar=c(0,0,0,0))
plot.graph(isomap.incidence.matrix(dist.grafik, k=3), pca.grafik$scores[,1], pca.grafik$scores[,2],
xaxt = "n", yaxt = "n", xlab = "", ylab = "", cex = 1.3)
legend("topright", inset = 0.02, legend = "k = 3", col = "grey", lty = 1, cex = 1.3)
segments(x0 = -8.57, y0 = -1.11, x1 = -10.83, y1 = -5.6, col = "black", lwd = 2, lty = "dashed")
text(x = -8.2, y = -1.4, labels = "A", font = 2, cex = 1.2)
text(x = -11, y = -5.1, labels = "B", font = 2, cex = 1.2)
for(i in 2:length(path)){
aa <- pca.grafik$scores[path[i-1], 1]
bb <- pca.grafik$scores[path[i-1], 2]
cc <- pca.grafik$scores[path[i], 1]
dd <- pca.grafik$scores[path[i], 2]
segments(aa, bb, cc , dd, lwd = 2)
}
Чтобы запустить этот скрипт, вам, очевидно, нужен пакет igraph,
Мне кажется, это самый короткий путь в зависимости от геодезического расстояния. 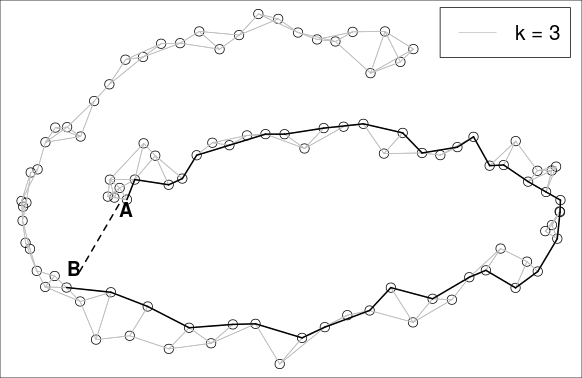
Надеюсь, поможет.
