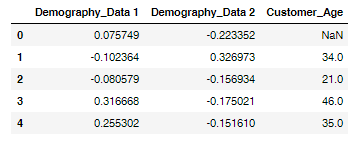
Я получаю неожиданное значение NaN при попытке pd.concat. Как с этим бороться? PCA против T-SNE
Я пытаюсь уменьшить размерность данных с помощью PCA, однако, когда я использую concat, он автоматически генерирует значение NaN. Также возраст клиента стал плавать, пока он был int. Может кто-нибудь сказать, пожалуйста, как я могу решить эту проблему? Также было бы очень признательно, если вы скажите мне, следует ли мне использовать PCA или tSNE для визуализации данных с 14 переменными (в которых есть столбец, который просто содержит 4 разные переменные (1,2,3,4) из 12000). значения, есть два столбца с логическими значениями).
# Separating out the Demographic Data.
x = Demo_Data.values
# Separating out the Target as regions.
y = df2.loc[:,['Customer_Age']].values
# Standardizing the features
scaler = StandardScaler()
x = scaler.fit_transform(x)
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
Demography_Data = pca.fit_transform(x)
principalDf = pd.DataFrame(data = Demography_Data
, columns = ['Demography_Data 1', 'Demography_Data 2'])
finalDf = pd.concat([principalDf, df2[['Customer_Age']]], axis = 1)
1 ответ
Решение
index в вашем DataFrameне совпадают:
>>> import pandas as pd
>>> df1 = pd.DataFrame([11,22,33])
>>> df2 = pd.DataFrame([111,222,333], index=[1,2,3])
>>> pd.concat((df1,df2),axis=1)
0 0
0 11.0 NaN
1 22.0 111.0
2 33.0 222.0
3 NaN 333.0
тем не мение:
>>> df2.index=df1.index
>>> pd.concat((df1,df2),axis=1)
0 0
0 11 111
1 22 222
2 33 333