corrplot показывает незначительные коэффициенты корреляции, даже если установлен insig = "blank"
Мне нравится использовать корреляционный график, используя corrplot функция с коэффициентами корреляции, напечатанными в ячейках (используя addCoef.col а также addCoefasPercent = TRUE). Мне также нравится удалять незначительные корреляции из сюжета (используя insig = "blank"). Проблема в том, что это работает только для цвета фона, но не для самого коэффициента, поэтому сам коэффициент все еще печатается! Увидеть:
set.seed(123)
par(cex=0.8) # trick for cor. coef font size, see http://stackru.com/q/26574054/684229
col1 <-rainbow(100, s = 1, v = 1, start = 0, end = 0.9, alpha = 1)
test <- matrix(data=rnorm(400),nrow=20,ncol=20)
cor.mtest <- function(mat, conf.level = 0.95){
mat <- as.matrix(mat)
n <- ncol(mat)
p.mat <- lowCI.mat <- uppCI.mat <- matrix(NA, n, n)
diag(p.mat) <- 0
diag(lowCI.mat) <- diag(uppCI.mat) <- 1
for(i in 1:(n-1)){
for(j in (i+1):n){
tmp <- cor.test(mat[,i], mat[,j], conf.level = conf.level)
p.mat[i,j] <- p.mat[j,i] <- tmp$p.value
lowCI.mat[i,j] <- lowCI.mat[j,i] <- tmp$conf.int[1]
uppCI.mat[i,j] <- uppCI.mat[j,i] <- tmp$conf.int[2]
}
}
return(list(p.mat, lowCI.mat, uppCI.mat))
}
cor1 <- cor.mtest(test, 0.95)
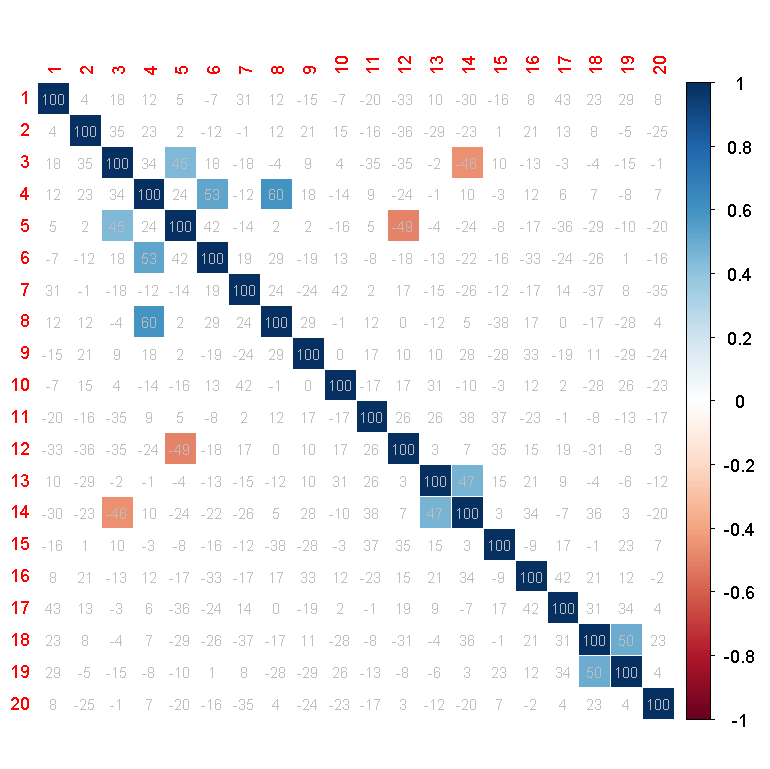
corrplot(cor(test), p.mat = cor1[[1]], insig = "blank", method = "color", addCoef.col="grey",
order = "AOE", tl.cex = 1/par("cex"),
cl.cex = 1/par("cex"), addCoefasPercent = TRUE)
Теперь вы можете видеть, что коэффициенты печатаются также для незначительных ячеек:
Просто чтобы увидеть, какие ячейки незначительны, вы можете использовать эту команду:
corrplot(cor(test), p.mat = cor1[[1]], insig = "pch", method = "color", addCoef.col="grey",
order = "AOE", tl.cex = 1/par("cex"),
cl.cex = 1/par("cex"), addCoefasPercent = TRUE)
Возможно, это ошибка пакета corrplot?
Как можно избавиться от печати коэффициента в незначительных ячейках?
2 ответа
Вы должны сделать небольшую работу для этого. Вам нужно определить вектор цветов вручную для значений p, которые передаются в addCoef.col
Если вы заказывали в алфавитном порядке, это прямо
mycol <- ifelse(c(cor1[[1]] < 0.05), "black", "white")
corrplot(cor(test), p.mat = cor1[[1]] , insig = "blank", method = "color",
addCoef.col=mycol ,
order = "original", tl.cex = 1/par("cex"),
cl.cex = 1/par("cex"), addCoefasPercent = TRUE)
Но так как вы хотите упорядочить по собственным значениям, вам нужно рассчитать порядок за пределами corrplot функция
ord <- corrMatOrder(cor(test), order="AOE")
M <- cor(test)[ord, ord]
pval <- psych::corr.test(data.frame(test), adjust="none")$p[ord, ord]
mycol <- ifelse(c(pval < 0.05), "black", "white")
corrplot(M, p.mat = pval , insig = "blank", method = "color", addCoef.col=mycol ,
order = "original", tl.cex = 1/par("cex"),
cl.cex = 1/par("cex"), addCoefasPercent = TRUE)
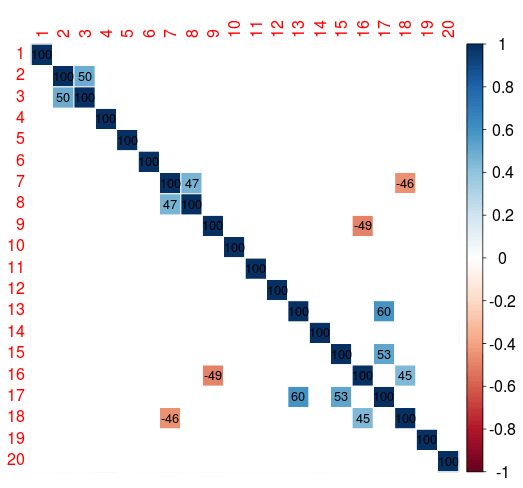
РЕДАКТИРОВАТЬ комментарии re @Masi
Чтобы обновить ограничения на цветовой панели, установите ограничения с помощью cl.lim
corrplot(cor(test), p.mat = cor1[[1]] , insig = "blank", method = "color",
addCoef.col=mycol , addCoefasPercent=TRUE,
order = "original", cl.lim = c(-100, 100))
Если вы не слишком разборчивы, вы также можете оставить фон белым и сделать addCoef.col = "white" вместо "grey" как у вас это изначально. Это избавит от необходимости указывать порядок и ifelse.