Пересортируйте непрерывные растровые данные в дискретные классы с дискретными цветами
Я бы хотел:
Реклассифицировать растр
rasна девять классов, используяreclassifyУкажите цветовую клавишу со значениями, написанными рядом с каждым цветом (см. Примерный график ниже). Цветовая клавиша не должна быть разделена, как на образце графика, а скорее показана в виде одной цветовой шкалы с 9 цветами.
Показать данные с помощью
levelplotфункция (это я могу сделать)
Спасибо за вашу помощь. AEZ
library(rasterVis)
Пожалуйста, используйте эти цвета:
col <- colorRampPalette(c("yellow4", "yellow", "orange", "red3", "darkred"))
levelplot(reclassras, col.regions = col)
Ниже dput моего растрового слоя:
ras=new("RasterLayer"
, file = new(".RasterFile"
, name = ""
, datanotation = "FLT4S"
, byteorder = "little"
, nodatavalue = -Inf
, NAchanged = FALSE
, nbands = 1L
, bandorder = "BIL"
, offset = 0L
, toptobottom = TRUE
, blockrows = 4L
, blockcols = 230L
, driver = ""
, open = FALSE
)
, data = new(".SingleLayerData"
, values = c(NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,
NA, 0.693928846555399, 0.788672037295368, NA, NA, NA, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,
NA, NA, NA, NA, NA, 0.63077312659247, 0.646355947246998, 0.877268680738184,
1.18198745233929, 1.17243141777504, 1.16780606010241, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,
NA, NA, 0.796574503892096, 0.619565545705973, 0.548990505935034,
0.595313378137405, 0.661379946396662, 0.780858688754648, 1.32843871685037,
1.3513059907622, 1.47504896477768, 1.33220740188043, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,
1.02493198234592, 0.871180733341381, 0.588561527602423, 0.495423050558386,
0.398352388696117, 0.609004794796874, 0.757493518742196, 0.968962672868491,
1.3989459779932, 1.66210327179263, 1.4993019355018, NA, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, 0.605206815154586,
0.542575959157491, 0.509368162877491, 0.461856452801692, 0.543152629728101,
0.630122267439296, 0.70810504838076, 0.788478557501378, 0.918580431438071,
0.925176992886461, 0.712139151650403, NA, NA, NA, NA, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, 0.523779588981426, 0.497759414675531,
0.539521493581999, 0.472379195067506, 0.503370029793095, 0.549265480778097,
0.570528210136693, 0.710933566259782, 0.726885712871992, 0.755087833088479,
0.692253167927366, 0.632483742020028, 0.572268262363865, 0.438810629423197,
0.495519373283201, 0.461307895792857, 0.413954466042644, 0.368257331706444,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, 0.777296738735527,
0.750850589920605, 0.513284076851117, 0.491285172798063, 0.555785245510632,
0.611268469163931, 0.813189823687993, 0.756674620262126, 0.872121251992992,
0.613545590220432, 0.481851214001446, 0.496306521393404, 0.437621615262168,
0.339745859876615, 0.303638244461432, 0.311408628670555, 0.307152533981348,
0.31488062771456, 0.324014538449246, NA, NA, NA, NA, NA, NA,
NA, NA, NA, NA, 0.682358704297568, 0.706551558881091, 0.502018311967365,
0.456684326929721, 0.473559918237202, 0.463389646751083, 0.441190726838794,
0.439217524749229, 0.584294316557062, 0.267633271553189, 0.256528025697032,
0.337787793432092, 0.330618969832041, 0.13803984358201, 0.157341965515659,
0.25899384267949, 0.217328457675264, 0.242480963818602, 0.272211051701126,
0.281644918669069, NA, NA, NA, NA, NA, NA, NA, NA, 0.414616765967516,
0.460428280883944, 0.439029889808256, 0.373948128082835, 0.352871572842356,
0.357951354005567, 0.328436868901166, 0.203201513096291, 0.141472917108049,
0.269096407445964, 0.15745212479733, 0.135179353417107, 0.219985246649717,
0.283005986377796, 0.209135929188723, 0.195612120899385, 0.163726230506747,
0.117276078938131, 0.169752099589576, 0.20586142557315, 0.244209631644345,
NA, NA, NA, NA, NA, 0.214238769725864, 0.208010897733397, 0.222208413054847,
0.212686463354896, 0.281732716326432, 0.300708262469288, 0.223204487650159,
0.200983714892431, 0.156651839437054, 0.224474592578023, 0.145079279318955,
0.137658404085203, 0.196204204670511, 0.157445860015357, 0.205907596393374,
0.250666664848486, 0.277186662044126, 0.265355210400489, 0.241808654124976,
0.199713317093066, 0.143711490248281, 0.0842822747178765, NA,
NA, NA, NA, 0.264092813395827, NA, 0.212441257972578, 0.189725290483582,
0.168955415365627, 0.158197543230828, 0.147705597579244, 0.157228804810736,
0.211249178305103, 0.222557582448186, 0.215124944446001, 0.210152099165574,
0.224764230646078, 0.192500267075076, 0.174923484036373, 0.121073670585186,
0.140937204004106, 0.213437474831745, 0.248947340425651, 0.265571783211699,
0.266099597629565, 0.254099129610638, 0.225777564711763, 0.178602219399,
NA, NA, NA, 0.245068225382459, 0.250977120426067, 0.255618295682899,
0.215763680642785, 0.192426673071868, 0.15695069948531, 0.113984884275593,
0.132801862169494, 0.166848777379759, 0.115940552527949, 0.157310632213977,
0.227022907248424, 0.239855727768449, 0.239340572413914, 0.231675634554819,
0.215388496476701, 0.185506053849589, 0.104971870404521, 0.112957653991221,
0.184468284077724, 0.222974948120878, 0.234747755547275, 0.240936436264378,
0.246124530363775, 0.242871371021741, NA, NA, NA, NA, 0.237419647069358,
0.238880206380025, 0.231523511204313, 0.179362897056799, 0.16381508377734,
0.133186183020653, 0.0908146529276369, 0.141301676274407, 0.17155039695241,
0.176667304881711, 0.192368230921037, 0.21608936753556, 0.226766198129308,
0.221609735358686, 0.196435659994756, 0.18030618027393, 0.178648959768383,
0.131173639965412, 0.0673928945097901, 0.144030029855376, 0.183644454323076,
0.164135425763568, 0.177071512975595, 0.218027665373501, 0.24020227724051,
NA, NA, NA, NA, 0.222719673381422, 0.203163737836875, 0.193893900175302,
0.173629261933378, 0.118612596801245, 0.120337304279883, 0.132270235949265,
0.145957944294342, 0.158947316774427, 0.171811369315789, 0.178481215022077,
0.182769116915529, 0.193576475278923, 0.187526186900056, 0.116642925509998,
0.0810130524927432, 0.155361056173399, 0.148275096869829, 0.110248050354185,
0.150980011296809, 0.149035884104822, 0.0943587027445095, NA,
NA, NA, NA, NA, NA, NA, NA, NA, 0.155642765186448, 0.165539811602326,
0.138858875725954, 0.115966143137369, 0.110762436834703, 0.116558297180592,
0.109461897495481, 0.137020886546595, 0.130569369589671, 0.106845763992692,
0.139732412594499, 0.167204357520135, 0.121743769239998, 0.0978888268526515,
0.161034204706105, 0.182178539628265, 0.179821789305561, 0.183479109899686,
0.160540641903203, 0.112272216175425, NA, NA, NA, NA, NA, NA,
NA, NA, NA, 0.163697841087448, 0.17328205868045, 0.160040552656876,
0.134655975429996, 0.12409849785916, 0.107880400167541, 0.0924636872864672,
0.103225290747982, 0.0767026155342135, 0.0661780888537736, 0.112966312114504,
0.16666404802081, 0.172067566098574, 0.172700784074864, 0.192001569629986,
0.206098509476636, 0.210359965210036, 0.209377663322926, 0.197655864616846,
0.180297988050893, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, 0.181675191082483,
0.176935554759326, 0.16390655974363, 0.154194456563158, 0.142896144640483,
0.133243173249838, 0.114015642056713, 0.0928738807683648, 0.116223803463035,
0.146600649670514, 0.180171520048675, 0.197023191340036, 0.203375499409087,
NA, NA, NA, NA, 0.220951932247611, 0.216562385948588, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, 0.176899006540279,
0.169734384672319, 0.164263225996517, 0.143295516102167, 0.13599420407605,
NA, 0.177143713627865, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, 0.189498742688049,
0.185499135750397, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,
NA, NA, NA, NA, NA, NA, NA, NA, NA, NA)
, offset = 0
, gain = 1
, inmemory = TRUE
, fromdisk = FALSE
, isfactor = FALSE
, attributes = list()
, haveminmax = TRUE
, min = 0.0661780888537736
, max = 1.66210327179263
, band = 1L
, unit = ""
, names = "myfile"
)
, legend = new(".RasterLegend"
, type = character(0)
, values = logical(0)
, color = logical(0)
, names = logical(0)
, colortable = logical(0)
)
, title = character(0)
, extent = new("Extent"
, xmin = 73.39696
, xmax = 96.59696
, ymin = 33.98298
, ymax = 49.18298
)
, rotated = FALSE
, rotation = new(".Rotation"
, geotrans = numeric(0)
, transfun = function ()
NULL
)
, ncols = 29L
, nrows = 19L
, crs = new("CRS"
, projargs = "+proj=longlat +a=6378140 +b=6356755.288157528 +no_defs"
)
, history = list()
, z = list()
)
1 ответ
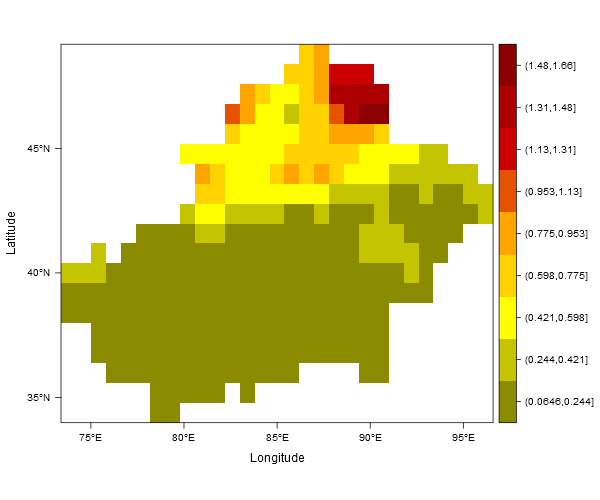
Вы можете cut ваш растр, а затем укажите цветовые границы с atи colorkey разрывается со списком, содержащим at а также labelsперешел к colorkey Arg:
levelplot(cut(ras, 9), col.regions=col,
at=0:9, margin=FALSE,
colorkey=list(labels=list(at=0:8 + 0.5, labels=levels(cut(ras[], 9)))))
Выше мы говорим, что мы хотим разделить ras в 9 бункеров равной ширины. Эти ячейки будут обозначены номерами от 1 до 9, и at=0:9 указывает, что мы хотим, чтобы цвета менялись при значениях от 0 до 9. Чтобы правильно пометить цветовой ключ, мы передаем список с именем labelsс аргументом at указание, где мы хотим, чтобы метки были расположены, и аргумент labels предоставление соответствующих ярлыков. Обратите внимание, что вы можете передать все, что хотите labels элемент labels список передан colorkey аргумент (например, чтобы соответствовать формату вашего примера графика, вы можете использовать что-то вроде labels=list(at=0:8 + 0.5, labels=sub('\\((.+),(.+)]', '>\\1 ~ \\2', levels(cut(ras[], 9))))).
Вы могли бы вместо этого использовать ratify чтобы принудить растровый слой к фактору и установить уровни фактора для нужного текста метки, но это может быть немного затруднительно, если уровни отсутствуют.