Чтение *.mhd/*. Raw формата в python
Может кто-нибудь сказать, пожалуйста, как я могу прочитать набор данных, содержащий файлы.mhd /.raw в Python?
3 ответа
Самый простой способ - использовать SimpleITK (MedPy также использует ITK для файлов.mhd/.raw). команда
pip install SimpleITK
работает для многих версий Python. Для чтения.mhd/.raw вы можете использовать этот код из kaggle
import SimpleITK as sitk
import numpy as np
'''
This funciton reads a '.mhd' file using SimpleITK and return the image array, origin and spacing of the image.
'''
def load_itk(filename):
# Reads the image using SimpleITK
itkimage = sitk.ReadImage(filename)
# Convert the image to a numpy array first and then shuffle the dimensions to get axis in the order z,y,x
ct_scan = sitk.GetArrayFromImage(itkimage)
# Read the origin of the ct_scan, will be used to convert the coordinates from world to voxel and vice versa.
origin = np.array(list(reversed(itkimage.GetOrigin())))
# Read the spacing along each dimension
spacing = np.array(list(reversed(itkimage.GetSpacing())))
return ct_scan, origin, spacing
Использование Skimage может быть еще проще после установки SimpleITK
import skimage.io as io
img = io.imread('file.mhd', plugin='simpleitk')
Это даст вам массив с сортировкой z,y,x.
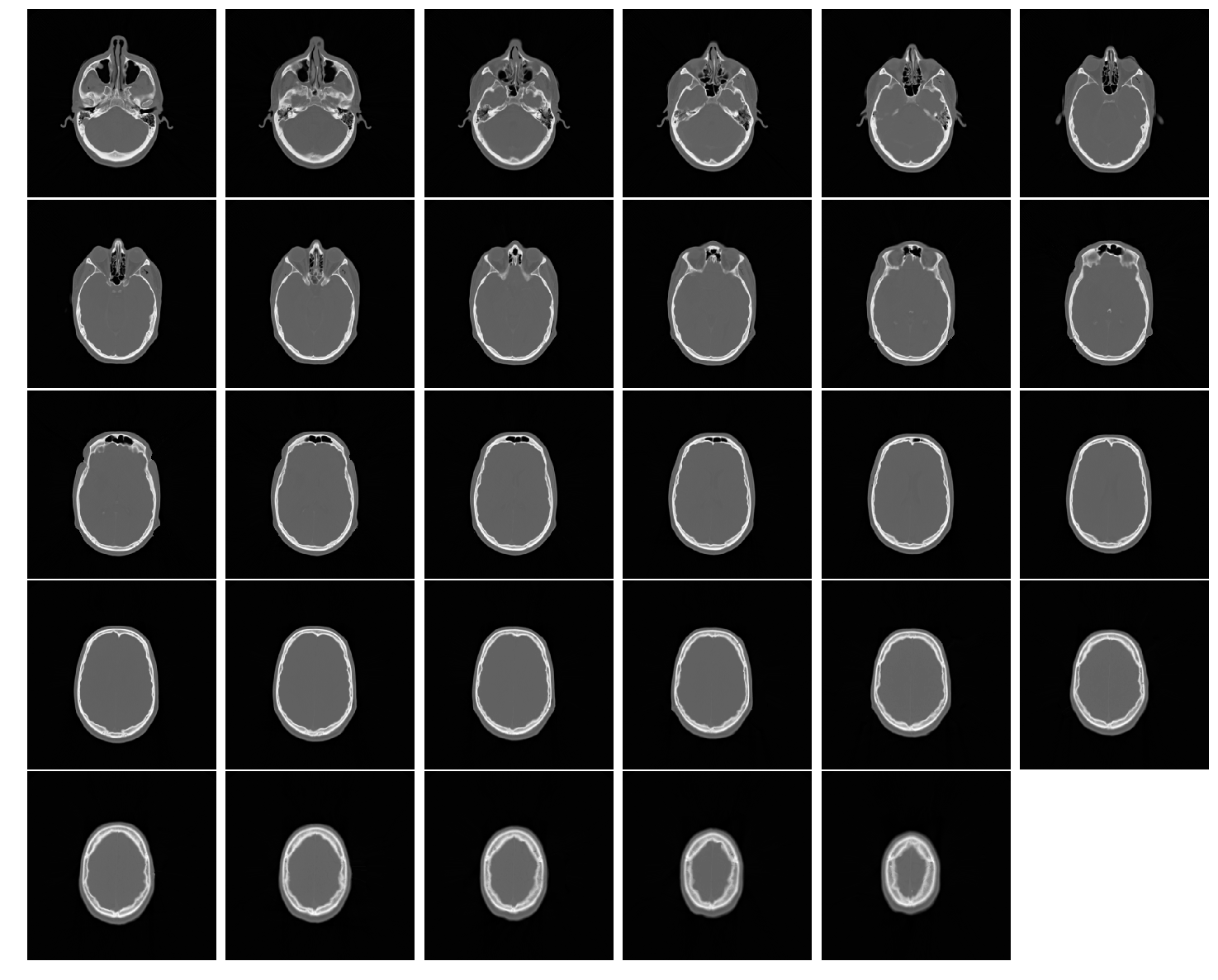
Добавив указанные выше сообщения, вы можете начать с файла CT-Scan .mhd, загруженного отсюда, и отображать / сохранять 29 изображений с помощью следующего кода (при условии, что у вас есть как заголовок, так и необработанные файлы, загруженные в текущий каталог):
import SimpleITK as sitk
import matplotlib.pylab as plt
ct_scans = sitk.GetArrayFromImage(sitk.ReadImage("training_001_ct.mhd", sitk.sitkFloat32))
plt.figure(figsize=(20,16))
plt.gray()
plt.subplots_adjust(0,0,1,1,0.01,0.01)
for i in range(ct_scans.shape[0]):
plt.subplot(5,6,i+1), plt.imshow(ct_scans[i]), plt.axis('off')
# use plt.savefig(...) here if you want to save the images as .jpg, e.g.,
plt.show()
Вот тот же файл.mhd CT-сканирования, который читается с помощью SimpleITK и анимированные:
Вы можете попробовать использовать MedPy или этот скрипт mhd_utils