Светящийся (неоновый) эффект в гнуплоте
Я хочу воспроизвести этот эффект в gnuplot:
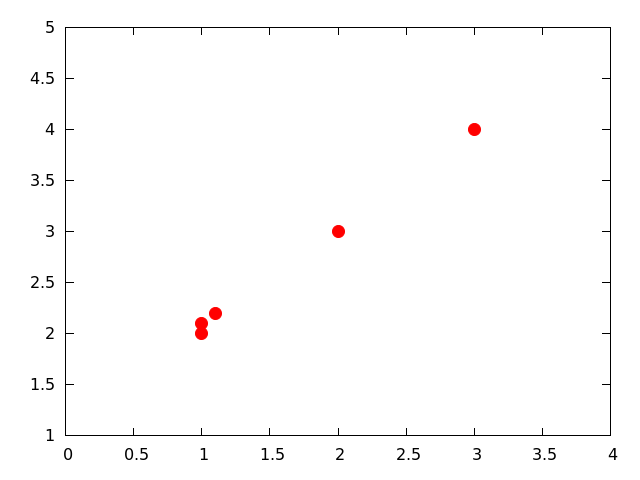
Как я могу достичь этого? Если это невозможно, какое программное обеспечение я могу использовать для его воспроизведения?
3 ответа
Использование 2d-ядра для каждого пикселя может быть сделано внутри gnuplot. Таким образом, более плотные скопления становятся ярче, чем отдельные пиксели. Проверьте show palette rgbformulae и соответствующая глава в помощь, чтобы изменить цвета.
set term wxt size 300,300 background rgb 0
set view map
set samp 140
set dgrid3d 180,180, gauss kdensity2d 0.2,0.2
set palette rgbform 4,4,3
splot "+" us 1:(sin($1/3)**2*20):(1) with pm3d notitle
Отказ от ответственности: Это можно сделать с помощью gnuplot, как указано в этом ответе, но вы, вероятно, должны рассмотреть другой инструмент для рисования этого конкретного типа графика.
Существует по крайней мере один способ сделать это, с предварительной обработкой данных. Идея состоит в том, чтобы имитировать эффект свечения, используя ядро Гаусса для смазывания точек данных. Рассмотрим следующие данные, содержащиеся в файле с именем data:
1 2
1 2.1
1.1 2.2
2 3
3 4
Я специально поместил первые три точки близко друг к другу, чтобы иметь возможность наблюдать усиленное свечение соседних точек. Эти данные выглядят так:
Теперь мы размазываем точки данных, используя двумерное ядро Гаусса. Я написал следующий код Python, чтобы помочь с этим. Код имеет ограничение 4 стандартных отклонения (sx а также sy) вокруг каждой точки. Если вы хотите, чтобы свечение было кругом, вы должны выбрать стандартные отклонения, чтобы sx / sy соотношение такое же, как отношение длин осей x/y в gnuplot. В противном случае точки будут выглядеть как эллипсы. Это код:
import numpy as np
import sys
filename = str(sys.argv[1])
sx = float(sys.argv[2])
sy = float(sys.argv[3])
def f(x,y,x0,y0,sx,sy):
return np.exp(-(x-x0)**2/2./sx**2 -(y-y0)**2/2./sy**2)
datafile = open(filename, 'r')
data = []
for datapoint in datafile:
a, b = datapoint.split()
data.append([float(a),float(b)])
xmin = data[0][0]
xmax = data[0][0]
ymin = data[0][1]
ymax = data[0][1]
for i in range(1, len(data)):
if(data[i][0] < xmin):
xmin = data[i][0]
if(data[i][0] > xmax):
xmax = data[i][0]
if(data[i][1] < ymin):
ymin = data[i][1]
if(data[i][1] > ymax):
ymax = data[i][1]
xmin -= 4.*sx
xmax += 4.*sx
ymin -= 4.*sy
ymax += 4.*sy
dx = (xmax - xmin) / 250.
dy = (ymax - ymin) / 250.
for i in np.arange(xmin,xmax+dx, dx):
for j in np.arange(ymin,ymax+dy, dy):
s = 0.
for k in range(0, len(data)):
d2 = (i - data[k][0])**2 + (j - data[k][1])**2
if( d2 < (4.*sx)**2 + (4.*sy)**2):
s += f(i,j,data[k][0],data[k][1],sx,sy)
print i, j, s
Он используется следующим образом:
python script.py data sx sy
где script.py это имя файла, в котором находится код, data имя файла данных, и sx а также sy стандартные отклонения
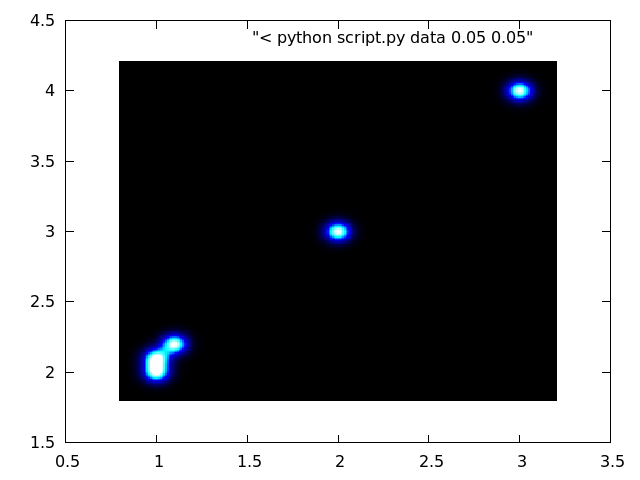
Теперь вернемся к gnuplot, мы определим палитру, которая имитирует светящийся рисунок. Для изолированных точек суммированные гауссианы дают 1 в положении точки; для перекрывающихся точек это дает значения больше 1. Вы должны учитывать это при определении палитры. Ниже приведен только пример:
set cbrange [0:3]
unset colorbox
set palette defined (0 "black", 0.5 "blue", 0.75 "cyan", 1 "white", 3 "white")
plot "< python script.py data 0.05 0.05" w image
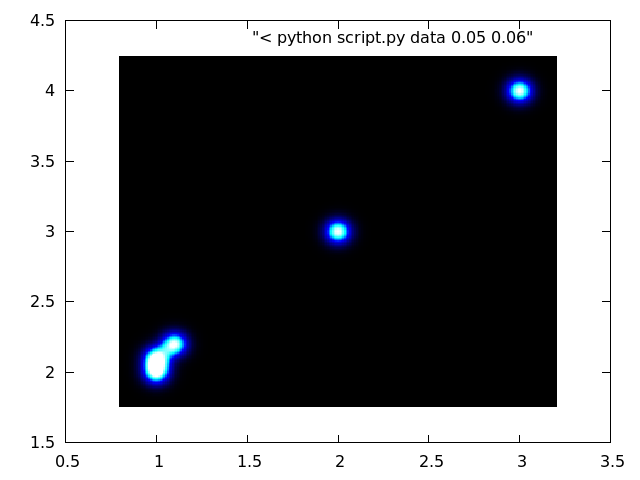
Вы можете видеть, что точки на самом деле являются эллипсами, потому что отношение длин осей не совпадает с отношением стандартных отклонений в разных направлениях. Это можно легко исправить:
plot "< python script.py data 0.05 0.06" w image
Установите черный фон, а затем нанесите свой набор данных несколько раз в разные цвета с уменьшением размера точек.
set term wxt backgr rgb "black"
plot sin(x) w p pt 7 ps 2 lc rgb 0x00003f not, \
sin(x) w p pt 7 ps 1.5 lc rgb 0x00007f not, \
sin(x) w p pt 7 ps 1 lc rgb 0x0000af not, \
sin(x) w p pt 7 ps .5 lc rgb 0x0000ff
В качестве альтернативы, некоторая комбинация splot with pm3d,set dgrid3d gauss kdensity2d, а также set view map, в сочетании с подходящей палитрой, можно использовать, см. мой другой ответ.