Как правильно построить проецируемые данные в сетке ggplot2?
Я использовал с помощью ggplot2 построить климатические данные с привязкой к сетке за годы. Обычно это проецируемые файлы NetCDF. Ячейки имеют квадратные координаты модели, но в зависимости от того, какую проекцию использует модель, в реальном мире это может быть не так.
Мой обычный подход - сначала переназначить данные на подходящую регулярную сетку, а затем построить график. Это вносит небольшую модификацию в данные, обычно это приемлемо.
Тем не менее, я решил, что это уже недостаточно хорошо: я хочу отобразить проецируемые данные напрямую, без переотображения, как другие программы (например, ncl) можно, если я не ошибаюсь, сделать, не касаясь выходных значений модели.
Однако я сталкиваюсь с некоторыми проблемами. Ниже я подробно опишу возможные решения, от самых простых до самых сложных, и их проблемы. Можем ли мы их преодолеть?
РЕДАКТИРОВАТЬ: благодаря ответу @LoBu я получил эту прекрасную функцию, которая включает в себя решение. Если вам это нравится, пожалуйста, ответьте upvote @LoBu!
Начальная настройка
#Load packages
library(raster)
library(ggplot2)
#This gives you the starting data, 's'
load(url('https://files.fm/down.php?i=kew5pxw7&n=loadme.Rdata'))
#If you cannot download the data, maybe you can try to manually download it from http://s000.tinyupload.com/index.php?file_id=04134338934836605121
#Check the data projection, it's Lambert Conformal Conic
projection(s)
#The data (precipitation) has a 'model' grid (125x125, units are integers from 1 to 125)
#for each point a lat-lon value is also assigned
pr <- s[[1]]
lon <- s[[2]]
lat <- s[[3]]
#Lets get the data into data.frames
#Gridded in model units:
pr_df_basic <- as.data.frame(pr, xy=TRUE)
colnames(pr_df_basic) <- c('lon', 'lat', 'pr')
#Projected points:
pr_df <- data.frame(lat=lat[], lon=lon[], pr=pr[])
Мы создали два кадра данных, один с координатами модели, другой с реальными поперечными точками (центрами) для каждой ячейки модели.
Необязательно: использование домена меньшего размера
Если вы хотите более четко видеть формы ячеек, вы можете поместить данные в подмножество и извлечь только небольшое количество ячеек модели. Только будьте осторожны, что вам может понадобиться отрегулировать размеры точек, границы сюжета и другие удобства. Вы можете подмножество, как это, а затем повторить вышеупомянутую часть кода (минус load()):
s <- crop(s, extent(c(100,120,30,50)))
Если вы хотите полностью понять проблему, возможно, вы захотите попробовать большой и маленький домены. код идентичен, меняются только размеры точек и границы карты. Значения ниже приведены для большого полного домена. Хорошо, теперь давайте строить сюжет!
Начать с плитки
Наиболее очевидным решением является использование плиток. Давай попробуем.
my_theme <- theme_bw() + theme(panel.ontop=TRUE, panel.background=element_blank())
my_cols <- scale_color_distiller(palette='Spectral')
my_fill <- scale_fill_distiller(palette='Spectral')
#Really unprojected square plot:
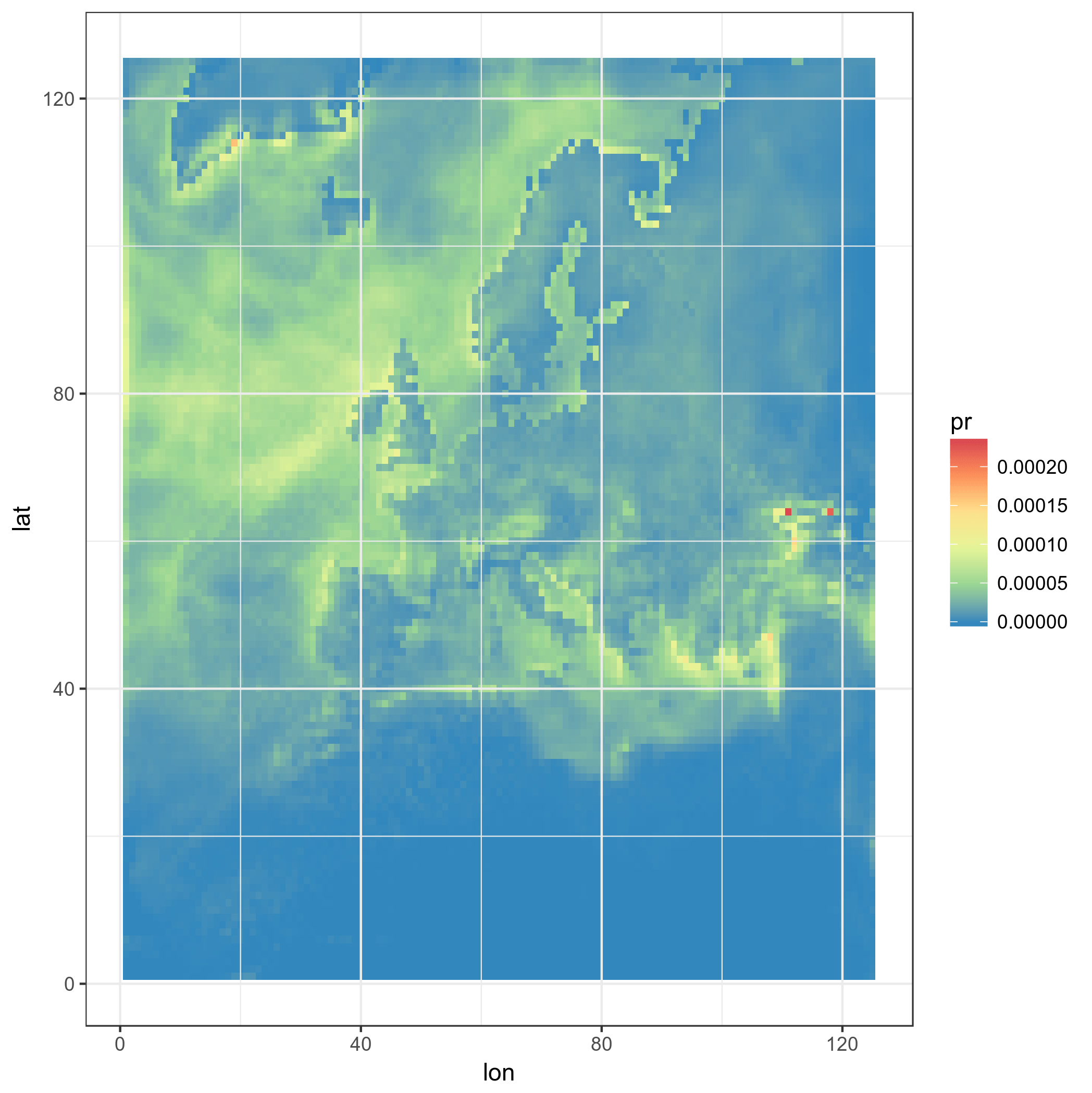
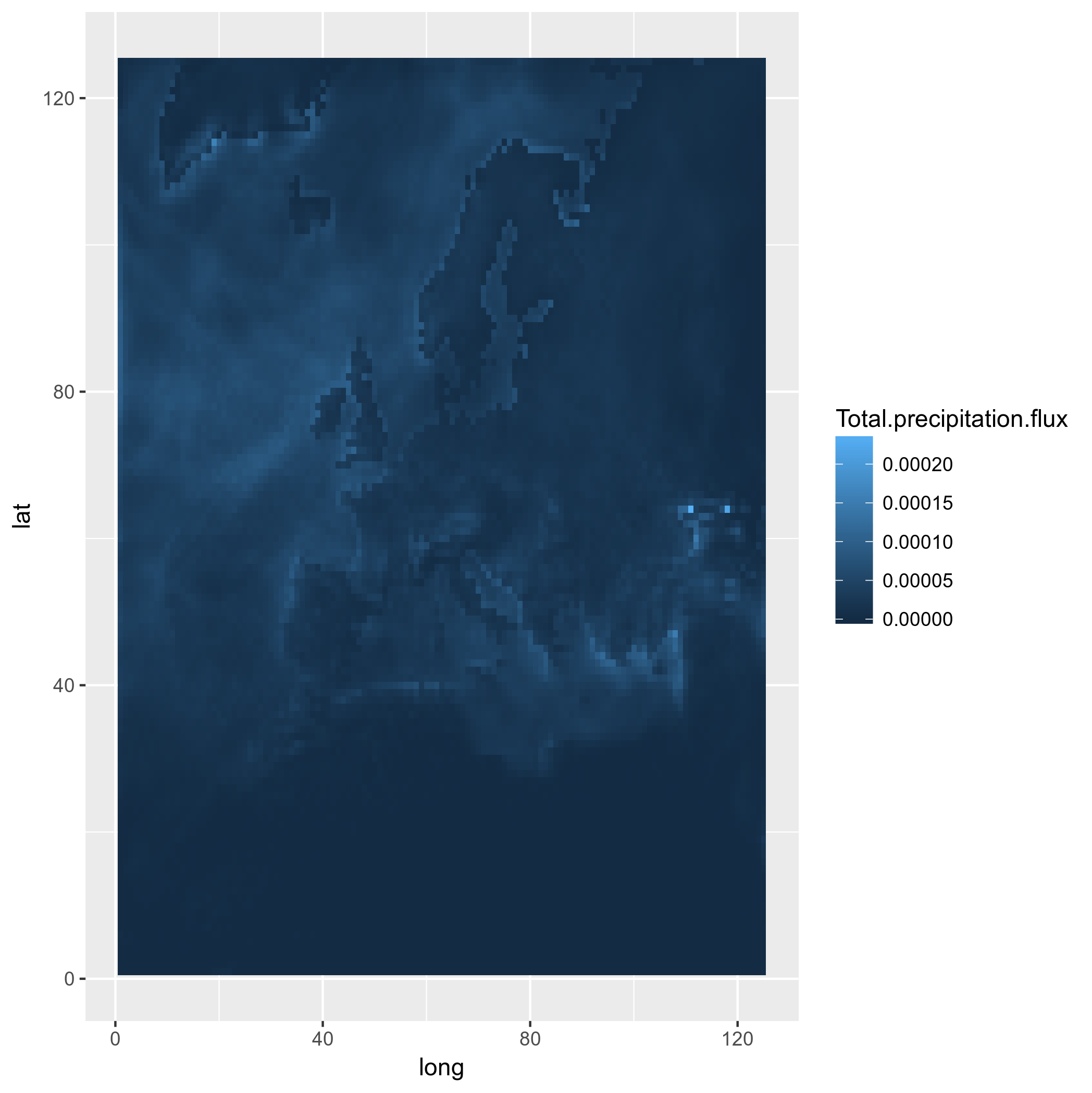
ggplot(pr_df_basic, aes(y=lat, x=lon, fill=pr)) + geom_tile() + my_theme + my_fill
Хорошо, теперь кое-что более продвинутое: мы используем настоящий LAT-LON, используя квадратные плитки
ggplot(pr_df, aes(y=lat, x=lon, fill=pr)) + geom_tile(width=1.2, height=1.2) +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_theme + my_fill +
coord_quickmap(xlim=range(pr_df$lon), ylim=range(pr_df$lat)) #the result is weird boxes...
Хорошо, но это не настоящие квадраты модели, это взлом. Кроме того, блоки моделей расходятся в верхней части домена и все ориентированы одинаково. Не хорошо. Давайте спроецируем сами квадраты, хотя мы уже знаем, что это не правильно... возможно, это выглядит хорошо.
#This takes a while, maybe you can trust me with the result
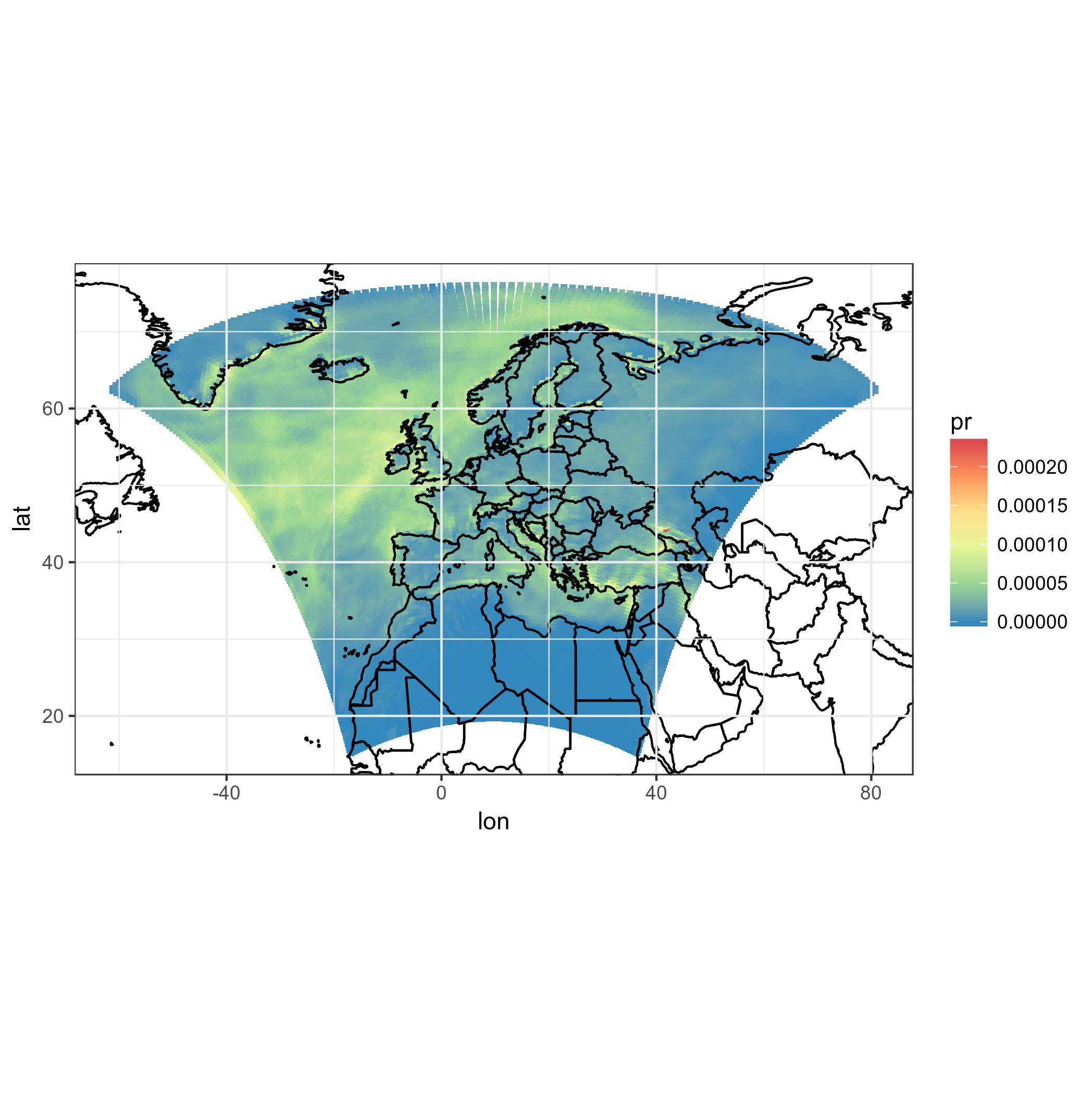
ggplot(pr_df, aes(y=lat, x=lon, fill=pr)) + geom_tile(width=1.5, height=1.5) +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_theme + my_fill +
coord_map('lambert', lat0=30, lat1=65, xlim=c(-20, 39), ylim=c(19, 75))
Прежде всего, это занимает много времени. Неприемлимо. Кроме того, опять же: это не правильные модели клеток.
Попробуйте с точками, а не с плитками
Может быть, мы можем использовать круглые или квадратные точки вместо плиток и проецировать их тоже!
#Basic 'unprojected' point plot
ggplot(pr_df, aes(y=lat, x=lon, color=pr)) + geom_point(size=2) +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_cols + my_theme +
coord_quickmap(xlim=range(pr_df$lon), ylim=range(pr_df$lat))
Мы можем использовать квадратные точки... и проектировать! Мы приближаемся, хотя знаем, что это все еще не правильно.
#In the following plot pointsize, xlim and ylim were manually set. Setting the wrong values leads to bad results.
#Also the lambert projection values were tired and guessed from the model CRS
ggplot(pr_df, aes(y=lat, x=lon, color=pr)) +
geom_point(size=2, shape=15) +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_theme + my_cols +
coord_map('lambert', lat0=30, lat1=65, xlim=c(-20, 39), ylim=c(19, 75))
Достойные результаты, но не полностью автоматические и построение точек не достаточно хорошо. Я хочу настоящие модели клеток, с их формой, видоизмененной проекцией!
Полигоны, может быть?
Итак, как вы можете видеть, я нахожусь после способа правильного построения прямоугольников модели в правильной форме и положении. Конечно, квадраты модели, которые являются квадратами в модели, после проецирования становятся формами, которые больше не являются регулярными. Так может я смогу использовать полигоны и спроецировать их? Я пытался использовать rasterToPolygons а также fortify и следуйте этому посту, но не смогли этого сделать. Я попробовал это:
pr2poly <- rasterToPolygons(pr)
#http://mazamascience.com/WorkingWithData/?p=1494
pr2poly@data$id <- rownames(pr2poly@data)
tmp <- fortify(pr2poly, region = "id")
tmp2 <- merge(tmp, pr2poly@data, by = "id")
ggplot(tmp2, aes(x=long, y=lat, group = group, fill=Total.precipitation.flux)) + geom_polygon() + my_fill
Хорошо, давайте попробуем заменить лат-лон...
tmp2$long <- lon[]
tmp2$lat <- lat[]
#Mh, does not work! See below:
ggplot(tmp2, aes(x=long, y=lat, group = group, fill=Total.precipitation.flux)) + geom_polygon() + my_fill
(извините, что я изменил цветовую гамму на графиках)
Мммм, даже не стоит пытаться с проекцией. Может быть, я должен попытаться вычислить широты углов модельных ячеек, создать для этого многоугольники и перепроектировать это?
Заключение
- Я хочу нанести данные проектируемой модели на ее собственную сетку, но я не смог этого сделать. Использование плиток некорректно, использование точек - хакерство, а использование полигонов, по-видимому, не работает по неизвестным причинам.
- При проецировании через
coord_map()линии сетки и метки осей неверны. Это делает спроектированные ggplots непригодными для публикаций.
2 ответа
После нескольких копаний кажется, что ваша модель основана на регулярной сетке 50 км в конической проекции Ламберта. Тем не менее, у вас есть координаты в netcdf - это латинские координаты WGS84 центра "ячеек".
Учитывая это, более простой подход состоит в том, чтобы восстановить ячейки в исходной проекции, а затем построить полигоны после преобразования в sf объект, в конце концов после репроекции. Нечто подобное должно работать (обратите внимание, что вам нужно будет установить devel версия ggplot2 от github для него на работу)
load(url('https://files.fm/down.php?i=kew5pxw7&n=loadme.Rdata'))
library(raster)
library(sf)
library(tidyverse)
library(maps)
devtools::install_github("hadley/ggplot2")
# ____________________________________________________________________________
# Transform original data to a SpatialPointsDataFrame in 4326 proj ####
coords = data.frame(lat = values(s[[2]]), lon = values(s[[3]]))
spPoints <- SpatialPointsDataFrame(coords,
data = data.frame(data = values(s[[1]])),
proj4string = CRS("+init=epsg:4326"))
# ____________________________________________________________________________
# Convert back the lat-lon coordinates of the points to the original ###
# projection of the model (lcc), then convert the points to polygons in lcc
# projection and convert to an `sf` object to facilitate plotting
orig_grid = spTransform(spPoints, projection(s))
polys = as(SpatialPixelsDataFrame(orig_grid, orig_grid@data, tolerance = 0.149842),"SpatialPolygonsDataFrame")
polys_sf = as(polys, "sf")
points_sf = as(orig_grid, "sf")
# ____________________________________________________________________________
# Plot using ggplot - note that now you can reproject on the fly to any ###
# projection using `coord_sf`
# Plot in original projection (note that in this case the cells are squared):
my_theme <- theme_bw() + theme(panel.ontop=TRUE, panel.background=element_blank())
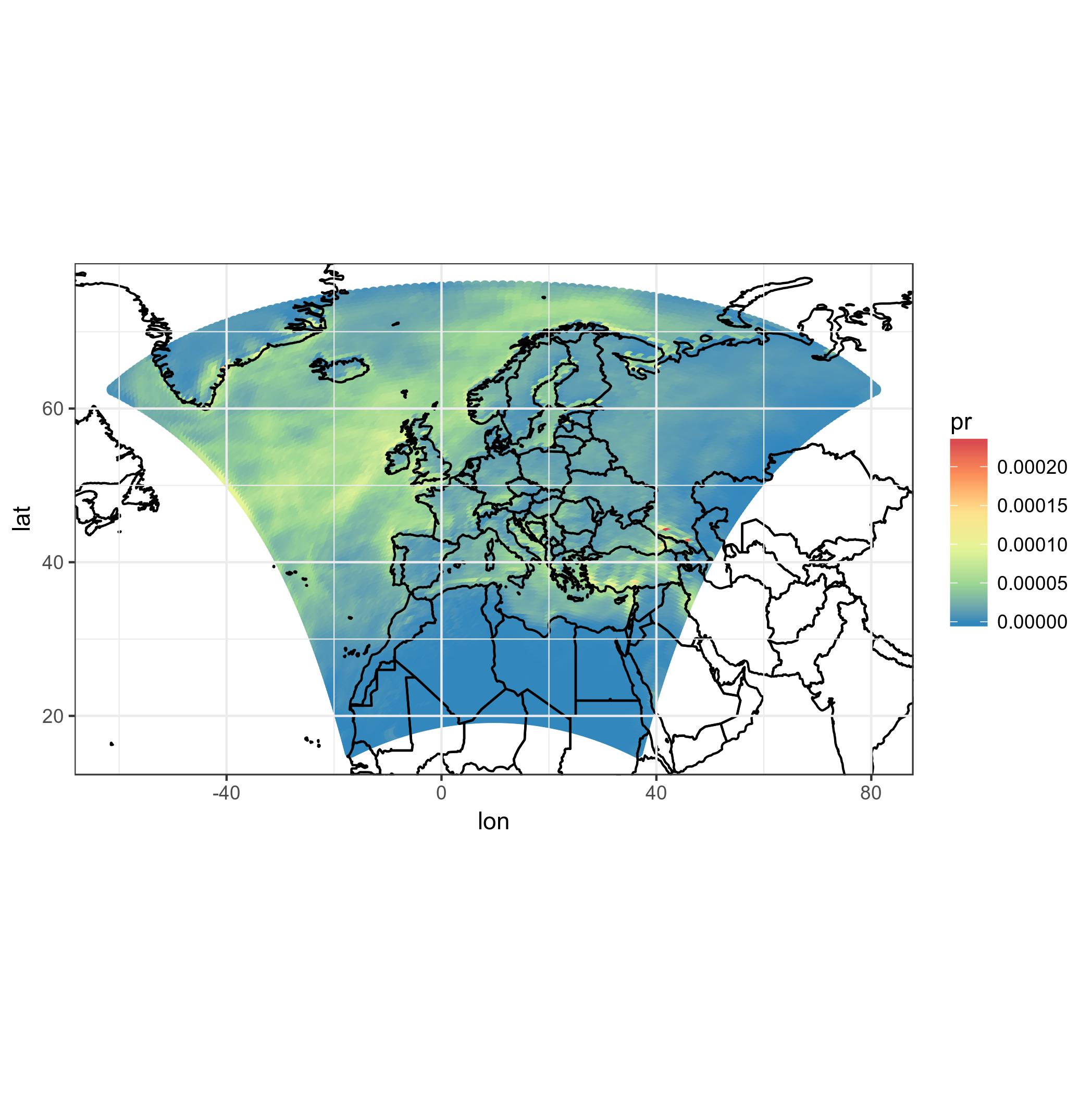
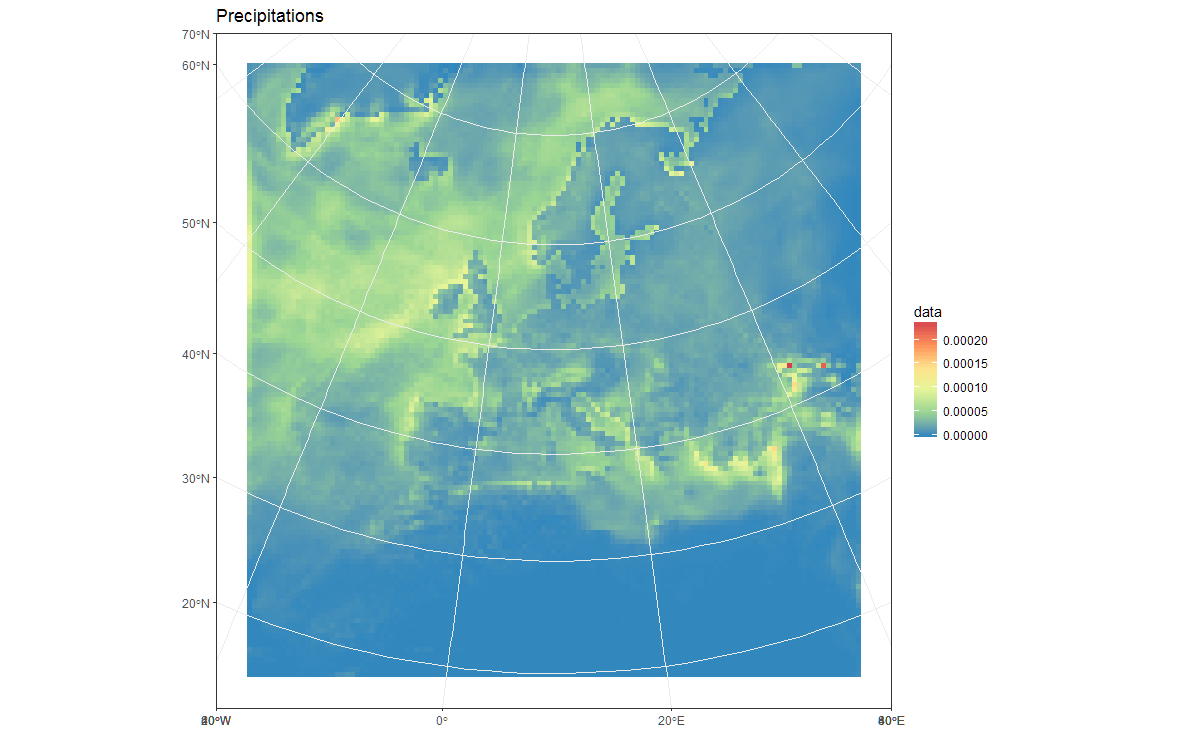
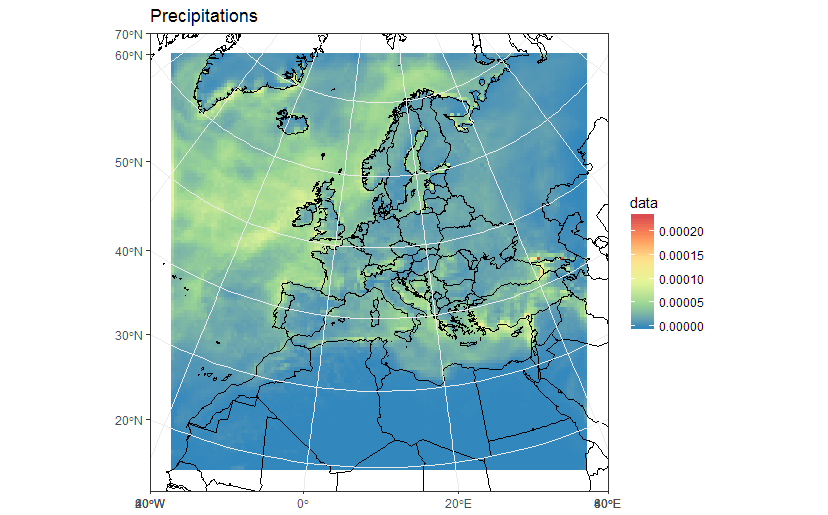
ggplot(polys_sf) +
geom_sf(aes(fill = data)) +
scale_fill_distiller(palette='Spectral') +
ggtitle("Precipitations") +
coord_sf() +
my_theme
# Now Plot in WGS84 latlon projection and add borders:
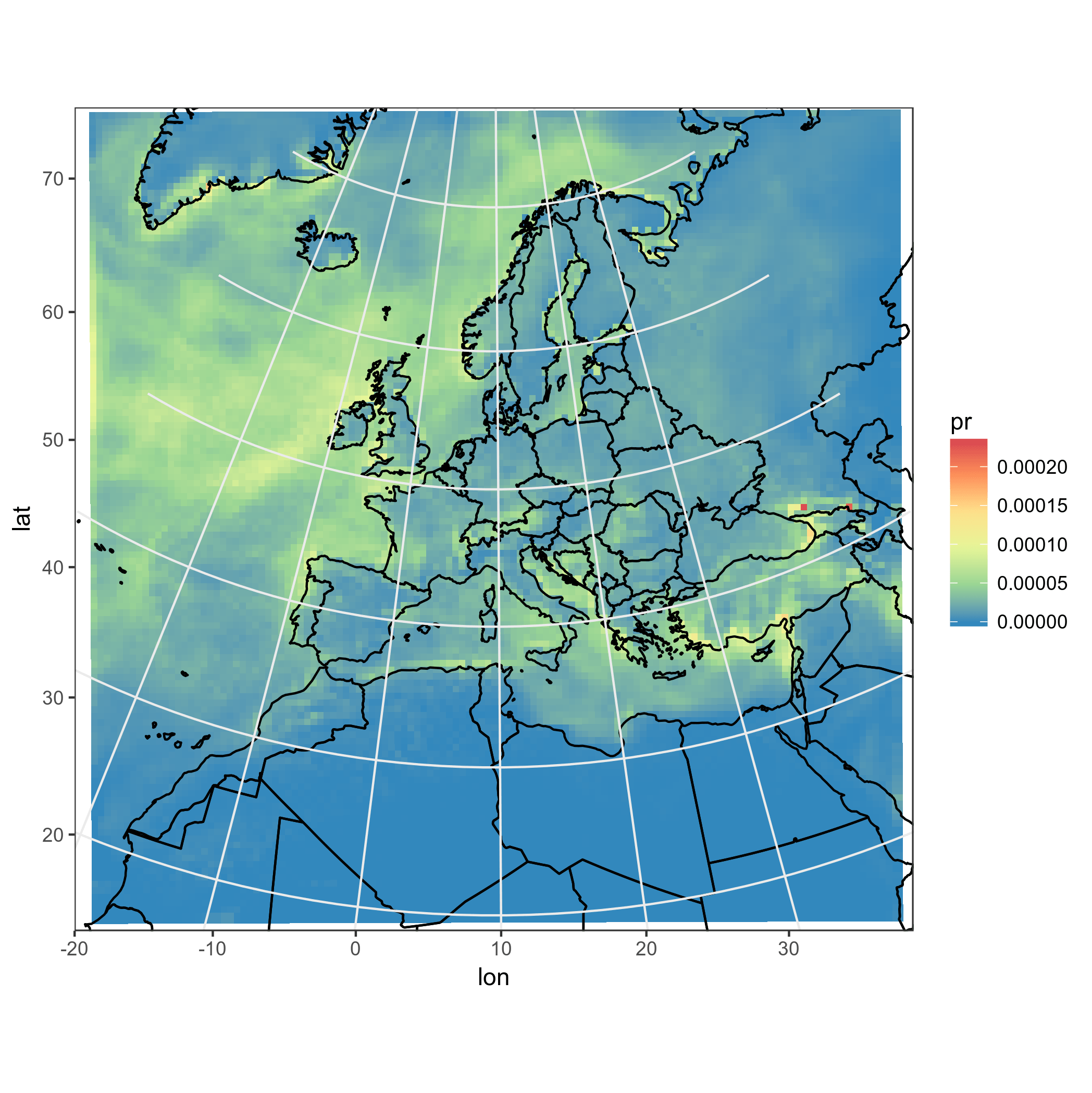
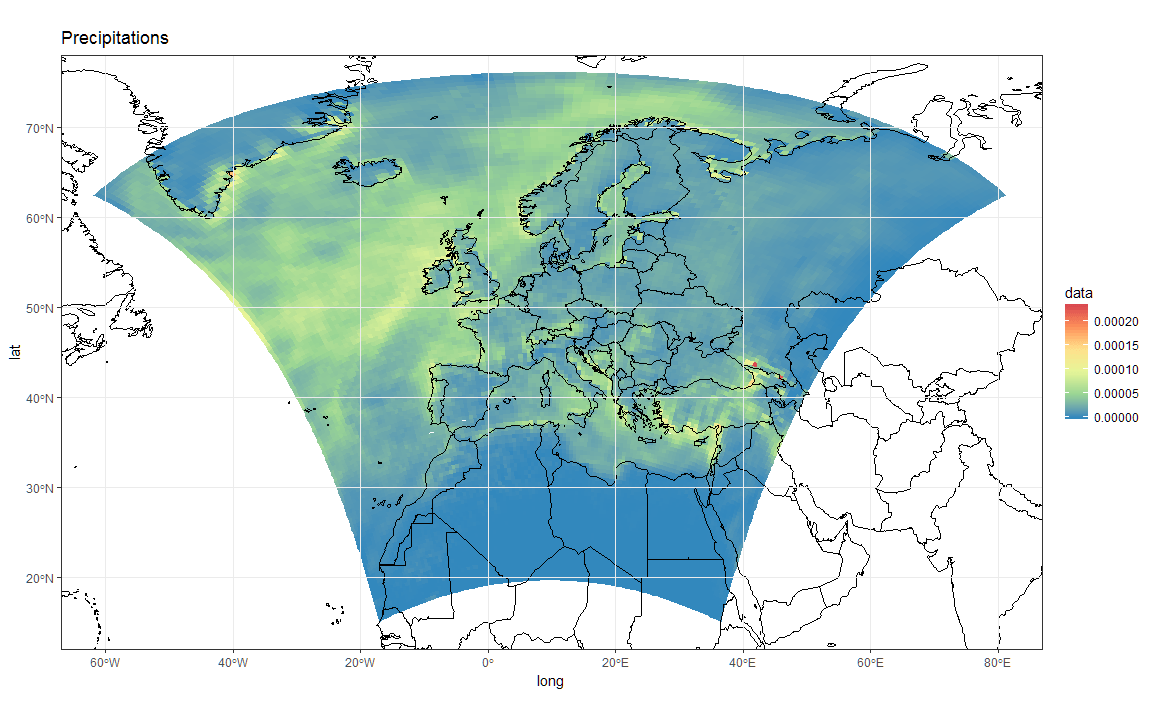
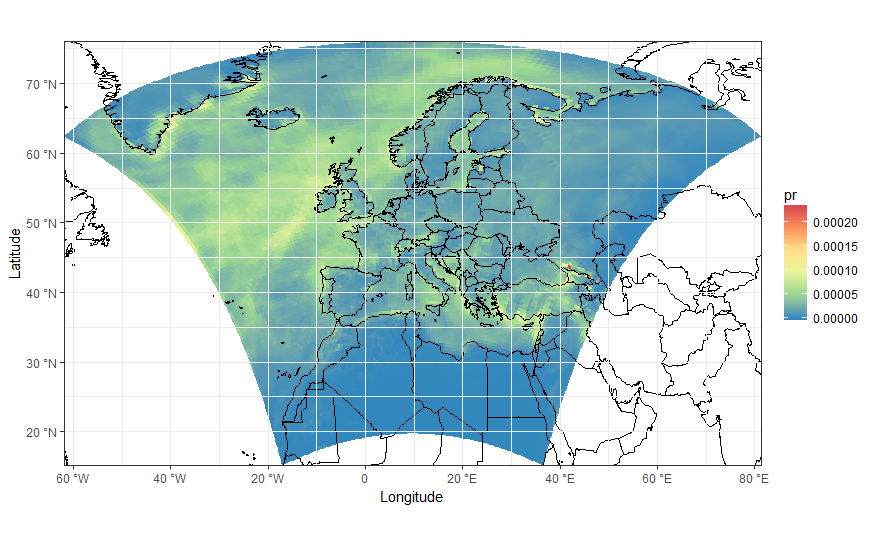
ggplot(polys_sf) +
geom_sf(aes(fill = data)) +
scale_fill_distiller(palette='Spectral') +
ggtitle("Precipitations") +
borders('world', colour='black')+
coord_sf(crs = st_crs(4326), xlim = c(-60, 80), ylim = c(15, 75))+
my_theme
Однако для добавления границ в исходную проекцию также необходимо нанести изображение в виде границы sf объект. Заимствование отсюда:
Преобразование объекта "карта" в объект "SpatialPolygon"
Примерно так будет работать:
library(maptools)
borders <- map("world", fill = T, plot = F)
IDs <- seq(1,1627,1)
borders <- map2SpatialPolygons(borders, IDs=borders$names,
proj4string=CRS("+proj=longlat +datum=WGS84")) %>%
as("sf")
ggplot(polys_sf) +
geom_sf(aes(fill = data), color = "transparent") +
geom_sf(data = borders, fill = "transparent", color = "black") +
scale_fill_distiller(palette='Spectral') +
ggtitle("Precipitations") +
coord_sf(crs = st_crs(projection(s)),
xlim = st_bbox(polys_sf)[c(1,3)],
ylim = st_bbox(polys_sf)[c(2,4)]) +
my_theme
Как примечание, теперь, когда мы "восстановили" правильную пространственную привязку, также возможно построить правильную raster набор данных. Например:
r <- s[[1]]
extent(r) <- extent(orig_grid) + 50000
даст вам правильное raster в r:
r
class : RasterLayer
band : 1 (of 36 bands)
dimensions : 125, 125, 15625 (nrow, ncol, ncell)
resolution : 50000, 50000 (x, y)
extent : -3150000, 3100000, -3150000, 3100000 (xmin, xmax, ymin, ymax)
coord. ref. : +proj=lcc +lat_1=30. +lat_2=65. +lat_0=48. +lon_0=9.75 +x_0=-25000. +y_0=-25000. +ellps=sphere +a=6371229. +b=6371229. +units=m +no_defs
data source : in memory
names : Total.precipitation.flux
values : 0, 0.0002373317 (min, max)
z-value : 1998-01-16 10:30:00
zvar : pr
Посмотрите, что теперь разрешение составляет 50 км, а экстент находится в метрических координатах. Вы могли бы таким образом построить / работать с r используя функции для raster данные, такие как:
library(rasterVis)
gplot(r) + geom_tile(aes(fill = value)) +
scale_fill_distiller(palette="Spectral", na.value = "transparent") +
my_theme
library(mapview)
mapview(r, legend = TRUE)
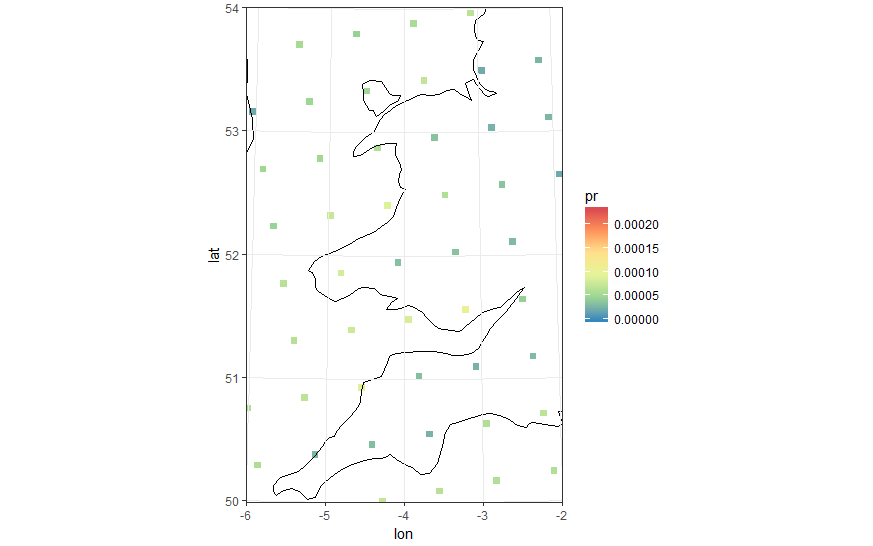
"Увеличение", чтобы увидеть точки, которые являются центрами клеток. Вы можете видеть, что они в прямоугольной сетке.
Я вычислил вершины многоугольников следующим образом.
Конвертировать 125x125 широт и долгот в матрицу
Инициализировать матрицу 126x126 для вершин ячеек (углов).
Рассчитайте вершины ячеек как среднее положение каждой группы точек 2x2.
Добавьте вершины ячеек для краев и углов (предположим, что ширина и высота ячейки равны ширине и высоте смежных ячеек).
Создайте data.frame с каждой ячейкой, имеющей четыре вершины, чтобы мы получили строки 4x125x125.
Код становится
pr <- s[[1]]
lon <- s[[2]]
lat <- s[[3]]
#Lets get the data into data.frames
#Gridded in model units:
#Projected points:
lat_m <- as.matrix(lat)
lon_m <- as.matrix(lon)
pr_m <- as.matrix(pr)
#Initialize emptry matrix for vertices
lat_mv <- matrix(,nrow = 126,ncol = 126)
lon_mv <- matrix(,nrow = 126,ncol = 126)
#Calculate centre of each set of (2x2) points to use as vertices
lat_mv[2:125,2:125] <- (lat_m[1:124,1:124] + lat_m[2:125,1:124] + lat_m[2:125,2:125] + lat_m[1:124,2:125])/4
lon_mv[2:125,2:125] <- (lon_m[1:124,1:124] + lon_m[2:125,1:124] + lon_m[2:125,2:125] + lon_m[1:124,2:125])/4
#Top edge
lat_mv[1,2:125] <- lat_mv[2,2:125] - (lat_mv[3,2:125] - lat_mv[2,2:125])
lon_mv[1,2:125] <- lon_mv[2,2:125] - (lon_mv[3,2:125] - lon_mv[2,2:125])
#Bottom Edge
lat_mv[126,2:125] <- lat_mv[125,2:125] + (lat_mv[125,2:125] - lat_mv[124,2:125])
lon_mv[126,2:125] <- lon_mv[125,2:125] + (lon_mv[125,2:125] - lon_mv[124,2:125])
#Left Edge
lat_mv[2:125,1] <- lat_mv[2:125,2] + (lat_mv[2:125,2] - lat_mv[2:125,3])
lon_mv[2:125,1] <- lon_mv[2:125,2] + (lon_mv[2:125,2] - lon_mv[2:125,3])
#Right Edge
lat_mv[2:125,126] <- lat_mv[2:125,125] + (lat_mv[2:125,125] - lat_mv[2:125,124])
lon_mv[2:125,126] <- lon_mv[2:125,125] + (lon_mv[2:125,125] - lon_mv[2:125,124])
#Corners
lat_mv[c(1,126),1] <- lat_mv[c(1,126),2] + (lat_mv[c(1,126),2] - lat_mv[c(1,126),3])
lon_mv[c(1,126),1] <- lon_mv[c(1,126),2] + (lon_mv[c(1,126),2] - lon_mv[c(1,126),3])
lat_mv[c(1,126),126] <- lat_mv[c(1,126),125] + (lat_mv[c(1,126),125] - lat_mv[c(1,126),124])
lon_mv[c(1,126),126] <- lon_mv[c(1,126),125] + (lon_mv[c(1,126),125] - lon_mv[c(1,126),124])
pr_df_orig <- data.frame(lat=lat[], lon=lon[], pr=pr[])
pr_df <- data.frame(lat=as.vector(lat_mv[1:125,1:125]), lon=as.vector(lon_mv[1:125,1:125]), pr=as.vector(pr_m))
pr_df$id <- row.names(pr_df)
pr_df <- rbind(pr_df,
data.frame(lat=as.vector(lat_mv[1:125,2:126]), lon=as.vector(lon_mv[1:125,2:126]), pr = pr_df$pr, id = pr_df$id),
data.frame(lat=as.vector(lat_mv[2:126,2:126]), lon=as.vector(lon_mv[2:126,2:126]), pr = pr_df$pr, id = pr_df$id),
data.frame(lat=as.vector(lat_mv[2:126,1:125]), lon=as.vector(lon_mv[2:126,1:125]), pr = pr_df$pr, id= pr_df$id))
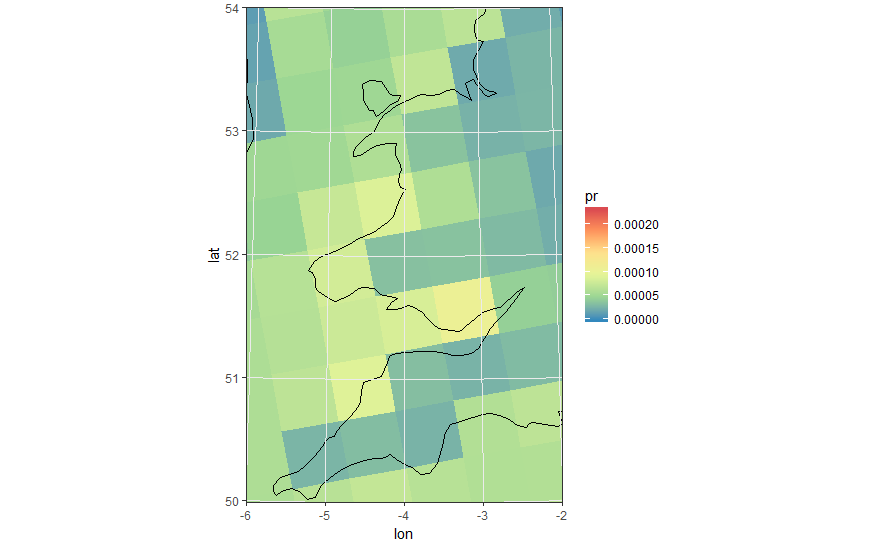
То же увеличенное изображение с многоугольниками
ewbrks <- seq(-180,180,20)
nsbrks <- seq(-90,90,10)
ewlbls <- unlist(lapply(ewbrks, function(x) ifelse(x < 0, paste(abs(x), "°W"), ifelse(x > 0, paste(abs(x), "°E"),x))))
nslbls <- unlist(lapply(nsbrks, function(x) ifelse(x < 0, paste(abs(x), "°S"), ifelse(x > 0, paste(abs(x), "°N"),x))))
Замена geom_tile & geom_point на geom_polygon
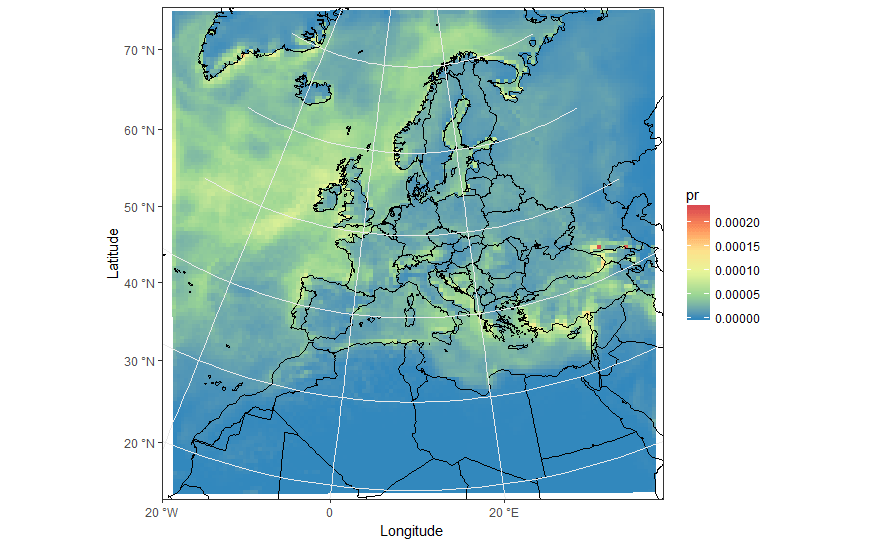
ggplot(pr_df, aes(y=lat, x=lon, fill=pr, group = id)) + geom_polygon() +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_theme + my_fill +
coord_quickmap(xlim=range(pr_df$lon), ylim=range(pr_df$lat)) +
scale_x_continuous(breaks = ewbrks, labels = ewlbls, expand = c(0, 0)) +
scale_y_continuous(breaks = nsbrks, labels = nslbls, expand = c(0, 0)) +
labs(x = "Longitude", y = "Latitude")
ggplot(pr_df, aes(y=lat, x=lon, fill=pr, group = id)) + geom_polygon() +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_theme + my_fill +
coord_map('lambert', lat0=30, lat1=65, xlim=c(-20, 39), ylim=c(19, 75)) +
scale_x_continuous(breaks = ewbrks, labels = ewlbls, expand = c(0, 0)) +
scale_y_continuous(breaks = nsbrks, labels = nslbls, expand = c(0, 0)) +
labs(x = "Longitude", y = "Latitude")
Редактировать - обойти тики оси
Я не смог найти быстрого решения для линий сетки и меток для широты. Возможно, где-то есть пакет R, который решит вашу проблему с гораздо меньшим количеством кода!
Ручная установка необходимых nsbreaks и создание data.frame
ewbrks <- seq(-180,180,20)
nsbrks <- c(20,30,40,50,60,70)
nsbrks_posn <- c(-16,-17,-16,-15,-14.5,-13)
ewlbls <- unlist(lapply(ewbrks, function(x) ifelse(x < 0, paste0(abs(x), "° W"), ifelse(x > 0, paste0(abs(x), "° E"),x))))
nslbls <- unlist(lapply(nsbrks, function(x) ifelse(x < 0, paste0(abs(x), "° S"), ifelse(x > 0, paste0(abs(x), "° N"),x))))
latsdf <- data.frame(lon = rep(c(-100,100),length(nsbrks)), lat = rep(nsbrks, each =2), label = rep(nslbls, each =2), posn = rep(nsbrks_posn, each =2))
Удалите метки оси Y и соответствующие линии сетки, а затем добавьте обратно "вручную", используя geom_line а также geom_text
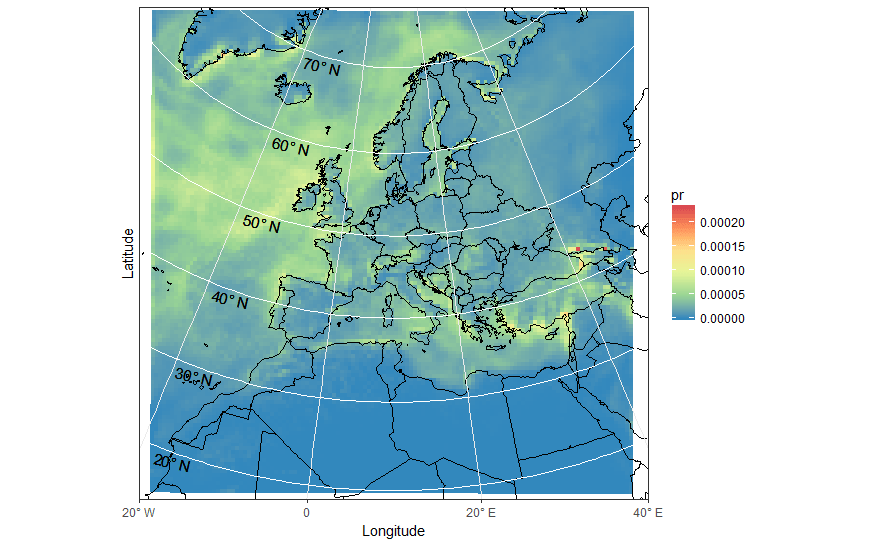
ggplot(pr_df, aes(y=lat, x=lon, fill=pr, group = id)) + geom_polygon() +
borders('world', xlim=range(pr_df$lon), ylim=range(pr_df$lat), colour='black') + my_theme + my_fill +
coord_map('lambert', lat0=30, lat1=65, xlim=c(-20, 40), ylim=c(19, 75)) +
scale_x_continuous(breaks = ewbrks, labels = ewlbls, expand = c(0, 0)) +
scale_y_continuous(expand = c(0, 0), breaks = NULL) +
geom_line(data = latsdf, aes(x=lon, y=lat, group = lat), colour = "white", size = 0.5, inherit.aes = FALSE) +
geom_text(data = latsdf, aes(x = posn, y = (lat-1), label = label), angle = -13, size = 4, inherit.aes = FALSE) +
labs(x = "Longitude", y = "Latitude") +
theme( axis.text.y=element_blank(),axis.ticks.y=element_blank())