R - изображение пиксельной матрицы?
Как бы вы сделали изображение из матрицы в R?
Значения матрицы соответствуют интенсивности пикселей на изображении (хотя меня сейчас интересуют только значения 0,1 белого или черного цвета), а номера столбцов и строк соответствуют вертикальному и горизонтальному расположению на изображении.
Под созданием изображения я подразумеваю его отображение на экране и сохранение в формате jpg.
7 ответов
Вы можете отобразить его на экране проще всего, используя "изображение":
m = matrix(runif(100),10,10)
par(mar=c(0, 0, 0, 0))
image(m, useRaster=TRUE, axes=FALSE)
Вы также можете взглянуть на растровый пакет...
Настройте сюжет без полей:
par(mar = rep(0, 4))
Представьте матрицу в оттенках серого, как в ответе spacedman, но полностью заполнив устройство:
m = matrix(runif(100),10,10)
image(m, axes = FALSE, col = grey(seq(0, 1, length = 256)))
Оберните это в вызов png() для создания файла:
png("simpleIm.png")
par(mar = rep(0, 4))
image(m, axes = FALSE, col = grey(seq(0, 1, length = 256)))
dev.off()
Если вам нужно сделать это с пространственными осями (по умолчанию [0,1] для X и Y), используйте image.default(x, y, z, ...) Форма, где х и у дают центральное положение пикселей в z. x а также y может иметь длину dim(z) + 1, чтобы дать угловые координаты для этого соглашения.
Центры пикселей (это по умолчанию для изображения):
x <- seq(0, 1, length = nrow(m))
y <- seq(0, 1, length = ncol(m))
image(x, y, m, col = grey(seq(0, 1, length = 256)))
Углы пикселей (нужно 1 лишние x и y, а 0 теперь находится в самом нижнем левом углу):
x <- seq(0, 1, length = nrow(m) + 1)
y <- seq(0, 1, length = ncol(m) + 1)
image(x, y, m, col = grey(seq(0, 1, length = 256)))
Обратите внимание, что из R 2.13 image.default получает аргумент useRaster который использует очень эффективную функцию новой графики rasterImage а не старый image который эффективно несколько звонков rect под капотом нарисовать каждый пиксель в виде многоугольника.
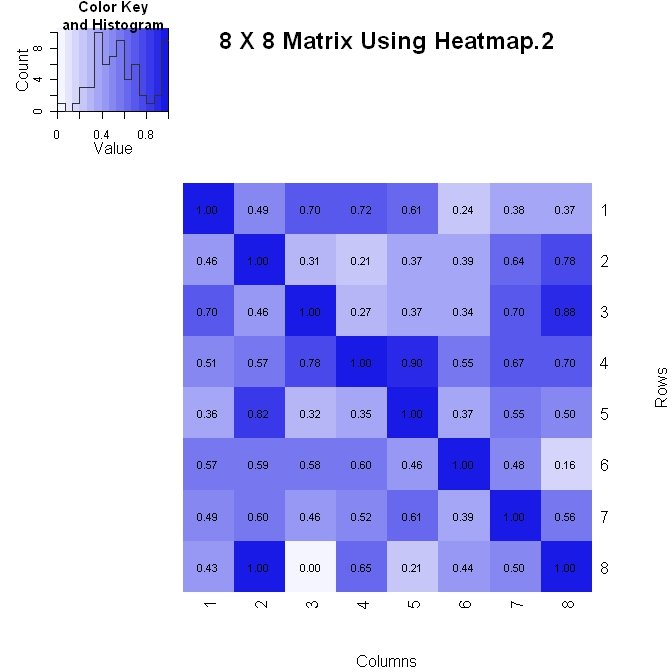
Я делаю матрицу (где вертикальная ось увеличивается при спуске) одним из двух способов. Ниже приведен первый способ с использованием heatmap.2(). Он имеет больший контроль над тем, как числовые значения форматируются на графике (см. Инструкцию formatC ниже), но с ним немного сложнее справиться при изменении макета.
library(gplots)
#Build the matrix data to look like a correlation matrix
x <- matrix(rnorm(64), nrow=8)
x <- (x - min(x))/(max(x) - min(x)) #Scale the data to be between 0 and 1
for (i in 1:8) x[i, i] <- 1.0 #Make the diagonal all 1's
#Format the data for the plot
xval <- formatC(x, format="f", digits=2)
pal <- colorRampPalette(c(rgb(0.96,0.96,1), rgb(0.1,0.1,0.9)), space = "rgb")
#Plot the matrix
x_hm <- heatmap.2(x, Rowv=FALSE, Colv=FALSE, dendrogram="none", main="8 X 8 Matrix Using Heatmap.2", xlab="Columns", ylab="Rows", col=pal, tracecol="#303030", trace="none", cellnote=xval, notecol="black", notecex=0.8, keysize = 1.5, margins=c(5, 5))
Вы можете создать тепловую карту матрицы.
library(pheatmap)
# Create a 10x10 matrix of random numbers
m = matrix(runif(100), 10, 10)
# Save output to jpeg
jpeg("heatmap.jpg")
pheatmap(m, cluster_row = FALSE, cluster_col = FALSE, color=gray.colors(2,start=1,end=0))
dev.off()
Увидеть ?pheatmap для большего количества вариантов.
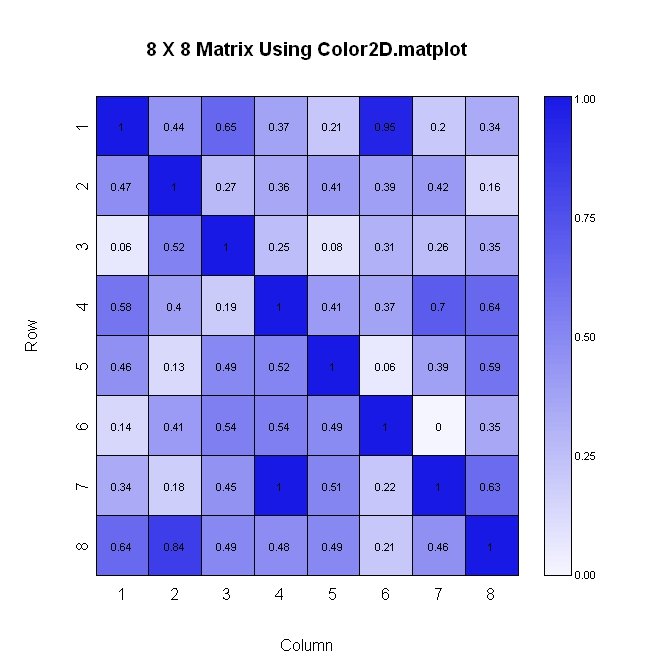
Вот второй способ (опять же, где вертикальная ось увеличивается, спускаясь вниз). Этот метод проще для разметки, но он имеет меньший контроль над форматом числовых значений, отображаемых на графике.
library(plotrix)
#Build the matrix data to look like a correlation matrix
n <- 8
x <- matrix(runif(n*n), nrow=n)
xmin <- 0
xmax <- 1
for (i in 1:n) x[i, i] <- 1.0 #Make the diagonal all 1's
#Generate the palette for the matrix and the legend. Generate labels for the legend
palmat <- color.scale(x, c(1, 0.4), c(1, 0.4), c(0.96, 1))
palleg <- color.gradient(c(1, 0.4), c(1, 0.4), c(0.96, 1), nslices=100)
lableg <- c(formatC(xmin, format="f", digits=2), formatC(1*(xmax-xmin)/4, format="f", digits=2), formatC(2*(xmax-xmin)/4, format="f", digits=2), formatC(3*(xmax-xmin)/4, format="f", digits=2), formatC(xmax, format="f", digits=2))
#Set up the plot area and plot the matrix
par(mar=c(5, 5, 5, 8))
color2D.matplot(x, cellcolors=palmat, main=paste(n, " X ", n, " Matrix Using Color2D.matplot", sep=""), show.values=2, vcol=rgb(0,0,0), axes=FALSE, vcex=0.7)
axis(1, at=seq(1, n, 1)-0.5, labels=seq(1, n, 1), tck=-0.01, padj=-1)
#In the axis() statement below, note that the labels are decreasing. This is because
#the above color2D.matplot() statement has "axes=FALSE" and a normal axis()
#statement was used.
axis(2, at=seq(1, n, 1)-0.5, labels=seq(n, 1, -1), tck=-0.01, padj=0.7)
#Plot the legend
pardat <- par()
color.legend(pardat$usr[2]+0.5, 0, pardat$usr[2]+1, pardat$usr[2], paste(" ", lableg, sep=""), palleg, align="rb", gradient="y", cex=0.7)
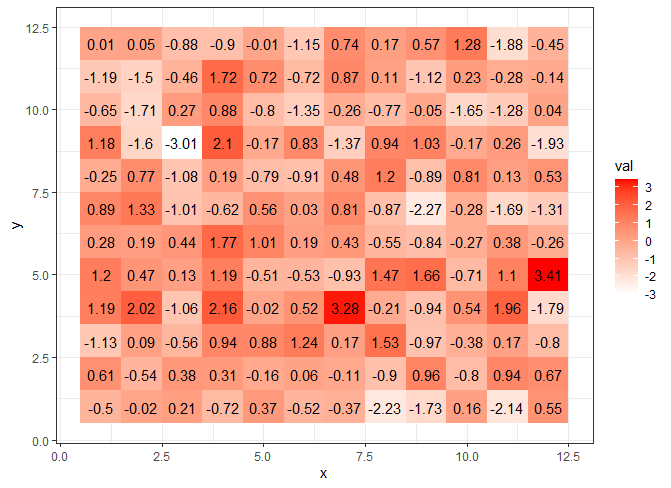
С
ggplot2:
library(tidyverse)
n <- 12
m <- matrix(rnorm(n*n),n,n)
rownames(m) <- colnames(m) <- 1:n
df <- as.data.frame(m) %>% gather(key='y', value='val')
df$y <- as.integer(df$y)
df$x <- rep(1:n, n)
ggplot(df, aes(x, y, fill= val)) +
geom_tile() +
geom_text(aes(x, y, label=round(val,2))) +
scale_fill_gradient(low = "white", high = "red") +
theme_bw()