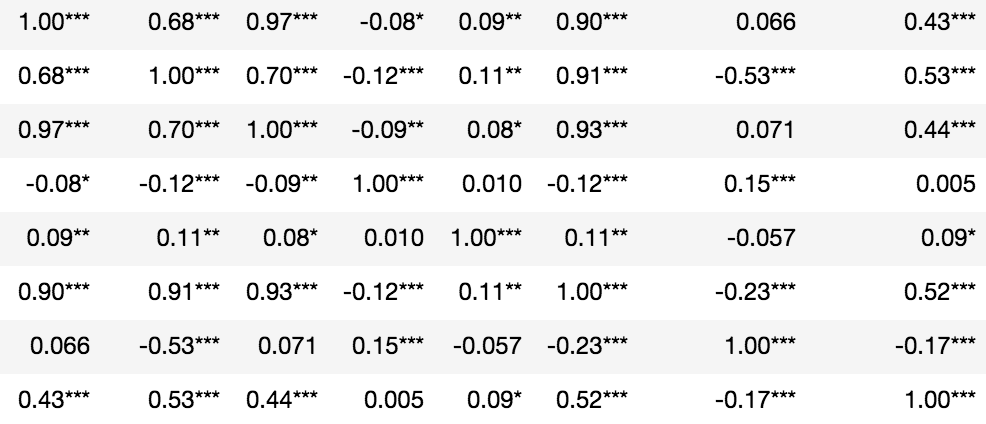
Соотношение колонн панд со статистической значимостью
Каков наилучший способ, учитывая df для pandas, чтобы получить корреляцию между его столбцами df.1 а также df.2?
Я не хочу, чтобы результат подсчитывал строки с NaN, который pandas встроенная корреляция делает. Но я также хочу вывести pvalue или стандартная ошибка, которой нет у встроенной.
SciPy Кажется, что они попали в руки NaN, хотя я полагаю, что это действительно имеет значение.
Пример данных:
1 2
0 2 NaN
1 NaN 1
2 1 2
3 -4 3
4 1.3 1
5 NaN NaN
7 ответов
Ответ, предоставленный @Shashank, хорош. Однако, если вы хотите решение в чистом виде pandasВам может понравиться это:
import pandas as pd
from pandas.io.data import DataReader
from datetime import datetime
import scipy.stats as stats
gdp = pd.DataFrame(DataReader("GDP", "fred", start=datetime(1990, 1, 1)))
vix = pd.DataFrame(DataReader("VIXCLS", "fred", start=datetime(1990, 1, 1)))
#Do it with a pandas regression to get the p value from the F-test
df = gdp.merge(vix,left_index=True, right_index=True, how='left')
vix_on_gdp = pd.ols(y=df['VIXCLS'], x=df['GDP'], intercept=True)
print(df['VIXCLS'].corr(df['GDP']), vix_on_gdp.f_stat['p-value'])
Результаты:
-0.0422917932738 0.851762475093
Те же результаты, что и у функции stats:
#Do it with stats functions.
df_clean = df.dropna()
stats.pearsonr(df_clean['VIXCLS'], df_clean['GDP'])
Результаты:
(-0.042291793273791969, 0.85176247509284908)
Для расширения возможностей я предлагаю вам подход, основанный на уродливых циклах:
#Add a third field
oil = pd.DataFrame(DataReader("DCOILWTICO", "fred", start=datetime(1990, 1, 1)))
df = df.merge(oil,left_index=True, right_index=True, how='left')
#construct two arrays, one of the correlation and the other of the p-vals
rho = df.corr()
pval = np.zeros([df.shape[1],df.shape[1]])
for i in range(df.shape[1]): # rows are the number of rows in the matrix.
for j in range(df.shape[1]):
JonI = pd.ols(y=df.icol(i), x=df.icol(j), intercept=True)
pval[i,j] = JonI.f_stat['p-value']
Результаты rho:
GDP VIXCLS DCOILWTICO
GDP 1.000000 -0.042292 0.870251
VIXCLS -0.042292 1.000000 -0.004612
DCOILWTICO 0.870251 -0.004612 1.000000
Результаты pval:
[[ 0.00000000e+00 8.51762475e-01 1.11022302e-16]
[ 8.51762475e-01 0.00000000e+00 9.83747425e-01]
[ 1.11022302e-16 9.83747425e-01 0.00000000e+00]]
Чтобы рассчитать все p-значения одновременно, вы можете использовать ниже calculate_pvalues функция:
df = pd.DataFrame({'A':[1,2,3], 'B':[2,5,3], 'C':[5,2,1], 'D':['text',2,3] })
calculate_pvalues(df)
Выход похож на
corr()(но с p-значениями):A B C A 0 0.7877 0.1789 B 0.7877 0 0.6088 C 0.1789 0.6088 0p-значения округлены до 4 десятичных знаков
- Столбец D игнорируется, так как содержит текст.
Ниже приведен код функции:
from scipy.stats import pearsonr
import pandas as pd
def calculate_pvalues(df):
df = df.dropna()._get_numeric_data()
dfcols = pd.DataFrame(columns=df.columns)
pvalues = dfcols.transpose().join(dfcols, how='outer')
for r in df.columns:
for c in df.columns:
pvalues[r][c] = round(pearsonr(df[r], df[c])[1], 4)
return pvalues
rho = df.corr()
rho = rho.round(2)
pval = calculate_pvalues(df) # toto_tico's answer
# create three masks
r1 = rho.applymap(lambda x: '{}*'.format(x))
r2 = rho.applymap(lambda x: '{}**'.format(x))
r3 = rho.applymap(lambda x: '{}***'.format(x))
# apply them where appropriate
rho = rho.mask(pval<=0.1,r1)
rho = rho.mask(pval<=0.05,r2)
rho = rho.mask(pval<=0.01,r3)
rho
# note I prefer readability over the conciseness of code,
# instead of six lines it could have been a single liner like this:
# [rho.mask(pval<=p,rho.applymap(lambda x: '{}*'.format(x)),inplace=True) for p in [.1,.05,.01]]
Вы можете использовать функции корреляции scipy.stats для получения значения p.
Например, если вы ищете корреляцию, такую как корреляция Пирсона, вы можете использовать функцию Pearsonr.
from scipy.stats import pearsonr
pearsonr([1, 2, 3], [4, 3, 7])
Дает вывод
(0.7205766921228921, 0.48775429164459994)
Где первое значение в кортеже - это значение корреляции, а второе - это p-значение.
В вашем случае вы можете использовать панд dropna функция для удаления NaN значения в первую очередь.
df_clean = df[['column1', 'column2']].dropna()
pearsonr(df_clean['column1'], df_clean['column2'])
В пандах v0.24.0 а method аргумент был добавлен к corr, Теперь вы можете сделать:
import pandas as pd
import numpy as np
from scipy.stats import pearsonr
df = pd.DataFrame({'A':[1,2,3], 'B':[2,5,3], 'C':[5,2,1]})
df.corr(method=lambda x, y: pearsonr(x, y)[1]) - np.eye(len(df.columns))
A B C
A 0.000000 0.787704 0.178912
B 0.787704 0.000000 0.608792
C 0.178912 0.608792 0.000000
Пожалуйста, обратите внимание на обходной путь с np.eye(len(df.columns)) что необходимо, потому что самокорреляции всегда установлены в 1.0 (см. https://github.com/pandas-dev/pandas/issues/25726).
Это был очень полезный код от oztalha. Я просто изменил форматирование (округленное до 2 цифр) везде, где r не было значимым.
rho = data.corr()
pval = calculate_pvalues(data) # toto_tico's answer
# create three masks
r1 = rho.applymap(lambda x: '{:.2f}*'.format(x))
r2 = rho.applymap(lambda x: '{:.2f}**'.format(x))
r3 = rho.applymap(lambda x: '{:.2f}***'.format(x))
r4 = rho.applymap(lambda x: '{:.2f}'.format(x))
# apply them where appropriate --this could be a single liner
rho = rho.mask(pval>0.1,r4)
rho = rho.mask(pval<=0.1,r1)
rho = rho.mask(pval<=0.05,r2)
rho = rho.mask(pval<=0.01,r3)
rho
Я попытался суммировать логику в функции, возможно, это не самый эффективный подход, но он предоставит вам вывод, аналогичный pandas df.corr(). Чтобы использовать это, просто поместите следующую функцию в ваш код и вызовите ее, предоставив объект dataframe, т.е. corr_pvalue (your_dataframe).
Я округлил значения до 4 знаков после запятой, если вы хотите получить другой вывод, измените значение в функции округления.
from scipy.stats import pearsonr
import numpy as np
import pandas as pd
def corr_pvalue(df):
numeric_df = df.dropna()._get_numeric_data()
cols = numeric_df.columns
mat = numeric_df.values
arr = np.zeros((len(cols),len(cols)), dtype=object)
for xi, x in enumerate(mat.T):
for yi, y in enumerate(mat.T[xi:]):
arr[xi, yi+xi] = map(lambda _: round(_,4), pearsonr(x,y))
arr[yi+xi, xi] = arr[xi, yi+xi]
return pd.DataFrame(arr, index=cols, columns=cols)
Я проверил это с пандами v0.18.1
Отличные ответы от @toto_tico и @Somendra-joshi. Тем не менее, он сбрасывает ненужные значения NA. В этом фрагменте я просто отбрасываю NA, которые относятся к вычисляемой в данный момент корреляции. В реальной реализации corr они делают то же самое.
def calculate_pvalues(df):
df = df._get_numeric_data()
dfcols = pd.DataFrame(columns=df.columns)
pvalues = dfcols.transpose().join(dfcols, how='outer')
for r in df.columns:
for c in df.columns:
if c == r:
df_corr = df[[r]].dropna()
else:
df_corr = df[[r,c]].dropna()
pvalues[r][c] = pearsonr(df_corr[r], df_corr[c])[1]
return pvalues
В одной строке кода с использованием списка:
>>> import pandas as pd
>>> from scipy.stats import pearsonr
>>> data = {'y':[0, 3, 2, 4, 3, 5, 4, 6, 5, 7, 6],
... 'x1':[0, 4, 2, 6, 2, 8, 6, 10, 4, 13, 5],
... 'x2':[0.0, 1.3, 0.2, 4.5, 1.3, 1.6, 3.5, 1.7, 1.6, 3.7, 1.7]}
>>> df = pd.DataFrame(data)
>>> pvals = pd.DataFrame([[pearsonr(df[c], df[y])[1] for y in df.columns] for c in df.columns],
... columns=df.columns, index=df.columns)
>>> pvals
y x1 x2
y 0.000000 0.000732 0.069996
x1 0.000732 0.000000 0.036153
x2 0.069996 0.036153 0.000000
>>>
Я был бы заинтересован в ловкой технике, которая могла бы объединить вышеуказанный фрейм данных с этим:
>>> df.corr()
y x1 x2
y 1.000000 0.857786 0.565208
x1 0.857786 1.000000 0.634093
x2 0.565208 0.634093 1.000000
Желаемый результат:
y x1 x2
y c 1.000000 0.857786 0.565208
p (0.0000) (0.0007) (0.0699)
x1 c 0.857786 1.000000 0.634093
p (0.0007) (0.0000) (0.0361)
x2 c 0.565208 0.634093 1.000000
p (0.0699) (0.0361) (0.0000)
Где есть мультииндекс и
c строки - коэффициент корреляции, а
p строки предоставляют значение pvalue.
Кто-нибудь думает?