Почему я получил тот же график?
Я использую networkx в ipython для анализа своего графа или сети, когда я сгенерировал максимальное связующее дерево и минимальное связующее дерево, я получил очень странный результат, что эти два графа одинаковы! Это мой код ниже:
a=nx.maximum_spanning_tree(pearson_net)
b=nx.minimum_spanning_tree(pearson_net)
pearson_net - это моя оригинальная сеть (граф), я хочу получить ребра этих двух графов, но эти ребра абсолютно одинаковы!
a.edges()
Это ребра графа а:
EdgeView([('600000.SH', '600015.SH'), ('600000.SH', '600016.SH'),
('600000.SH', '600030.SH'), ('600000.SH', '600036.SH'),
('600000.SH','600109.SH'), ('600000.SH', '600816.SH'),
('600000.SH','600837.SH'), ('600000.SH', '600999.SH'),
('600000.SH', '601009.SH'), ('600000.SH', '601099.SH'),
('600000.SH', '601166.SH'), ('600000.SH', '601288.SH'),
('600000.SH', '601318.SH'), ('600000.SH', '601328.SH'),
('600000.SH', '601336.SH'), ('600000.SH', '601377.SH'),
('600000.SH', '601398.SH'), ('600000.SH', '601555.SH'),
('600000.SH', '601601.SH'), ('600000.SH', '601628.SH'),
('600000.SH', '601688.SH'), ('600000.SH', '601788.SH'),
('600000.SH', '601818.SH'), ('600000.SH', '601939.SH'),
('600000.SH', '601988.SH'), ('600000.SH', '601998.SH'),
('600000.SH', '000001.SZ'), ('600000.SH', '000686.SZ'),
('600000.SH', '000728.SZ'), ('600000.SH', '000750.SZ'),
('600000.SH', '000776.SZ'), ('600000.SH', '000783.SZ'),
('600000.SH', '002142.SZ'), ('600000.SH', '002500.SZ'),
('600000.SH', '002673.SZ')])
а потом
b.edges()
Это ребра графа b:
EdgeView([('600000.SH', '600015.SH'), ('600000.SH', '600016.SH'),
('600000.SH', '600030.SH'), ('600000.SH', '600036.SH'),
('600000.SH','600109.SH'), ('600000.SH', '600816.SH'),
('600000.SH','600837.SH'), ('600000.SH', '600999.SH'),
('600000.SH', '601009.SH'), ('600000.SH', '601099.SH'),
('600000.SH', '601166.SH'), ('600000.SH', '601288.SH'),
('600000.SH', '601318.SH'), ('600000.SH', '601328.SH'),
('600000.SH', '601336.SH'), ('600000.SH', '601377.SH'),
('600000.SH', '601398.SH'), ('600000.SH', '601555.SH'),
('600000.SH', '601601.SH'), ('600000.SH', '601628.SH'),
('600000.SH', '601688.SH'), ('600000.SH', '601788.SH'),
('600000.SH', '601818.SH'), ('600000.SH', '601939.SH'),
('600000.SH', '601988.SH'), ('600000.SH', '601998.SH'),
('600000.SH', '000001.SZ'), ('600000.SH', '000686.SZ'),
('600000.SH', '000728.SZ'), ('600000.SH', '000750.SZ'),
('600000.SH', '000776.SZ'), ('600000.SH', '000783.SZ'),
('600000.SH', '002142.SZ'), ('600000.SH', '002500.SZ'),
('600000.SH', '002673.SZ')])
Я не могу понять этот результат. Почему Maximum_spanning_tree - это то же самое, что и imum_spanning_tree?
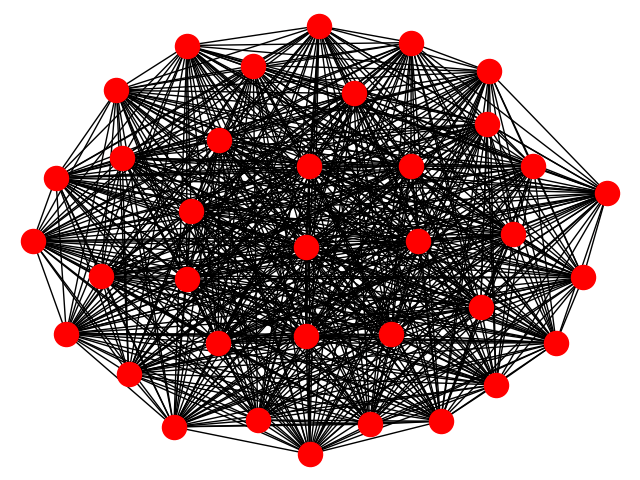
Это полный граф, один узел может быть связан с любым другим узлом. Это часть набора данных pearson_net'dnet: 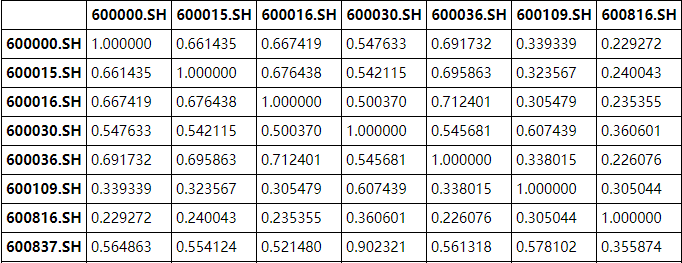 Столбцы и индекс - это узел графа, число (коэффициент, коррелированный по Пирсону) - это вес ребра.
Столбцы и индекс - это узел графа, число (коэффициент, коррелированный по Пирсону) - это вес ребра.
Это мой полный код:
pearson_net=nx.Graph()
for i in range(pearson):
for j in range(i+1,pearson):
pearson_net.add_edge(pearson.index[i],pearson.columns[j],......
weights=pearson.iloc[i][j])
tree1=nx.minimum_spanning_tree(pearson_net)
tree2=nx.maximum_spanning_tree(pearson_net)
"Пирсон" - это матрица коррелированного коэффициента, который ранее представлял собой набор данных.
1 ответ
Тестирование минимального и максимального связующего дерева
Нам нужно использовать минимальный пример для контроля результатов minimum_spanning_tree() а также maximum_spanning_tree() функции:
a_mat = [
[1.,0.661435,0.667419,0.547633],
[0.661435,1.,0.676438,0.542115],
[0.667419,0.676438,1.,0.500370],
[0.547633,0.542115,0.500370,1.]
]
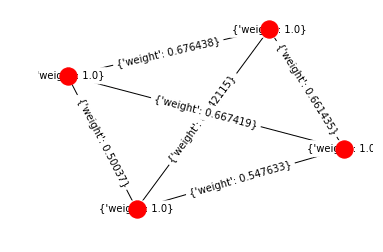
G = nx.from_numpy_matrix(np.array(a_mat))
pos = nx.spring_layout(G)
nx.draw_networkx_nodes(G,pos=pos)
nx.draw_networkx_edges(G,pos=pos)
nx.draw_networkx_edge_labels(G, pos=pos)
plt.axis('off')
plt.show()
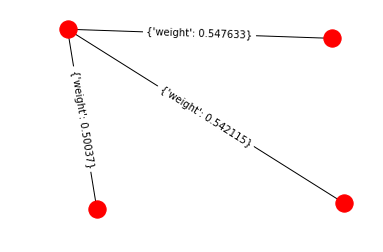
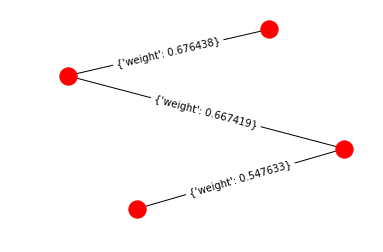
Из этого примера мы можем легко найти минимальное остовное дерево, добавив веса самых нижних ребер (0.50037, 0.547633, 0.542115)
В самом деле:
mi = nx.minimum_spanning_tree(G)
mi.edges(data=True)
[Из]:
EdgeDataView([(0, 3, {'weight': 0.547633}), (1, 3, {'weight': 0.542115}), (2, 3, {'weight': 0.50037})])
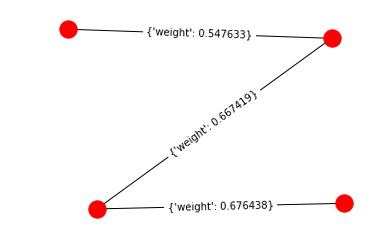
Для максимального остовного дерева мы можем предсказать из графика максимальную сумму весов ребер (0.661435, 0.667419,0.547633):
ma = nx.maximum_spanning_tree(G)
ma.edges(data=True)
[Из]:
EdgeDataView([(0, 2, {'weight': 0.667419}), (0, 3, {'weight': 0.547633}), (1, 2, {'weight': 0.676438})])
Из этого простого примера мы можем наблюдать, что две функции ведут себя как ожидалось.
Если вы покажете нам свой код, мы сможем найти ошибку для вас.
[Редактировать] Построение графика из Dataframe
Из вашего обновления явствует, что ваша матрица Пирсона является пандой Dataframe. Здесь та же процедура, начиная с Dataframe. Вы можете использовать специальный метод networkx nx.from_pandas_adjacency(),
import pandas as pd
df = pd.DataFrame(a_mat)
Создать график
pearson_net = nx.from_pandas_adjacency(df)
pos = nx.spring_layout(pearson_net)
nx.draw_networkx_nodes(pearson_net,pos=pos)
nx.draw_networkx_edges(pearson_net,pos=pos)
nx.draw_networkx_edge_labels(pearson_net, pos=pos)
plt.axis('off')
plt.show()
[Из]:
Вызов методов связующего дерева
tree1=nx.minimum_spanning_tree(pearson_net)
tree2=nx.maximum_spanning_tree(pearson_net)
tree1.edges(data=True)
[Из]:
EdgeDataView([(0, 3, {'weight': 0.547633}), (1, 3, {'weight': 0.542115}), (2, 3, {'weight': 0.50037})])
nx.draw_networkx_nodes(tree1,pos=pos)
nx.draw_networkx_edges(tree1,pos=pos)
nx.draw_networkx_edge_labels(tree1, pos=pos)
plt.axis('off')
plt.show()
[Из]:
tree2.edges(data=True)
[Из]:
EdgeDataView([(0, 2, {'weight': 0.667419}), (0, 3, {'weight': 0.547633}), (1, 2, {'weight': 0.676438})])
nx.draw_networkx_nodes(tree2,pos=pos)
nx.draw_networkx_edges(tree2,pos=pos)
nx.draw_networkx_edge_labels(tree2, pos=pos)
plt.axis('off')
plt.show()
[Из]: