Как объединить (объединить) фреймы данных (внутренний, внешний, левый, правый)?
Даны два фрейма данных:
df1 = data.frame(CustomerId = c(1:6), Product = c(rep("Toaster", 3), rep("Radio", 3)))
df2 = data.frame(CustomerId = c(2, 4, 6), State = c(rep("Alabama", 2), rep("Ohio", 1)))
df1
# CustomerId Product
# 1 Toaster
# 2 Toaster
# 3 Toaster
# 4 Radio
# 5 Radio
# 6 Radio
df2
# CustomerId State
# 2 Alabama
# 4 Alabama
# 6 Ohio
Как я могу сделать стиль базы данных, то есть, стиль SQL, объединяет? То есть как я могу получить:
- Внутреннее соединение
df1а такжеdf2:
Вернуть только те строки, в которых левая таблица имеет соответствующие ключи в правой таблице. - Внешнее соединение
df1а такжеdf2:
Возвращает все строки из обеих таблиц, объединяет записи слева, которые имеют соответствующие ключи в правой таблице. - Левое внешнее соединение (или просто левое соединение)
df1а такжеdf2
Вернуть все строки из левой таблицы и все строки с соответствующими ключами из правой таблицы. - Правое внешнее соединение
df1а такжеdf2
Вернуть все строки из правой таблицы и все строки с соответствующими ключами из левой таблицы.
Дополнительный кредит:
Как я могу сделать оператор выбора в стиле SQL?
13 ответов
Используя merge Функция и ее необязательные параметры:
Внутреннее соединение: merge(df1, df2) будет работать для этих примеров, потому что R автоматически объединяет кадры по общим именам переменных, но вы, скорее всего, захотите указать merge(df1, df2, by = "CustomerId") чтобы убедиться, что вы сопоставляете только те поля, которые вы хотели. Вы также можете использовать by.x а также by.y параметры, если совпадающие переменные имеют разные имена в разных фреймах данных.
Внешнее соединение: merge(x = df1, y = df2, by = "CustomerId", all = TRUE)
Левый внешний: merge(x = df1, y = df2, by = "CustomerId", all.x = TRUE)
Правый внешний: merge(x = df1, y = df2, by = "CustomerId", all.y = TRUE)
Перекрестное соединение: merge(x = df1, y = df2, by = NULL)
Как и в случае внутреннего соединения, вы, вероятно, захотите явно передать CustomerId в R в качестве соответствующей переменной. Я думаю, что почти всегда лучше явно указывать идентификаторы, по которым вы хотите объединить; безопаснее, если входные данные изменяются неожиданно и их легче читать позже.
Вы можете объединить несколько столбцов, дав by вектор, например, by = c("CustomerId", "OrderId"),
Если имена столбцов для объединения не совпадают, вы можете указать, например, by.x = "CustomerId_in_df1", by.y = "CustomerId_in_df2" где CustomerId_in_df1 имя столбца в первом кадре данных и CustomerId_in_df2 Имя столбца во втором фрейме данных. (Это также могут быть векторы, если вам нужно объединить несколько столбцов.)
Я бы порекомендовал проверить пакет sqldf Габора Гротендика, который позволяет вам выражать эти операции в SQL.
library(sqldf)
## inner join
df3 <- sqldf("SELECT CustomerId, Product, State
FROM df1
JOIN df2 USING(CustomerID)")
## left join (substitute 'right' for right join)
df4 <- sqldf("SELECT CustomerId, Product, State
FROM df1
LEFT JOIN df2 USING(CustomerID)")
Я считаю, что синтаксис SQL является более простым и более естественным, чем его эквивалент R (но это может просто отражать мою предвзятость RDBMS).
Посмотрите sqlf GitHub Габора для получения дополнительной информации о присоединениях.
Вы также можете делать соединения, используя удивительный пакет dplyr Хэдли Уикхэма.
library(dplyr)
#make sure that CustomerId cols are both type numeric
#they ARE not using the provided code in question and dplyr will complain
df1$CustomerId <- as.numeric(df1$CustomerId)
df2$CustomerId <- as.numeric(df2$CustomerId)
Мутирующие объединения: добавление столбцов в df1 с использованием совпадений в df2
#inner
inner_join(df1, df2)
#left outer
left_join(df1, df2)
#right outer
right_join(df1, df2)
#alternate right outer
left_join(df2, df1)
#full join
full_join(df1, df2)
Фильтрация объединений: отфильтровывать строки в df1, не изменять столбцы
semi_join(df1, df2) #keep only observations in df1 that match in df2.
anti_join(df1, df2) #drops all observations in df1 that match in df2.
Существует метод data.table для внутреннего объединения, который очень экономит время и память (и необходим для некоторых больших data.frames):
library(data.table)
dt1 <- data.table(df1, key = "CustomerId")
dt2 <- data.table(df2, key = "CustomerId")
joined.dt1.dt.2 <- dt1[dt2]
merge также работает на data.tables (так как это универсальный и вызывает merge.data.table)
merge(dt1, dt2)
data.table документирован на стеке потока:
Как сделать операцию слияния data.table
Перевод SQL-соединений по внешним ключам в синтаксис R data.table.
Эффективные альтернативы объединению для больших данных. Рамки R
Как сделать базовое левое внешнее соединение с data.table в R?
Еще одним вариантом является join функция найдена в пакете plyr
library(plyr)
join(df1, df2,
type = "inner")
# CustomerId Product State
# 1 2 Toaster Alabama
# 2 4 Radio Alabama
# 3 6 Radio Ohio
Варианты для type: inner, left, right, full,
От ?join: В отличие от merge, [ join ] сохраняет порядок x независимо от того, какой тип соединения используется.
Есть несколько хороших примеров этого в R Wiki. Я украду пару здесь:
Метод слияния
Поскольку ваши ключи называются одинаково, короткий способ сделать внутреннее соединение - это merge():
merge(df1,df2)
полное внутреннее объединение (все записи из обеих таблиц) может быть создано с ключевым словом "all":
merge(df1,df2, all=TRUE)
левое внешнее соединение df1 и df2:
merge(df1,df2, all.x=TRUE)
правое внешнее соединение df1 и df2:
merge(df1,df2, all.y=TRUE)
Вы можете перевернуть их, дать им пощечину и потереть их, чтобы получить два других внешних объединения, о которых вы спрашивали:)
Подстрочный метод
Левое внешнее соединение с df1 слева с использованием метода нижнего индекса будет:
df1[,"State"]<-df2[df1[ ,"Product"], "State"]
Другая комбинация внешних объединений может быть создана путем добавления примера нижнего индекса внешнего соединения. (да, я знаю, что это эквивалентно высказыванию "Я оставлю это как упражнение для читателя...")
Обновление методов data.table для объединения наборов данных. Ниже приведены примеры для каждого типа соединения. Есть два метода, один из [.data.table при передаче второго data.table в качестве первого аргумента в подмножество, другим способом является использование merge функция, которая отправила быстрый метод data.table.
Обновление 2016-04-01 - и это не первоапрельская шутка!
В версии 1.9.7 объединения data.table теперь могут использовать существующий индекс, который значительно сокращает время объединения. Ниже код и бенчмарк НЕ используют индексы data.table при объединении. Если вы ищете соединение почти в реальном времени, вы должны использовать индексы data.table.
df1 = data.frame(CustomerId = c(1:6), Product = c(rep("Toaster", 3), rep("Radio", 3)))
df2 = data.frame(CustomerId = c(2L, 4L, 7L), State = c(rep("Alabama", 2), rep("Ohio", 1))) # one value changed to show full outer join
library(data.table)
dt1 = as.data.table(df1)
dt2 = as.data.table(df2)
setkey(dt1, CustomerId)
setkey(dt2, CustomerId)
# right outer join keyed data.tables
dt1[dt2]
setkey(dt1, NULL)
setkey(dt2, NULL)
# right outer join unkeyed data.tables - use `on` argument
dt1[dt2, on = "CustomerId"]
# left outer join - swap dt1 with dt2
dt2[dt1, on = "CustomerId"]
# inner join - use `nomatch` argument
dt1[dt2, nomatch=0L, on = "CustomerId"]
# anti join - use `!` operator
dt1[!dt2, on = "CustomerId"]
# inner join
merge(dt1, dt2, by = "CustomerId")
# full outer join
merge(dt1, dt2, by = "CustomerId", all = TRUE)
# see ?merge.data.table arguments for other cases
Ниже бенчмарк тестирует базы R, sqldf, dplyr и data.table.
Бенчмарк тестирует наборы данных без ключей / без индекса. Вы можете добиться еще большей производительности, если используете ключи в ваших data.tables или индексы с sqldf. Base R и dplyr не имеют индексов или ключей, поэтому я не включил этот сценарий в тест.
Тестирование выполняется для наборов данных строк 5M-1, в столбце объединения есть общие значения 5M-2, поэтому каждый сценарий (левый, правый, полный, внутренний) можно протестировать, и объединение все еще не является тривиальным.
library(microbenchmark)
library(sqldf)
library(dplyr)
library(data.table)
n = 5e6
set.seed(123)
df1 = data.frame(x=sample(n,n-1L), y1=rnorm(n-1L))
df2 = data.frame(x=sample(n,n-1L), y2=rnorm(n-1L))
dt1 = as.data.table(df1)
dt2 = as.data.table(df2)
# inner join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x"),
sqldf = sqldf("SELECT * FROM df1 INNER JOIN df2 ON df1.x = df2.x"),
dplyr = inner_join(df1, df2, by = "x"),
data.table = dt1[dt2, nomatch = 0L, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 15546.0097 16083.4915 16687.117 16539.0148 17388.290 18513.216 10
# sqldf 44392.6685 44709.7128 45096.401 45067.7461 45504.376 45563.472 10
# dplyr 4124.0068 4248.7758 4281.122 4272.3619 4342.829 4411.388 10
# data.table 937.2461 946.0227 1053.411 973.0805 1214.300 1281.958 10
# left outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all.x = TRUE),
sqldf = sqldf("SELECT * FROM df1 LEFT OUTER JOIN df2 ON df1.x = df2.x"),
dplyr = left_join(df1, df2, by = c("x"="x")),
data.table = dt2[dt1, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 16140.791 17107.7366 17441.9538 17414.6263 17821.9035 19453.034 10
# sqldf 43656.633 44141.9186 44777.1872 44498.7191 45288.7406 47108.900 10
# dplyr 4062.153 4352.8021 4780.3221 4409.1186 4450.9301 8385.050 10
# data.table 823.218 823.5557 901.0383 837.9206 883.3292 1277.239 10
# right outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all.y = TRUE),
sqldf = sqldf("SELECT * FROM df2 LEFT OUTER JOIN df1 ON df2.x = df1.x"),
dplyr = right_join(df1, df2, by = "x"),
data.table = dt1[dt2, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 15821.3351 15954.9927 16347.3093 16044.3500 16621.887 17604.794 10
# sqldf 43635.5308 43761.3532 43984.3682 43969.0081 44044.461 44499.891 10
# dplyr 3936.0329 4028.1239 4102.4167 4045.0854 4219.958 4307.350 10
# data.table 820.8535 835.9101 918.5243 887.0207 1005.721 1068.919 10
# full outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all = TRUE),
#sqldf = sqldf("SELECT * FROM df1 FULL OUTER JOIN df2 ON df1.x = df2.x"), # not supported
dplyr = full_join(df1, df2, by = "x"),
data.table = merge(dt1, dt2, by = "x", all = TRUE))
#Unit: seconds
# expr min lq mean median uq max neval
# base 16.176423 16.908908 17.485457 17.364857 18.271790 18.626762 10
# dplyr 7.610498 7.666426 7.745850 7.710638 7.832125 7.951426 10
# data.table 2.052590 2.130317 2.352626 2.208913 2.470721 2.951948 10
Новое в 2014 году:
Особенно, если вы также заинтересованы в манипулировании данными в целом (включая сортировку, фильтрацию, поднаборы, суммирование и т. Д.), Вы должны обязательно взглянуть на dplyr, который поставляется с множеством функций, предназначенных для облегчения вашей работы, особенно с фреймами данных и некоторыми другими типами баз данных. Он даже предлагает довольно сложный интерфейс SQL и даже функцию для преобразования (большей части) кода SQL непосредственно в R.
Четыре связанные с присоединением функции в пакете dplyr:
inner_join(x, y, by = NULL, copy = FALSE, ...): вернуть все строки из x, где есть совпадающие значения в y, и все столбцы из x и yleft_join(x, y, by = NULL, copy = FALSE, ...): вернуть все строки из x и все столбцы из x и ysemi_join(x, y, by = NULL, copy = FALSE, ...): вернуть все строки из x, где есть соответствующие значения в y, оставив только столбцы из x.anti_join(x, y, by = NULL, copy = FALSE, ...): вернуть все строки из x, где в y нет совпадающих значений, оставив только столбцы из x
Это все здесь очень подробно.
Выбор столбцов может быть сделан select(df,"column"), Если для вас этого недостаточно, то есть sql() функция, в которую вы можете вводить код SQL как есть, и она будет выполнять указанную вами операцию так же, как вы писали в R все это время (для получения дополнительной информации, пожалуйста, обратитесь к виньетке dplyr/database). Например, если применять правильно, sql("SELECT * FROM hflights") выберет все столбцы из таблицы dplyr "hflights" ("tbl").
dplyr начиная с версии 0.4 реализовал все эти объединения, включая outer_join, но стоит отметить, что в первых нескольких выпусках он не предлагал external_join, и в результате было довольно много плохого хакерского обходного пользовательского кода, который довольно долго перемещался (Вы все еще можете найти это в SO и Kaggle ответах того периода).
Основные моменты релиза, связанные с присоединением:
- Обработка для типа POSIXct, часовых поясов, дубликатов, различных уровней факторов. Лучше ошибки и предупреждения.
- Новый суффиксный аргумент для управления тем, что суффиксные дублированные имена переменных получают (# 1296)
- Реализация правого соединения и внешнего соединения (#96)
- Мутирующие объединения, которые добавляют новые переменные в одну таблицу из соответствующих строк в другой. Фильтрация объединений, которые фильтруют наблюдения из одной таблицы на основе того, соответствуют ли они наблюдениям из другой таблицы.
- Теперь можно left_join по различным переменным в каждой таблице: df1 %>% left_join(df2, c("var1" = "var2"))
- * _join () больше не переставляет имена столбцов (# 324)
v0.1.3 (4/2014)
- имеет inner_join, left_join, semi_join, anti_join
- external_join еще не реализован, отступление - использование base::merge() (или plyr::join())
- еще не реализовал right_join и outer_join
- Хэдли упоминает другие преимущества здесь
- одно незначительное объединение функций, которое в настоящее время имеет dplyr, - это возможность иметь отдельные столбцы by.x, by.y, как, например, в Python pandas.
Методы обхода комментариев Хадли в этом выпуске:
- right_join (x, y) - это то же самое, что left_join (y, x) в терминах строк, только столбцы будут разных порядков. Легко обходится с помощью select (new_column_order)
- external_join в основном представляет собой union (left_join (x, y), right_join (x, y)) - т.е. сохраняет все строки в обоих фреймах данных.
Для случая левого соединения с 0..*:0..1 кардинальность или право соединиться с 0..1:0..* кардинальность можно назначить на месте односторонние столбцы от столяра (0..1 стол) прямо на Joinee (0..* таблица), и, таким образом, избежать создания совершенно новой таблицы данных. Для этого необходимо сопоставить ключевые столбцы от joinee в joiner и проиндексировать + упорядочить строки joiner соответственно для назначения.
Если ключ представляет собой один столбец, то мы можем использовать один вызов match() сделать сопоставление. Это тот случай, который я расскажу в этом ответе.
Вот пример, основанный на OP, за исключением того, что я добавил дополнительную строку в df2 с идентификатором 7, чтобы проверить случай несовпадающего ключа в столяре. Это эффективно df1 оставил присоединиться df2:
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L)));
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas'));
df1[names(df2)[-1L]] <- df2[match(df1[,1L],df2[,1L]),-1L];
df1;
## CustomerId Product State
## 1 1 Toaster <NA>
## 2 2 Toaster Alabama
## 3 3 Toaster <NA>
## 4 4 Radio Alabama
## 5 5 Radio <NA>
## 6 6 Radio Ohio
Выше я жестко закодировал предположение, что ключевой столбец является первым столбцом обеих входных таблиц. Я бы сказал, что в целом это не является необоснованным предположением, поскольку, если у вас есть data.frame с ключевым столбцом, было бы странно, если бы он не был установлен в качестве первого столбца data.frame из начало. И вы всегда можете изменить порядок столбцов, чтобы сделать это так. Преимущественным следствием этого предположения является то, что имя ключевого столбца не обязательно должно быть жестко закодировано, хотя я полагаю, что оно просто заменяет одно предположение другим. Краткость - еще одно преимущество целочисленной индексации, а также скорость. В нижеприведенных тестах я изменю реализацию на использование индексации имен строк в соответствии с конкурирующими реализациями.
Я думаю, что это особенно подходящее решение, если у вас есть несколько таблиц, которые вы хотите объединить против одной большой таблицы. Повторное восстановление всей таблицы для каждого слияния было бы ненужным и неэффективным.
С другой стороны, если вам нужно, чтобы участник оставался неизменным в ходе этой операции по какой-либо причине, то это решение не может быть использовано, поскольку оно изменяет участника напрямую. Хотя в этом случае вы можете просто сделать копию и выполнить на месте назначение (назначения) для копии.
В качестве дополнительного примечания я кратко рассмотрел возможные решения для сопоставления многоколоночных ключей. К сожалению, единственные подходящие решения, которые я нашел, были:
- неэффективные объединения. например
match(interaction(df1$a,df1$b),interaction(df2$a,df2$b))или та же идея сpaste(), - неэффективные декартовы союзы, например
outer(df1$a,df2$a,`==`) & outer(df1$b,df2$b,`==`), - база R
merge()и эквивалентные функции слияния на основе пакетов, которые всегда выделяют новую таблицу для возврата объединенного результата и, таким образом, не подходят для решения на основе назначения на месте.
Например, см. Сопоставление нескольких столбцов в разных фреймах данных и получение другого столбца в качестве результата, сопоставление двух столбцов с двумя другими столбцами, Сопоставление по нескольким столбцам и ответ на этот вопрос, в котором я изначально придумал решение на месте, Объединить два кадра данных с разным количеством строк в R.
Бенчмаркинг
Я решил сделать свой собственный сравнительный анализ, чтобы увидеть, как подход на месте назначения сравнивается с другими решениями, которые были предложены в этом вопросе.
Тестовый код:
library(microbenchmark);
library(data.table);
library(sqldf);
library(plyr);
library(dplyr);
solSpecs <- list(
merge=list(testFuncs=list(
inner=function(df1,df2,key) merge(df1,df2,key),
left =function(df1,df2,key) merge(df1,df2,key,all.x=T),
right=function(df1,df2,key) merge(df1,df2,key,all.y=T),
full =function(df1,df2,key) merge(df1,df2,key,all=T)
)),
data.table.unkeyed=list(argSpec='data.table.unkeyed',testFuncs=list(
inner=function(dt1,dt2,key) dt1[dt2,on=key,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2,key) dt2[dt1,on=key,allow.cartesian=T],
right=function(dt1,dt2,key) dt1[dt2,on=key,allow.cartesian=T],
full =function(dt1,dt2,key) merge(dt1,dt2,key,all=T,allow.cartesian=T) ## calls merge.data.table()
)),
data.table.keyed=list(argSpec='data.table.keyed',testFuncs=list(
inner=function(dt1,dt2) dt1[dt2,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2) dt2[dt1,allow.cartesian=T],
right=function(dt1,dt2) dt1[dt2,allow.cartesian=T],
full =function(dt1,dt2) merge(dt1,dt2,all=T,allow.cartesian=T) ## calls merge.data.table()
)),
sqldf.unindexed=list(testFuncs=list( ## note: must pass connection=NULL to avoid running against the live DB connection, which would result in collisions with the residual tables from the last query upload
inner=function(df1,df2,key) sqldf(paste0('select * from df1 inner join df2 using(',paste(collapse=',',key),')'),connection=NULL),
left =function(df1,df2,key) sqldf(paste0('select * from df1 left join df2 using(',paste(collapse=',',key),')'),connection=NULL),
right=function(df1,df2,key) sqldf(paste0('select * from df2 left join df1 using(',paste(collapse=',',key),')'),connection=NULL) ## can't do right join proper, not yet supported; inverted left join is equivalent
##full =function(df1,df2,key) sqldf(paste0('select * from df1 full join df2 using(',paste(collapse=',',key),')'),connection=NULL) ## can't do full join proper, not yet supported; possible to hack it with a union of left joins, but too unreasonable to include in testing
)),
sqldf.indexed=list(testFuncs=list( ## important: requires an active DB connection with preindexed main.df1 and main.df2 ready to go; arguments are actually ignored
inner=function(df1,df2,key) sqldf(paste0('select * from main.df1 inner join main.df2 using(',paste(collapse=',',key),')')),
left =function(df1,df2,key) sqldf(paste0('select * from main.df1 left join main.df2 using(',paste(collapse=',',key),')')),
right=function(df1,df2,key) sqldf(paste0('select * from main.df2 left join main.df1 using(',paste(collapse=',',key),')')) ## can't do right join proper, not yet supported; inverted left join is equivalent
##full =function(df1,df2,key) sqldf(paste0('select * from main.df1 full join main.df2 using(',paste(collapse=',',key),')')) ## can't do full join proper, not yet supported; possible to hack it with a union of left joins, but too unreasonable to include in testing
)),
plyr=list(testFuncs=list(
inner=function(df1,df2,key) join(df1,df2,key,'inner'),
left =function(df1,df2,key) join(df1,df2,key,'left'),
right=function(df1,df2,key) join(df1,df2,key,'right'),
full =function(df1,df2,key) join(df1,df2,key,'full')
)),
dplyr=list(testFuncs=list(
inner=function(df1,df2,key) inner_join(df1,df2,key),
left =function(df1,df2,key) left_join(df1,df2,key),
right=function(df1,df2,key) right_join(df1,df2,key),
full =function(df1,df2,key) full_join(df1,df2,key)
)),
in.place=list(testFuncs=list(
left =function(df1,df2,key) { cns <- setdiff(names(df2),key); df1[cns] <- df2[match(df1[,key],df2[,key]),cns]; df1; },
right=function(df1,df2,key) { cns <- setdiff(names(df1),key); df2[cns] <- df1[match(df2[,key],df1[,key]),cns]; df2; }
))
);
getSolTypes <- function() names(solSpecs);
getJoinTypes <- function() unique(unlist(lapply(solSpecs,function(x) names(x$testFuncs))));
getArgSpec <- function(argSpecs,key=NULL) if (is.null(key)) argSpecs$default else argSpecs[[key]];
initSqldf <- function() {
sqldf(); ## creates sqlite connection on first run, cleans up and closes existing connection otherwise
if (exists('sqldfInitFlag',envir=globalenv(),inherits=F) && sqldfInitFlag) { ## false only on first run
sqldf(); ## creates a new connection
} else {
assign('sqldfInitFlag',T,envir=globalenv()); ## set to true for the one and only time
}; ## end if
invisible();
}; ## end initSqldf()
setUpBenchmarkCall <- function(argSpecs,joinType,solTypes=getSolTypes(),env=parent.frame()) {
## builds and returns a list of expressions suitable for passing to the list argument of microbenchmark(), and assigns variables to resolve symbol references in those expressions
callExpressions <- list();
nms <- character();
for (solType in solTypes) {
testFunc <- solSpecs[[solType]]$testFuncs[[joinType]];
if (is.null(testFunc)) next; ## this join type is not defined for this solution type
testFuncName <- paste0('tf.',solType);
assign(testFuncName,testFunc,envir=env);
argSpecKey <- solSpecs[[solType]]$argSpec;
argSpec <- getArgSpec(argSpecs,argSpecKey);
argList <- setNames(nm=names(argSpec$args),vector('list',length(argSpec$args)));
for (i in seq_along(argSpec$args)) {
argName <- paste0('tfa.',argSpecKey,i);
assign(argName,argSpec$args[[i]],envir=env);
argList[[i]] <- if (i%in%argSpec$copySpec) call('copy',as.symbol(argName)) else as.symbol(argName);
}; ## end for
callExpressions[[length(callExpressions)+1L]] <- do.call(call,c(list(testFuncName),argList),quote=T);
nms[length(nms)+1L] <- solType;
}; ## end for
names(callExpressions) <- nms;
callExpressions;
}; ## end setUpBenchmarkCall()
harmonize <- function(res) {
res <- as.data.frame(res); ## coerce to data.frame
for (ci in which(sapply(res,is.factor))) res[[ci]] <- as.character(res[[ci]]); ## coerce factor columns to character
for (ci in which(sapply(res,is.logical))) res[[ci]] <- as.integer(res[[ci]]); ## coerce logical columns to integer (works around sqldf quirk of munging logicals to integers)
##for (ci in which(sapply(res,inherits,'POSIXct'))) res[[ci]] <- as.double(res[[ci]]); ## coerce POSIXct columns to double (works around sqldf quirk of losing POSIXct class) ----- POSIXct doesn't work at all in sqldf.indexed
res <- res[order(names(res))]; ## order columns
res <- res[do.call(order,res),]; ## order rows
res;
}; ## end harmonize()
checkIdentical <- function(argSpecs,solTypes=getSolTypes()) {
for (joinType in getJoinTypes()) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
if (length(callExpressions)<2L) next;
ex <- harmonize(eval(callExpressions[[1L]]));
for (i in seq(2L,len=length(callExpressions)-1L)) {
y <- harmonize(eval(callExpressions[[i]]));
if (!isTRUE(all.equal(ex,y,check.attributes=F))) {
ex <<- ex;
y <<- y;
solType <- names(callExpressions)[i];
stop(paste0('non-identical: ',solType,' ',joinType,'.'));
}; ## end if
}; ## end for
}; ## end for
invisible();
}; ## end checkIdentical()
testJoinType <- function(argSpecs,joinType,solTypes=getSolTypes(),metric=NULL,times=100L) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
bm <- microbenchmark(list=callExpressions,times=times);
if (is.null(metric)) return(bm);
bm <- summary(bm);
res <- setNames(nm=names(callExpressions),bm[[metric]]);
attr(res,'unit') <- attr(bm,'unit');
res;
}; ## end testJoinType()
testAllJoinTypes <- function(argSpecs,solTypes=getSolTypes(),metric=NULL,times=100L) {
joinTypes <- getJoinTypes();
resList <- setNames(nm=joinTypes,lapply(joinTypes,function(joinType) testJoinType(argSpecs,joinType,solTypes,metric,times)));
if (is.null(metric)) return(resList);
units <- unname(unlist(lapply(resList,attr,'unit')));
res <- do.call(data.frame,c(list(join=joinTypes),setNames(nm=solTypes,rep(list(rep(NA_real_,length(joinTypes))),length(solTypes))),list(unit=units,stringsAsFactors=F)));
for (i in seq_along(resList)) res[i,match(names(resList[[i]]),names(res))] <- resList[[i]];
res;
}; ## end testAllJoinTypes()
testGrid <- function(makeArgSpecsFunc,sizes,overlaps,solTypes=getSolTypes(),joinTypes=getJoinTypes(),metric='median',times=100L) {
res <- expand.grid(size=sizes,overlap=overlaps,joinType=joinTypes,stringsAsFactors=F);
res[solTypes] <- NA_real_;
res$unit <- NA_character_;
for (ri in seq_len(nrow(res))) {
size <- res$size[ri];
overlap <- res$overlap[ri];
joinType <- res$joinType[ri];
argSpecs <- makeArgSpecsFunc(size,overlap);
checkIdentical(argSpecs,solTypes);
cur <- testJoinType(argSpecs,joinType,solTypes,metric,times);
res[ri,match(names(cur),names(res))] <- cur;
res$unit[ri] <- attr(cur,'unit');
}; ## end for
res;
}; ## end testGrid()
Вот пример из примера, основанного на OP, который я продемонстрировал ранее:
## OP's example, supplemented with a non-matching row in df2
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L))),
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas')),
'CustomerId'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'CustomerId'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),CustomerId),
setkey(as.data.table(df2),CustomerId)
))
);
## prepare sqldf
initSqldf();
sqldf('create index df1_key on df1(CustomerId);'); ## upload and create an sqlite index on df1
sqldf('create index df2_key on df2(CustomerId);'); ## upload and create an sqlite index on df2
checkIdentical(argSpecs);
testAllJoinTypes(argSpecs,metric='median');
## join merge data.table.unkeyed data.table.keyed sqldf.unindexed sqldf.indexed plyr dplyr in.place unit
## 1 inner 644.259 861.9345 923.516 9157.752 1580.390 959.2250 270.9190 NA microseconds
## 2 left 713.539 888.0205 910.045 8820.334 1529.714 968.4195 270.9185 224.3045 microseconds
## 3 right 1221.804 909.1900 923.944 8930.668 1533.135 1063.7860 269.8495 218.1035 microseconds
## 4 full 1302.203 3107.5380 3184.729 NA NA 1593.6475 270.7055 NA microseconds
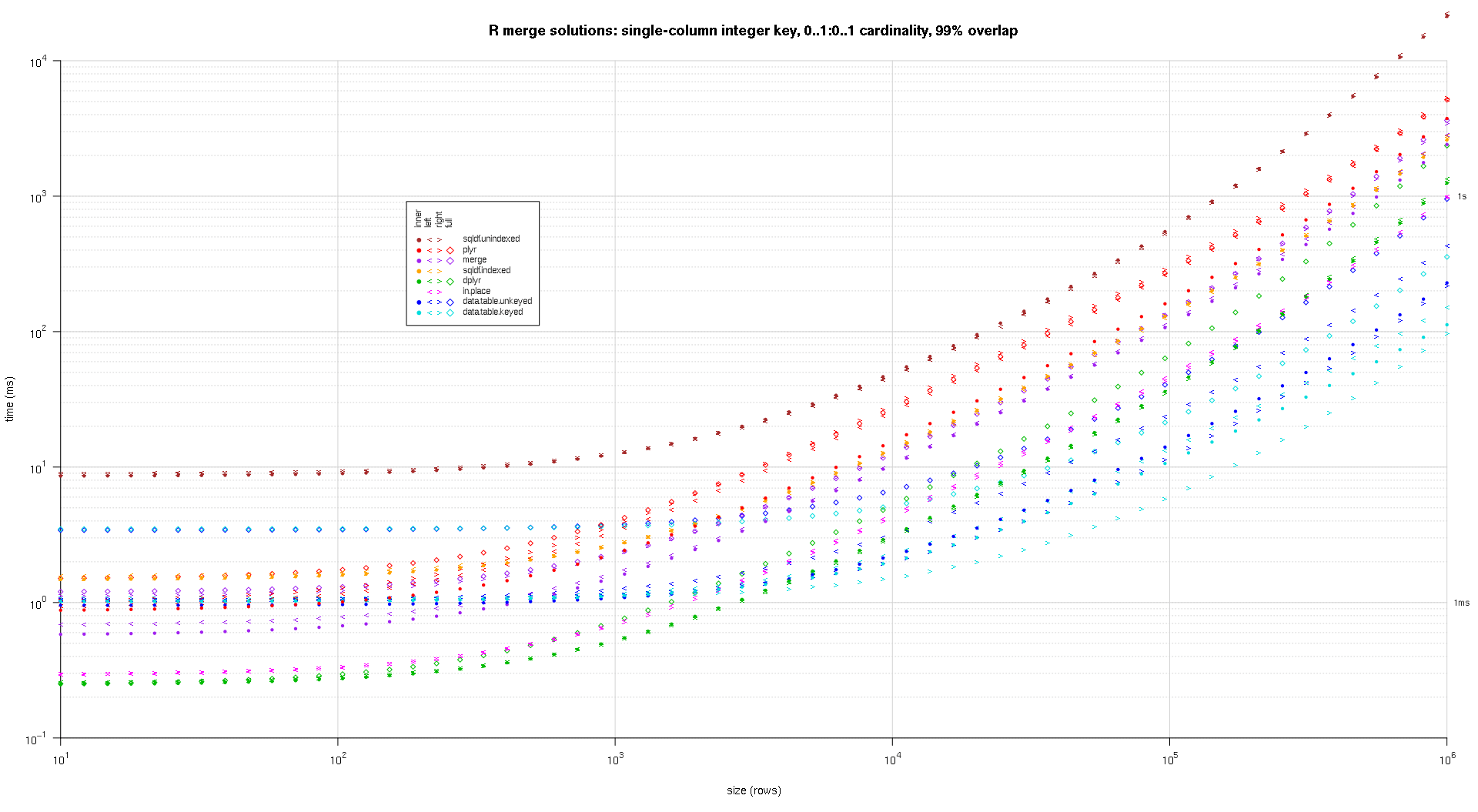
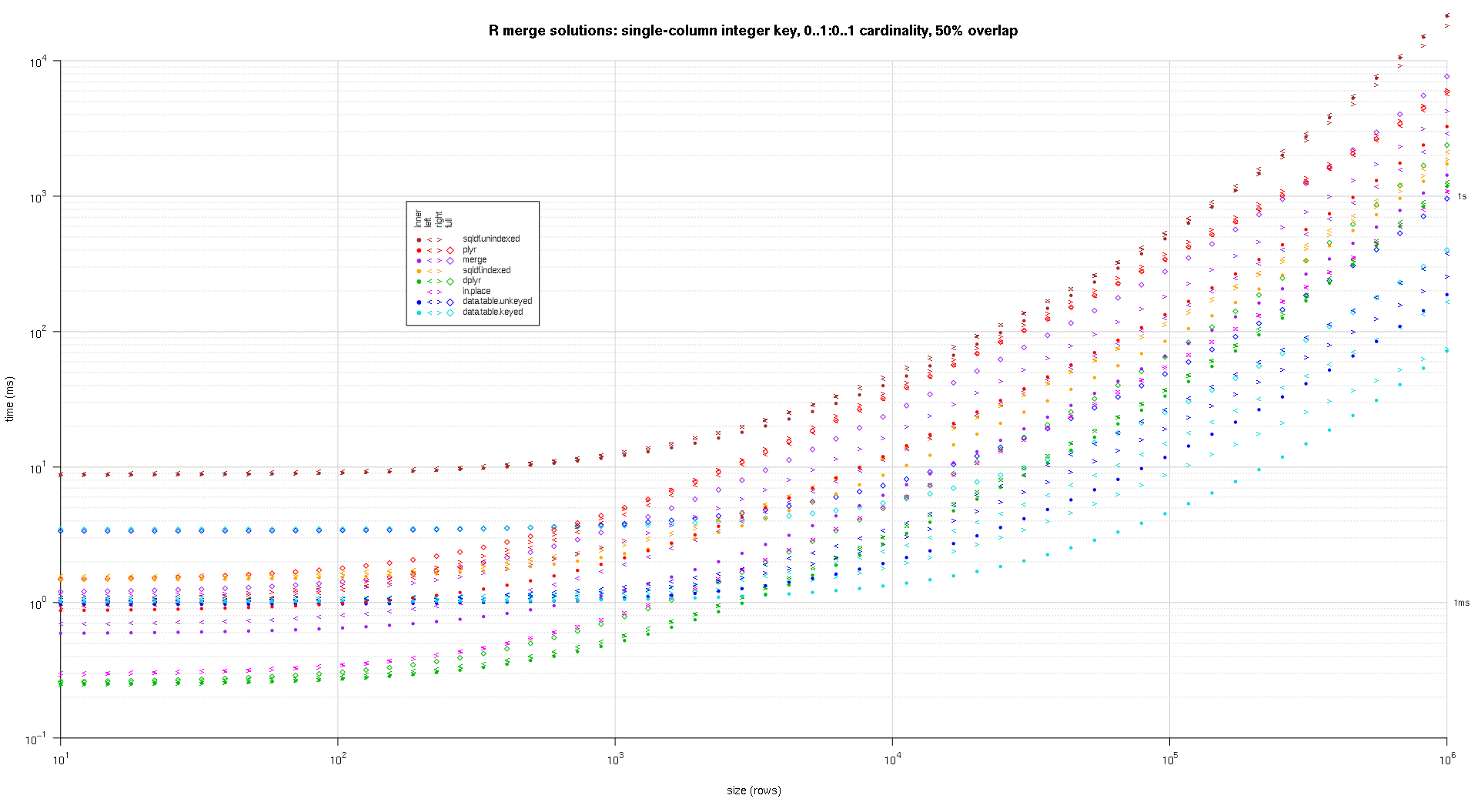
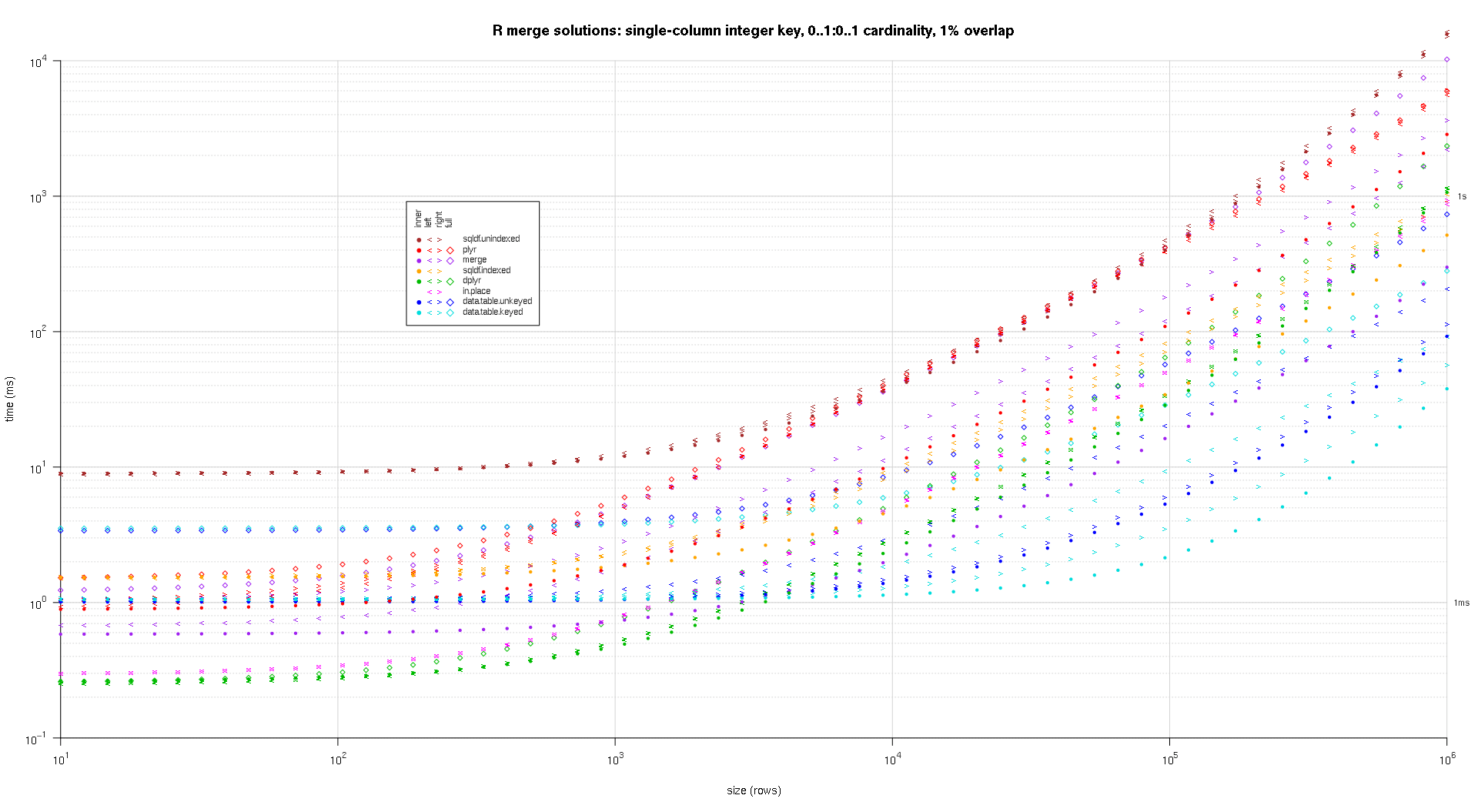
Здесь я сравниваю случайные входные данные, пробуя разные шкалы и разные комбинации клавиш для двух таблиц ввода. Этот тест по-прежнему ограничен случаем целочисленного ключа из одного столбца. Кроме того, чтобы гарантировать, что решение на месте будет работать как для левого, так и для правого объединений одних и тех же таблиц, все данные случайных тестов используют 0..1:0..1 кардинальное. Это реализуется путем выборки без замены ключевого столбца первого data.frame при генерации ключевого столбца второго data.frame.
makeArgSpecs.singleIntegerKey.optionalOneToOne <- function(size,overlap) {
com <- as.integer(size*overlap);
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(id=sample(size),y1=rnorm(size),y2=rnorm(size)),
df2 <- data.frame(id=sample(c(if (com>0L) sample(df1$id,com) else integer(),seq(size+1L,len=size-com))),y3=rnorm(size),y4=rnorm(size)),
'id'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'id'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),id),
setkey(as.data.table(df2),id)
))
);
## prepare sqldf
initSqldf();
sqldf('create index df1_key on df1(id);'); ## upload and create an sqlite index on df1
sqldf('create index df2_key on df2(id);'); ## upload and create an sqlite index on df2
argSpecs;
}; ## end makeArgSpecs.singleIntegerKey.optionalOneToOne()
## cross of various input sizes and key overlaps
sizes <- c(1e1L,1e3L,1e6L);
overlaps <- c(0.99,0.5,0.01);
system.time({ res <- testGrid(makeArgSpecs.singleIntegerKey.optionalOneToOne,sizes,overlaps); });
## user system elapsed
## 22024.65 12308.63 34493.19
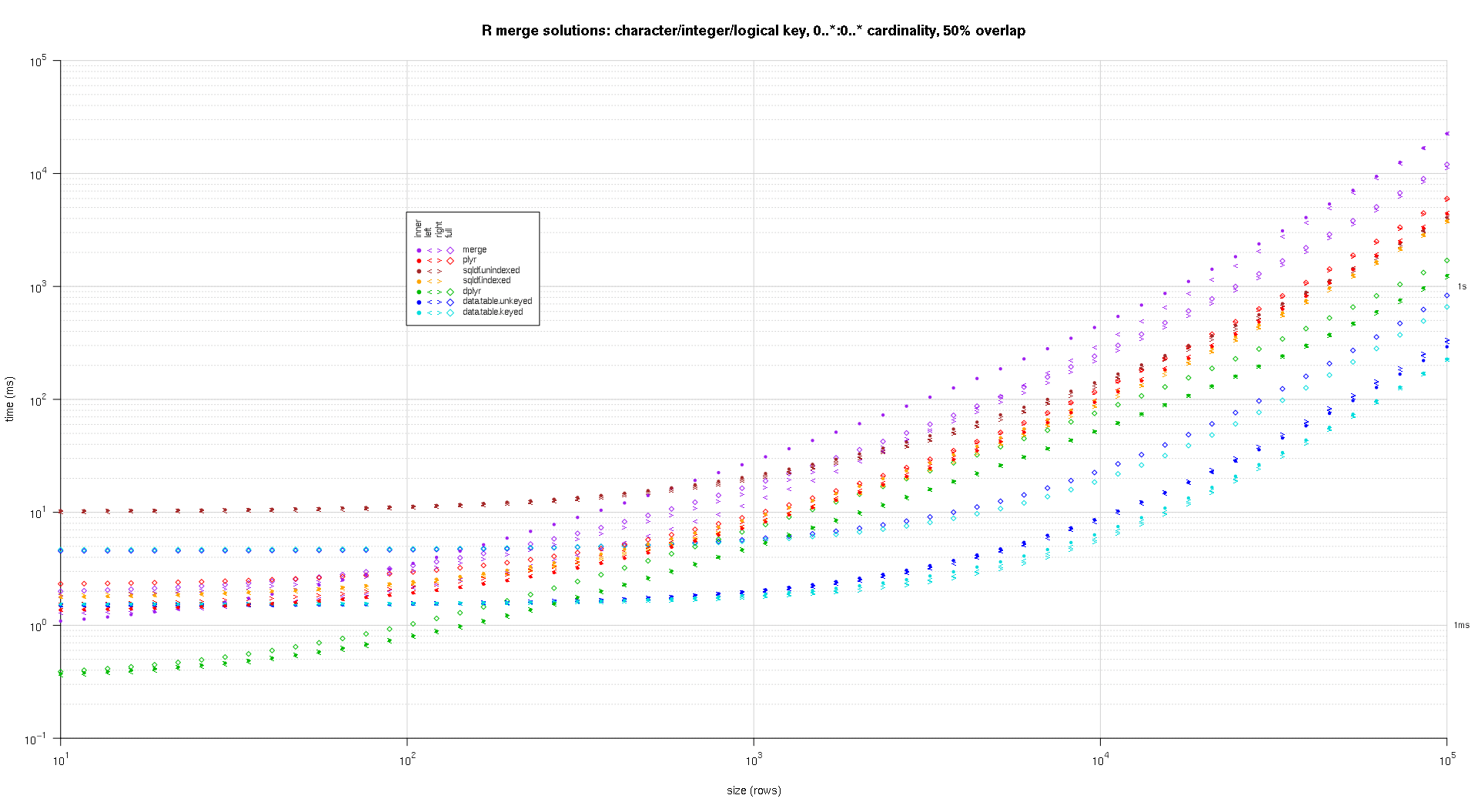
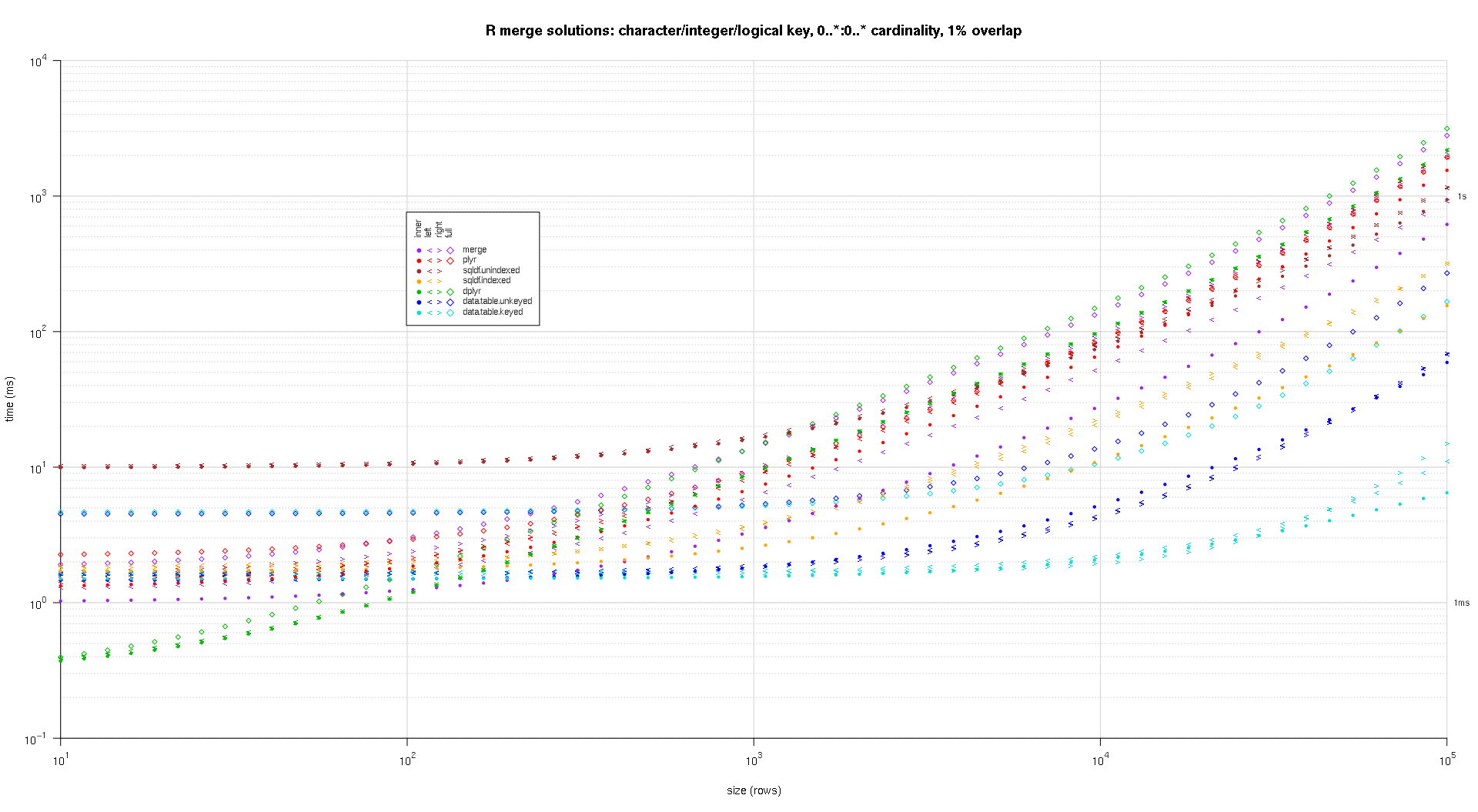
Я написал некоторый код для создания графиков log-log приведенных выше результатов. Я создал отдельный график для каждого процента перекрытия. Это немного сумбурно, но мне нравится, когда все типы решений и типы соединений представлены на одном графике.
Я использовал сплайн-интерполяцию, чтобы показать плавную кривую для каждой комбинации типа решение / объединение, нарисованную отдельными символами pch. Тип соединения фиксируется символом pch, используя точку для внутреннего, левого и правого угловых скобок для левого и правого и ромб для полного. Тип решения определяется цветом, как показано в легенде.
plotRes <- function(res,titleFunc,useFloor=F) {
solTypes <- setdiff(names(res),c('size','overlap','joinType','unit')); ## derive from res
normMult <- c(microseconds=1e-3,milliseconds=1); ## normalize to milliseconds
joinTypes <- getJoinTypes();
cols <- c(merge='purple',data.table.unkeyed='blue',data.table.keyed='#00DDDD',sqldf.unindexed='brown',sqldf.indexed='orange',plyr='red',dplyr='#00BB00',in.place='magenta');
pchs <- list(inner=20L,left='<',right='>',full=23L);
cexs <- c(inner=0.7,left=1,right=1,full=0.7);
NP <- 60L;
ord <- order(decreasing=T,colMeans(res[res$size==max(res$size),solTypes],na.rm=T));
ymajors <- data.frame(y=c(1,1e3),label=c('1ms','1s'),stringsAsFactors=F);
for (overlap in unique(res$overlap)) {
x1 <- res[res$overlap==overlap,];
x1[solTypes] <- x1[solTypes]*normMult[x1$unit]; x1$unit <- NULL;
xlim <- c(1e1,max(x1$size));
xticks <- 10^seq(log10(xlim[1L]),log10(xlim[2L]));
ylim <- c(1e-1,10^((if (useFloor) floor else ceiling)(log10(max(x1[solTypes],na.rm=T))))); ## use floor() to zoom in a little more, only sqldf.unindexed will break above, but xpd=NA will keep it visible
yticks <- 10^seq(log10(ylim[1L]),log10(ylim[2L]));
yticks.minor <- rep(yticks[-length(yticks)],each=9L)*1:9;
plot(NA,xlim=xlim,ylim=ylim,xaxs='i',yaxs='i',axes=F,xlab='size (rows)',ylab='time (ms)',log='xy');
abline(v=xticks,col='lightgrey');
abline(h=yticks.minor,col='lightgrey',lty=3L);
abline(h=yticks,col='lightgrey');
axis(1L,xticks,parse(text=sprintf('10^%d',as.integer(log10(xticks)))));
axis(2L,yticks,parse(text=sprintf('10^%d',as.integer(log10(yticks)))),las=1L);
axis(4L,ymajors$y,ymajors$label,las=1L,tick=F,cex.axis=0.7,hadj=0.5);
for (joinType in rev(joinTypes)) { ## reverse to draw full first, since it's larger and would be more obtrusive if drawn last
x2 <- x1[x1$joinType==joinType,];
for (solType in solTypes) {
if (any(!is.na(x2[[solType]]))) {
xy <- spline(x2$size,x2[[solType]],xout=10^(seq(log10(x2$size[1L]),log10(x2$size[nrow(x2)]),len=NP)));
points(xy$x,xy$y,pch=pchs[[joinType]],col=cols[solType],cex=cexs[joinType],xpd=NA);
}; ## end if
}; ## end for
}; ## end for
## custom legend
## due to logarithmic skew, must do all distance calcs in inches, and convert to user coords afterward
## the bottom-left corner of the legend will be defined in normalized figure coords, although we can convert to inches immediately
leg.cex <- 0.7;
leg.x.in <- grconvertX(0.275,'nfc','in');
leg.y.in <- grconvertY(0.6,'nfc','in');
leg.x.user <- grconvertX(leg.x.in,'in');
leg.y.user <- grconvertY(leg.y.in,'in');
leg.outpad.w.in <- 0.1;
leg.outpad.h.in <- 0.1;
leg.midpad.w.in <- 0.1;
leg.midpad.h.in <- 0.1;
leg.sol.w.in <- max(strwidth(solTypes,'in',leg.cex));
leg.sol.h.in <- max(strheight(solTypes,'in',leg.cex))*1.5; ## multiplication factor for greater line height
leg.join.w.in <- max(strheight(joinTypes,'in',leg.cex))*1.5; ## ditto
leg.join.h.in <- max(strwidth(joinTypes,'in',leg.cex));
leg.main.w.in <- leg.join.w.in*length(joinTypes);
leg.main.h.in <- leg.sol.h.in*length(solTypes);
leg.x2.user <- grconvertX(leg.x.in+leg.outpad.w.in*2+leg.main.w.in+leg.midpad.w.in+leg.sol.w.in,'in');
leg.y2.user <- grconvertY(leg.y.in+leg.outpad.h.in*2+leg.main.h.in+leg.midpad.h.in+leg.join.h.in,'in');
leg.cols.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.join.w.in*(0.5+seq(0L,length(joinTypes)-1L)),'in');
leg.lines.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in-leg.sol.h.in*(0.5+seq(0L,length(solTypes)-1L)),'in');
leg.sol.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.main.w.in+leg.midpad.w.in,'in');
leg.join.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in+leg.midpad.h.in,'in');
rect(leg.x.user,leg.y.user,leg.x2.user,leg.y2.user,col='white');
text(leg.sol.x.user,leg.lines.y.user,solTypes[ord],cex=leg.cex,pos=4L,offset=0);
text(leg.cols.x.user,leg.join.y.user,joinTypes,cex=leg.cex,pos=4L,offset=0,srt=90); ## srt rotation applies *after* pos/offset positioning
for (i in seq_along(joinTypes)) {
joinType <- joinTypes[i];
points(rep(leg.cols.x.user[i],length(solTypes)),ifelse(colSums(!is.na(x1[x1$joinType==joinType,solTypes[ord]]))==0L,NA,leg.lines.y.user),pch=pchs[[joinType]],col=cols[solTypes[ord]]);
}; ## end for
title(titleFunc(overlap));
readline(sprintf('overlap %.02f',overlap));
}; ## end for
}; ## end plotRes()
titleFunc <- function(overlap) sprintf('R merge solutions: single-column integer key, 0..1:0..1 cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,T);
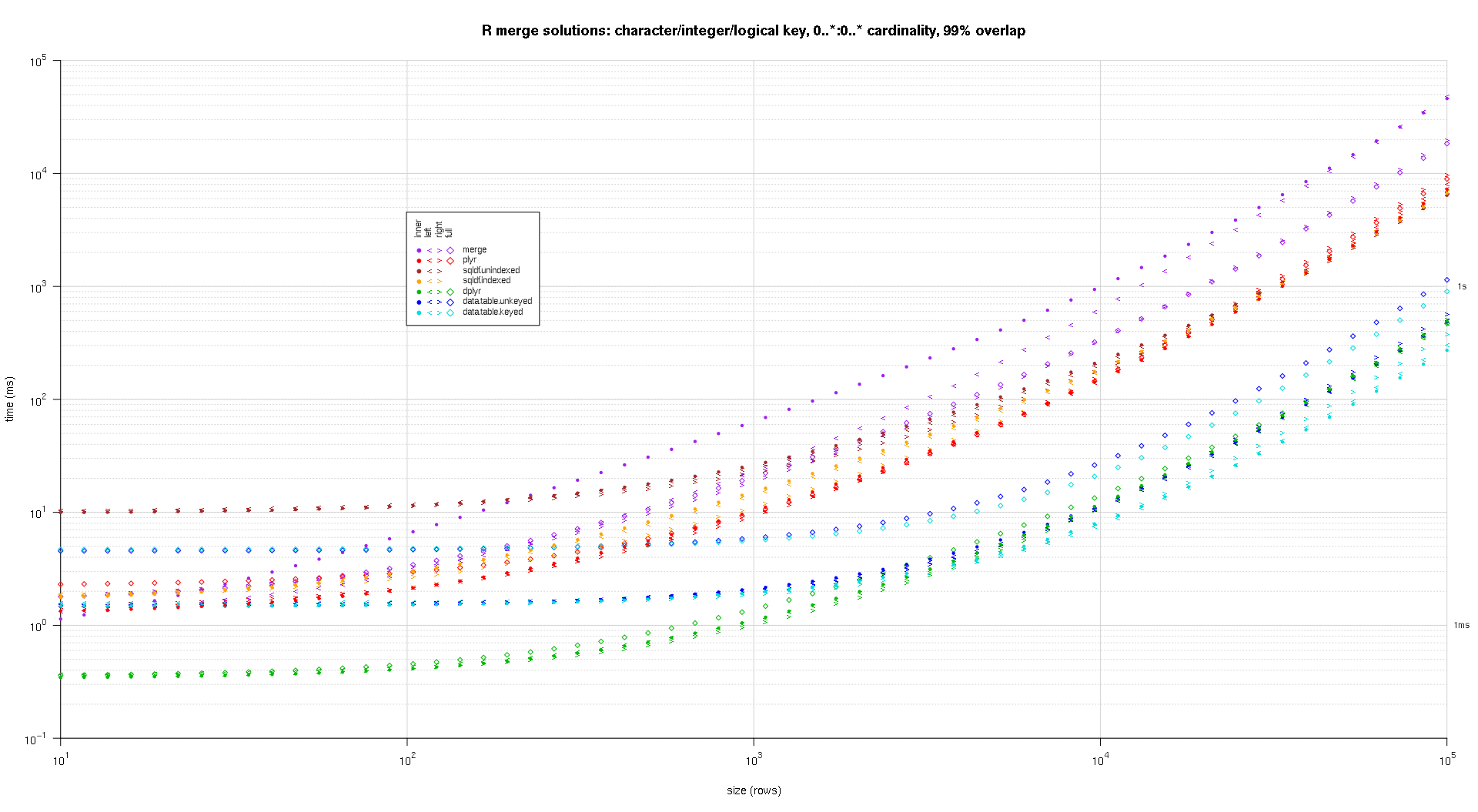
Вот второй крупномасштабный тест, который более тяжелый, с точки зрения количества и типов ключевых столбцов, а также количества элементов. Для этого теста я использую три ключевых столбца: один символ, одно целое и один логический, без ограничений по количеству элементов (то есть 0..*:0..*). (В общем случае не рекомендуется определять ключевые столбцы с двойными или сложными значениями из-за сложностей сравнения с плавающей точкой, и в основном никто никогда не использует необработанный тип, тем более для ключевых столбцов, поэтому я не включил эти типы в ключ Кроме того, ради информации, я изначально пытался использовать четыре ключевых столбца, включая ключевой столбец POSIXct, но тип POSIXct не очень хорошо работал с sqldf.indexed решение по какой-то причине, возможно, из-за аномалий сравнения с плавающей точкой, поэтому я удалил его.)
makeArgSpecs.assortedKey.optionalManyToMany <- function(size,overlap,uniquePct=75) {
## number of unique keys in df1
u1Size <- as.integer(size*uniquePct/100);
## (roughly) divide u1Size into bases, so we can use expand.grid() to produce the required number of unique key values with repetitions within individual key columns
## use ceiling() to ensure we cover u1Size; will truncate afterward
u1SizePerKeyColumn <- as.integer(ceiling(u1Size^(1/3)));
## generate the unique key values for df1
keys1 <- expand.grid(stringsAsFactors=F,
idCharacter=replicate(u1SizePerKeyColumn,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=sample(u1SizePerKeyColumn),
idLogical=sample(c(F,T),u1SizePerKeyColumn,T)
##idPOSIXct=as.POSIXct('2016-01-01 00:00:00','UTC')+sample(u1SizePerKeyColumn)
)[seq_len(u1Size),];
## rbind some repetitions of the unique keys; this will prepare one side of the many-to-many relationship
## also scramble the order afterward
keys1 <- rbind(keys1,keys1[sample(nrow(keys1),size-u1Size,T),])[sample(size),];
## common and unilateral key counts
com <- as.integer(size*overlap);
uni <- size-com;
## generate some unilateral keys for df2 by synthesizing outside of the idInteger range of df1
keys2 <- data.frame(stringsAsFactors=F,
idCharacter=replicate(uni,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=u1SizePerKeyColumn+sample(uni),
idLogical=sample(c(F,T),uni,T)
##idPOSIXct=as.POSIXct('2016-01-01 00:00:00','UTC')+u1SizePerKeyColumn+sample(uni)
);
## rbind random keys from df1; this will complete the many-to-many relationship
## also scramble the order afterward
keys2 <- rbind(keys2,keys1[sample(nrow(keys1),com,T),])[sample(size),];
##keyNames <- c('idCharacter','idInteger','idLogical','idPOSIXct');
keyNames <- c('idCharacter','idInteger','idLogical');
## note: was going to use raw and complex type for two of the non-key columns, but data.table doesn't seem to fully support them
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- cbind(stringsAsFactors=F,keys1,y1=sample(c(F,T),size,T),y2=sample(size),y3=rnorm(size),y4=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
df2 <- cbind(stringsAsFactors=F,keys2,y5=sample(c(F,T),size,T),y6=sample(size),y7=rnorm(size),y8=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
keyNames
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
keyNames
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkeyv(as.data.table(df1),keyNames),
setkeyv(as.data.table(df2),keyNames)
))
);
## prepare sqldf
initSqldf();
sqldf(paste0('create index df1_key on df1(',paste(collapse=',',keyNames),');')); ## upload and create an sqlite index on df1
sqldf(paste0('create index df2_key on df2(',paste(collapse=',',keyNames),');')); ## upload and create an sqlite index on df2
argSpecs;
}; ## end makeArgSpecs.assortedKey.optionalManyToMany()
sizes <- c(1e1L,1e3L,1e5L); ## 1e5L instead of 1e6L to respect more heavy-duty inputs
overlaps <- c(0.99,0.5,0.01);
solTypes <- setdiff(getSolTypes(),'in.place');
system.time({ res <- testGrid(makeArgSpecs.assortedKey.optionalManyToMany,sizes,overlaps,solTypes); });
## user system elapsed
## 38895.50 784.19 39745.53
Полученные графики, используя тот же код построения, приведенный выше:
titleFunc <- function(overlap) sprintf('R merge solutions: character/integer/logical key, 0..*:0..* cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,F);
Я неожиданно обнаружил, что при объединении двух фреймов данных с ~1 миллионом строк, один с двумя столбцами, а другой с ~20 merge(..., all.x = TRUE, all.y = TRUE) быть быстрее тогда dplyr::full_join(), Это с dplyr v0.4
Слияние занимает ~17 секунд, full_join - ~65 секунд.
Впрочем, немного еды, так как я обычно по умолчанию использую dplyr для манипуляций.
- С помощью
mergeФункция, которую мы можем выбрать переменную левой или правой таблицы, точно так же, как мы все знакомы с оператором select в SQL (EX: выберите a.* ... или Select b.* из.....) Мы должны добавить дополнительный код, который будет подмножеством из вновь объединенной таблицы.
SQL:-
select a.* from df1 a inner join df2 b on a.CustomerId=b.CustomerIdР:-
merge(df1, df2, by.x = "CustomerId", by.y = "CustomerId")[,names(df1)]
Так же
SQL:-
select b.* from df1 a inner join df2 b on a.CustomerId=b.CustomerIdР:-
merge(df1, df2, by.x = "CustomerId", by.y = "CustomerId")[,names(df2)]
Для внутреннего объединения всех столбцов вы также можете использовать fintersect из пакета data.table или intersect из пакета dplyr в качестве альтернативы merge без указания by-columns. это даст строки, равные между двумя кадрами данных:
merge(df1, df2)
# V1 V2
# 1 B 2
# 2 C 3
dplyr::intersect(df1, df2)
# V1 V2
# 1 B 2
# 2 C 3
data.table::fintersect(setDT(df1), setDT(df2))
# V1 V2
# 1: B 2
# 2: C 3
Пример данных:
df1 <- data.frame(V1 = LETTERS[1:4], V2 = 1:4)
df2 <- data.frame(V1 = LETTERS[2:3], V2 = 2:3)
Обновление присоединиться. Еще одно важное соединение в стиле SQL- это " соединение обновления", при котором столбцы в одной таблице обновляются (или создаются) с использованием другой таблицы.
Изменение примеров таблиц OP...
sales = data.frame(
CustomerId = c(1, 1, 1, 3, 4, 6),
Year = 2000:2005,
Product = c(rep("Toaster", 3), rep("Radio", 3))
)
cust = data.frame(
CustomerId = c(1, 1, 4, 6),
Year = c(2001L, 2002L, 2002L, 2002L),
State = state.name[1:4]
)
sales
# CustomerId Year Product
# 1 2000 Toaster
# 1 2001 Toaster
# 1 2002 Toaster
# 3 2003 Radio
# 4 2004 Radio
# 6 2005 Radio
cust
# CustomerId Year State
# 1 2001 Alabama
# 1 2002 Alaska
# 4 2002 Arizona
# 6 2002 Arkansas
Предположим, мы хотим добавить состояние клиента из cust на стол покупок, sales, игнорируя столбец года. С помощью базы R мы можем определить совпадающие строки, а затем скопировать значения в:
sales$State <- cust$State[ match(sales$CustomerId, cust$CustomerId) ]
# CustomerId Year Product State
# 1 2000 Toaster Alabama
# 1 2001 Toaster Alabama
# 1 2002 Toaster Alabama
# 3 2003 Radio <NA>
# 4 2004 Radio Arizona
# 6 2005 Radio Arkansas
# cleanup for the next example
sales$State <- NULL
Как можно увидеть здесь, match выбирает первую подходящую строку из таблицы клиентов.
Обновите объединение с несколькими столбцами. Приведенный выше подход хорошо работает, когда мы объединяем только один столбец и удовлетворены первым соответствием. Предположим, мы хотим, чтобы год измерения в таблице клиентов соответствовал году продажи.
Как говорит ответ @ bgoldst, match с interaction может быть вариантом для этого случая. Проще говоря, можно использовать data.table:
library(data.table)
setDT(sales); setDT(cust)
sales[, State := cust[sales, on=.(CustomerId, Year), x.State]]
# CustomerId Year Product State
# 1: 1 2000 Toaster <NA>
# 2: 1 2001 Toaster Alabama
# 3: 1 2002 Toaster Alaska
# 4: 3 2003 Radio <NA>
# 5: 4 2004 Radio <NA>
# 6: 6 2005 Radio <NA>
# cleanup for next example
sales[, State := NULL]
Скользящее обновление присоединяется. В качестве альтернативы мы можем выбрать последнее состояние, в котором был найден клиент:
sales[, State := cust[sales, on=.(CustomerId, Year), roll=TRUE, x.State]]
# CustomerId Year Product State
# 1: 1 2000 Toaster <NA>
# 2: 1 2001 Toaster Alabama
# 3: 1 2002 Toaster Alaska
# 4: 3 2003 Radio <NA>
# 5: 4 2004 Radio Arizona
# 6: 6 2005 Radio Arkansas
Три приведенных выше примера фокусируются на создании / добавлении нового столбца. Смотрите соответствующий R FAQ для примера обновления / изменения существующего столбца.