Ошибки, выполняющие оценку максимального правдоподобия по трехпараметрическому Weibull cdf
Я работаю с кумулятивным появлением мух с течением времени (взятых с нерегулярными интервалами) в течение многих лет (хотя сначала я просто пытаюсь сделать один год работы). Кумулятивное возникновение следует сигмоидальному шаблону, и я хочу создать оценку максимального правдоподобия 3-параметрической кумулятивной функции распределения Вейбулла. Трехпараметрические модели, которые я пытался использовать в fitdistrplus Пакет продолжает давать мне ошибку. Я думаю, что это как-то связано с тем, как мои данные структурированы, но я не могу понять это. Очевидно, я хочу, чтобы он читал каждую точку как x (градусные дни) и y (Появление) значение, но, кажется, не может прочитать два столбца. Основная ошибка, которую я получаю, гласит: "Нечисловой аргумент математической функции" или (с немного другим кодом) "данные должны быть числовым вектором, длина которого больше 1". Ниже мой код, включая добавленные столбцы в df_dd_em рамки данных для совокупного появления и процента появления в случае, если это полезно.
degree_days <- c(998.08,1039.66,1111.29,1165.89,1236.53,1293.71,
1347.66,1387.76,1445.47,1493.44,1553.23,1601.97,
1670.28,1737.29,1791.94,1849.20,1920.91,1967.25,
2036.64,2091.85,2152.89,2199.13,2199.13,2263.09,
2297.94,2352.39,2384.03,2442.44,2541.28,2663.90,
2707.36,2773.82,2816.39,2863.94)
emergence <- c(0,0,0,1,1,0,2,3,17,10,0,0,0,2,0,3,0,0,1,5,0,0,0,0,
0,0,0,0,1,0,0,0,0,0)
cum_em <- cumsum(emergence)
df_dd_em <- data.frame (degree_days, emergence, cum_em)
df_dd_em$percent <- ave(df_dd_em$emergence, FUN = function(df_dd_em) 100*(df_dd_em)/46)
df_dd_em$cum_per <- ave(df_dd_em$cum_em, FUN = function(df_dd_em) 100*(df_dd_em)/46)
x <- pweibull(df_dd_em[c(1,3)],shape=5)
dframe2.mle <- fitdist(x, "weibull",method='mle')
2 ответа
Вот мое лучшее предположение о том, что вы ищете:
Настроить данные:
dd <- data.frame(degree_days=c(998.08,1039.66,1111.29,1165.89,1236.53,1293.71,
1347.66,1387.76,1445.47,1493.44,1553.23,1601.97,
1670.28,1737.29,1791.94,1849.20,1920.91,1967.25,
2036.64,2091.85,2152.89,2199.13,2199.13,2263.09,
2297.94,2352.39,2384.03,2442.44,2541.28,2663.90,
2707.36,2773.82,2816.39,2863.94),
emergence=c(0,0,0,1,1,0,2,3,17,10,0,0,0,2,0,3,0,0,1,5,0,0,0,0,
0,0,0,0,1,0,0,0,0,0))
dd <- transform(dd,cum_em=cumsum(emergence))
На самом деле мы собираемся соответствовать распределению с "цензурой по интервалу" (т. Е. Вероятности появления между последовательными наблюдениями дня степени: эта версия предполагает, что первое наблюдение относится к наблюдениям до наблюдения дня первой степени, вы можете изменить его так, чтобы оно соответствовало наблюдениям после последнего наблюдения).
library(bbmle)
## y*log(p) allowing for 0/0 occurrences:
y_log_p <- function(y,p) ifelse(y==0 & p==0,0,y*log(p))
NLLfun <- function(scale,shape,x=dd$degree_days,y=dd$emergence) {
prob <- pmax(diff(pweibull(c(-Inf,x), ## or (c(x,Inf))
shape=shape,scale=scale)),1e-6)
## multinomial probability
-sum(y_log_p(y,prob))
}
library(bbmle)
Возможно, мне следовало бы использовать что-то более систематическое, например, метод моментов (то есть сопоставление среднего и дисперсии распределения Вейбулла со средним и дисперсией данных), но я просто немного взломал, чтобы найти правдоподобные начальные значения:
## preliminary look (method of moments would be better)
scvec <- 10^(seq(0,4,length=101))
plot(scvec,sapply(scvec,NLLfun,shape=1))
Важно использовать parscale дать R знать, что параметры находятся в очень разных масштабах:
startvals <- list(scale=1000,shape=1)
m1 <- mle2(NLLfun,start=startvals,
control=list(parscale=unlist(startvals)))
Теперь попробуйте использовать Weibull с тремя параметрами (как первоначально требовалось) - требуется лишь небольшая модификация того, что у нас уже есть:
library(FAdist)
NLLfun2 <- function(scale,shape,thres,
x=dd$degree_days,y=dd$emergence) {
prob <- pmax(diff(pweibull3(c(-Inf,x),shape=shape,scale=scale,thres)),
1e-6)
## multinomial probability
-sum(y_log_p(y,prob))
}
startvals2 <- list(scale=1000,shape=1,thres=100)
m2 <- mle2(NLLfun2,start=startvals2,
control=list(parscale=unlist(startvals2)))
Похоже, трехпараметрическая подгонка намного лучше:
library(emdbook)
AICtab(m1,m2)
## dAIC df
## m2 0.0 3
## m1 21.7 2
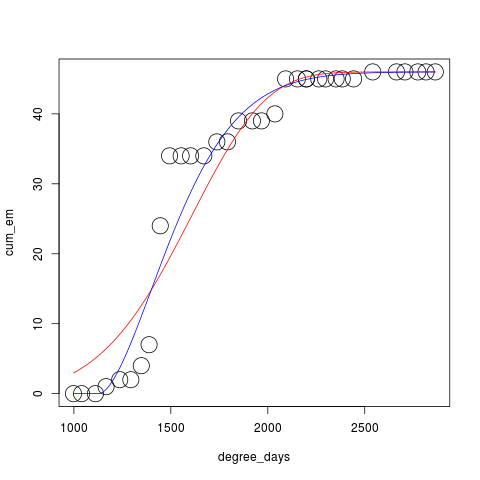
А вот графическое резюме:
with(dd,plot(cum_em~degree_days,cex=3))
with(as.list(coef(m1)),curve(sum(dd$emergence)*
pweibull(x,shape=shape,scale=scale),col=2,
add=TRUE))
with(as.list(coef(m2)),curve(sum(dd$emergence)*
pweibull3(x,shape=shape,
scale=scale,thres=thres),col=4,
add=TRUE))
(может также сделать это более элегантно с ggplot2...)
- Они не кажутся удивительно хорошими, но они нормальные. (В принципе, вы можете выполнить критерий соответствия по критерию хи-квадрат, основываясь на ожидаемом количестве аварийных ситуаций за интервал и учитывая тот факт, что вы выбрали трехпараметрическую модель, хотя значения могут быть немного низкими...)
- Доверительные интервалы по подгонке немного неприятны; ваш выбор (1) начальная загрузка; (2) параметрическая начальная загрузка (параметры повторной выборки, предполагающие многомерное нормальное распределение данных); (3) дельта-метод.
- С помощью
bbmle::mle2позволяет легко делать такие вещи, как получение доверительных интервалов профиля:
confint(m1)
## 2.5 % 97.5 %
## scale 1576.685652 1777.437283
## shape 4.223867 6.318481
dd <- data.frame(degree_days=c(998.08,1039.66,1111.29,1165.89,1236.53,1293.71,
1347.66,1387.76,1445.47,1493.44,1553.23,1601.97,
1670.28,1737.29,1791.94,1849.20,1920.91,1967.25,
2036.64,2091.85,2152.89,2199.13,2199.13,2263.09,
2297.94,2352.39,2384.03,2442.44,2541.28,2663.90,
2707.36,2773.82,2816.39,2863.94),
emergence=c(0,0,0,1,1,0,2,3,17,10,0,0,0,2,0,3,0,0,1,5,0,0,0,0,
0,0,0,0,1,0,0,0,0,0))
dd$cum_em <- cumsum(dd$emergence)
dd$percent <- ave(dd$emergence, FUN = function(dd) 100*(dd)/46)
dd$cum_per <- ave(dd$cum_em, FUN = function(dd) 100*(dd)/46)
dd <- transform(dd)
#start 3 parameter model
library(FAdist)
## y*log(p) allowing for 0/0 occurrences:
y_log_p <- function(y,p) ifelse(y==0 & p==0,0,y*log(p))
NLLfun2 <- function(scale,shape,thres,
x=dd$degree_days,y=dd$percent) {
prob <- pmax(diff(pweibull3(c(-Inf,x),shape=shape,scale=scale,thres)),
1e-6)
## multinomial probability
-sum(y_log_p(y,prob))
}
startvals2 <- list(scale=1000,shape=1,thres=100)
m2 <- mle2(NLLfun2,start=startvals2,
control=list(parscale=unlist(startvals2)))
summary(m2)
#graphical summary
windows(5,5)
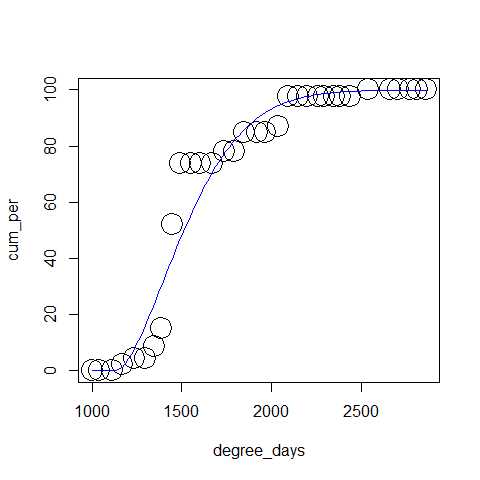
with(dd,plot(cum_per~degree_days,cex=3))
with(as.list(coef(m2)),curve(sum(dd$percent)*
pweibull3(x,shape=shape,
scale=scale,thres=thres),col=4,
add=TRUE))