R Shiny ggiraph и d3heatmap Проблемы совместимости
Я пытаюсь добавить интерактивную тепловую карту в мое приложение Shiny, но у меня также есть интерактивные графики с использованием ggiraph. В настоящее время я использую пакет d3heatmap, но тепловые карты не отображаются в приложении. Я создал игрушечный пример, чтобы проиллюстрировать это:
library(shiny)
library(ggiraph)
library(d3heatmap)
ui <- fluidPage(
d3heatmapOutput('d3'),
ggiraphOutput('gg')
)
server <- function(input, output, session) {
# Create heatmap
output$d3 <- renderD3heatmap({
d3heatmap(matrix(1:100, nrow = 100, ncol = 100))
})
# Create ggiraph
output$gg <- renderggiraph({
p <- ggplot(iris, aes(x = Sepal.Length, y = Petal.Width,
color = Species, tooltip = iris$Species) ) +
geom_point_interactive()
ggiraph(code = {print(p)})
})
}
shinyApp(ui = ui, server = server)
Вместе рендерится только ggiraph, а тепловая карта - нет. Однако, если вы закомментируете код ggiraph, тепловая карта будет отображаться. Я попытался изменить порядок загрузки пакетов, но это все равно не сработало.
В настоящее время я работаю на R 3.2.2 (я должен использовать эту версию, потому что серверы компании работают только на этой версии, и ни у моего менеджера, ни у меня нет прав на ее обновление). Я попытался загрузить пакеты блестящие карты, heatmaply и heatmap.2, но из-за проблем с версией установки не увенчались успехом.
Итак, прямо сейчас, я только что использовал pheatmap для создания тепловых карт, но они не интерактивны (то есть я не могу получить значения при наведении курсора на отдельные ячейки и не могу увеличить масштаб). Есть ли обходной путь для этого, или есть другие интерактивные пакеты тепловых карт, которые будут работать? Я хотел бы избежать изменения всех моих графиков ggiraph на графические графики, поскольку их много в моем коде.
Пожалуйста, дайте мне знать, если вам нужна какая-либо другая информация. Любые предложения будут высоко ценится!
1 ответ
(просто чтобы вы знали, что я автор ggiraph) Существует конфликт между ggiraph и d3heatmap, потому что ggiraph использует d3.js версии 4, а d3heatmap использует D3.js версии 3. Я не думаю, что есть решение для разрешения этого конфликта.
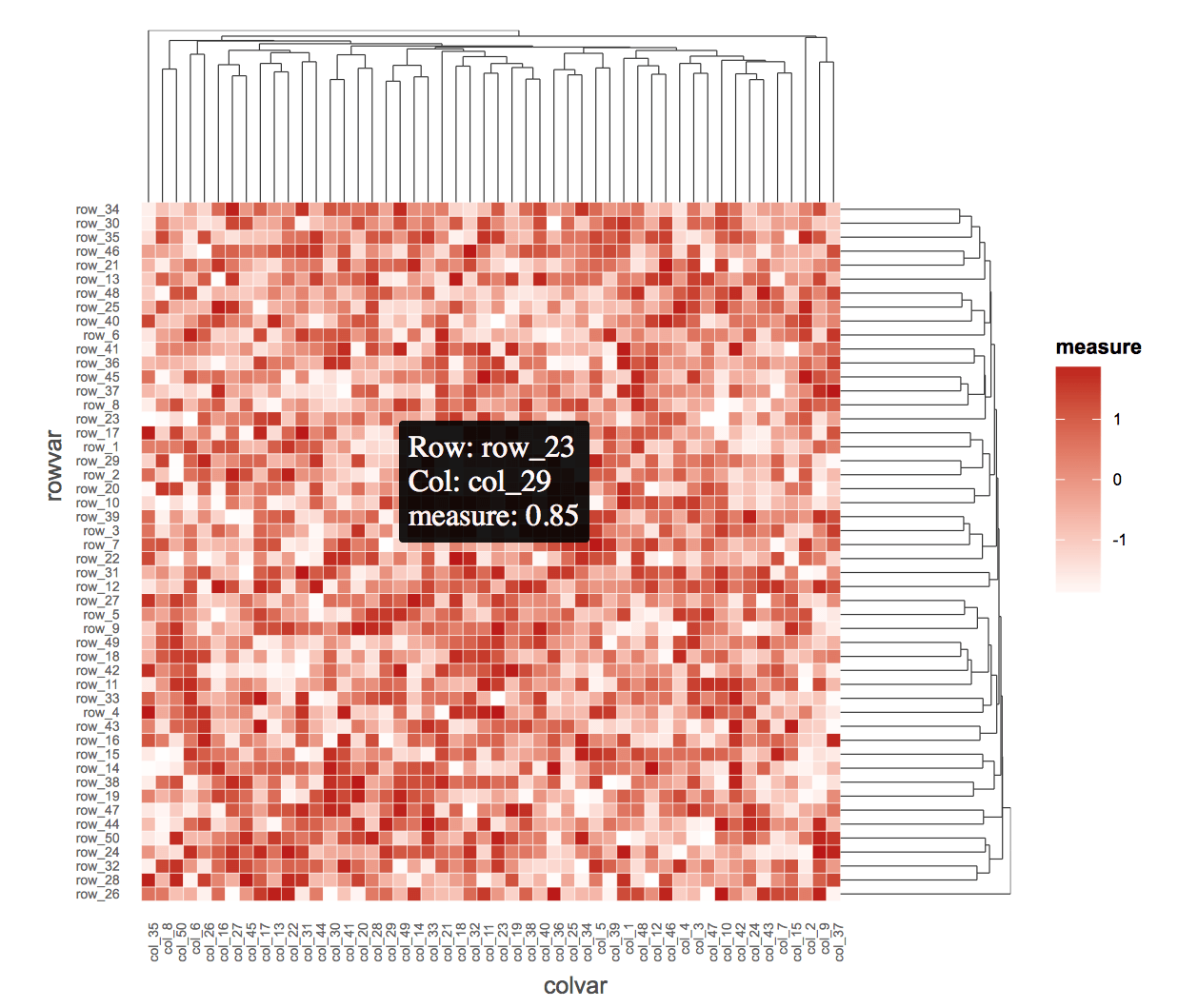
Однако создать интерактивную тепловую карту с помощью ggplot2/ggiraph не так уж и сложно. Увидеть ниже:
library(dplyr)
library(tidyr)
library(ggplot2)
library(ggiraph)
library(ggdendro)
# mydata <- cor(mtcars)
mydata <- matrix(runif(2500, min = -2, max = 2), ncol = 50)
row.names(mydata) <- paste0("row_", seq_len(nrow(mydata)))
colnames(mydata) <- paste0("col_", seq_len(ncol(mydata)))
# dendrogram for rows
hc <- hclust(dist(mydata), "ave")
dhr <- as.dendrogram(hc)
order_r <- rownames(mydata)[hc$order]
# dendrogram for columns
hc <- hclust(dist(t(mydata)), "ave")
dhc <- as.dendrogram(hc)
order_c <- colnames(mydata)[hc$order]
# the data
expr_set <- bind_cols(
data_frame(rowvar = rownames(mydata)),
as.data.frame(mydata)
)
expr_set <- gather(expr_set, colvar, measure, -rowvar)
expr_set$rowvar <- factor( expr_set$rowvar, levels = order_r )
expr_set$colvar <- factor( expr_set$colvar, levels = order_c )
expr_set <- arrange(expr_set, rowvar, colvar)
# get data for dendrograms - IMHO, ggdendro is the hero here...
data_c <- dendro_data(dhc, type = "rectangle")
data_c <- segment(data_c) %>% mutate(
y = y + length(order_r) + .5,
yend = yend + length(order_r) + .5
)
data_r <- dendro_data(dhr, type = "rectangle")
data_r <- segment(data_r)
data_r <- data_r %>%
mutate( x_ = y + length(order_c) + .5,
xend_ = yend + length(order_c) + .5,
y_ = x,
yend_ = xend )
expr_set <- expr_set %>%
mutate(
tooltip = sprintf("Row: %s<br/>Col: %s<br/>measure: %.02f",
rowvar, colvar, measure) ,
data_id = sprintf("%s_%s", rowvar, colvar)
)
# all data are tidy and can be now used with ggplot
p <- ggplot(data = expr_set, aes(x = colvar, y = rowvar) ) +
geom_tile_interactive(aes(fill = measure, tooltip = tooltip, data_id = data_id), colour = "white") +
scale_fill_gradient(low = "white", high = "#BC120A") +
geom_segment(
data = data_c,
mapping = aes(x = x, y = yend, xend = xend, yend = y),
colour = "gray20", size = .2) +
geom_segment(
data = data_r,
mapping = aes(x = x_, y = y_, xend = xend_, yend = yend_),
colour = "gray20", size = .2) +
coord_equal()
# cosmetics
p <- p + theme_minimal() +
theme(
legend.position = "right",
panel.grid.minor = element_line(color = "transparent"),
panel.grid.major = element_line(color = "transparent"),
axis.ticks.length = unit(2, units = "mm"),
plot.title = element_text(face = "bold", hjust = 0.5, size = 12),
axis.title = element_text(size = 9, colour = "gray30"),
axis.text.y = element_text(hjust = 1, size = 5, colour = "gray40"),
axis.text.x = element_text(angle = 90, hjust = 1, size = 5, colour = "gray40"),
legend.title=element_text(face = "bold", hjust = 0.5, size=8),
legend.text=element_text(size=6)
)
ggiraph(ggobj = p)
Надеюсь, поможет
Я знаю, что некоторое время назад на этот вопрос был дан ответ, но я столкнулся с той же проблемой, и я не смог использовать ggplot2 потому что работать с моими Shinyзаявление. Вheatmaplyпакет выделяется быстрее и проще в реализации. Я выполнил мини-тест (n= 20). сggplot2заняло в среднем 64 секунды. Сheatmaplyэто заняло всего 2 секунды. оба метода используют'ave' метод hclust. Надеюсь, это полезно.
мини-тест n = 20 для ggplot vs heatmaply
вот код, который я использовал:
library(tidyr)
library(ggplot2)
library(ggiraph)
library(ggdendro)
library(heatmaply)
# mydata <- cor(mtcars)
create_data <- function(){
df <- matrix(runif(2500, min = -2, max = 2), ncol = 50)
row.names(df) <- paste0("row_", seq_len(nrow(df)))
colnames(df) <- paste0("col_", seq_len(ncol(df)))
return(df)
}
gg2heat <- function(mydata){
# dendrogram for rows
hc <- hclust(dist(mydata), "ave")
dhr <- as.dendrogram(hc)
order_r <- rownames(mydata)[hc$order]
# dendrogram for columns
hc <- hclust(dist(t(mydata)), "ave")
dhc <- as.dendrogram(hc)
order_c <- colnames(mydata)[hc$order]
# the data
expr_set <- bind_cols(
data_frame(rowvar = rownames(mydata)),
as.data.frame(mydata)
)
expr_set <- gather(expr_set, colvar, measure, -rowvar)
expr_set$rowvar <- factor( expr_set$rowvar, levels = order_r )
expr_set$colvar <- factor( expr_set$colvar, levels = order_c )
expr_set <- arrange(expr_set, rowvar, colvar)
# get data for dendrograms - IMHO, ggdendro is the hero here...
data_c <- dendro_data(dhc, type = "rectangle")
data_c <- segment(data_c) %>% mutate(
y = y + length(order_r) + .5,
yend = yend + length(order_r) + .5
)
data_r <- dendro_data(dhr, type = "rectangle")
data_r <- segment(data_r)
data_r <- data_r %>%
mutate( x_ = y + length(order_c) + .5,
xend_ = yend + length(order_c) + .5,
y_ = x,
yend_ = xend )
expr_set <- expr_set %>%
mutate(
tooltip = sprintf("Row: %s<br/>Col: %s<br/>measure: %.02f",
rowvar, colvar, measure) ,
data_id = sprintf("%s_%s", rowvar, colvar)
)
# all data are tidy and can be now used with ggplot
p <- ggplot(data = expr_set, aes(x = colvar, y = rowvar) ) +
geom_tile_interactive(aes(fill = measure, tooltip = tooltip, data_id = data_id), colour = "white") +
scale_fill_gradient(low = "white", high = "#BC120A") +
geom_segment(
data = data_c,
mapping = aes(x = x, y = yend, xend = xend, yend = y),
colour = "gray20", size = .2) +
geom_segment(
data = data_r,
mapping = aes(x = x_, y = y_, xend = xend_, yend = yend_),
colour = "gray20", size = .2) +
coord_equal()
# cosmetics
p <- p + theme_minimal() +
theme(
legend.position = "right",
panel.grid.minor = element_line(color = "transparent"),
panel.grid.major = element_line(color = "transparent"),
axis.ticks.length = unit(2, units = "mm"),
plot.title = element_text(face = "bold", hjust = 0.5, size = 12),
axis.title = element_text(size = 9, colour = "gray30"),
axis.text.y = element_text(hjust = 1, size = 5, colour = "gray40"),
axis.text.x = element_text(angle = 90, hjust = 1, size = 5, colour = "gray40"),
legend.title=element_text(face = "bold", hjust = 0.5, size=8),
legend.text=element_text(size=6)
)
ggiraph(ggobj = p)
}
htmp_gg <- c()
htmp_maply <-c()
for (i in 1:20){
df <- create_data()
time_gg <- (system.time(gg2heat(df)))[3]
htmp_gg<- append(htmp_gg, values = time_gg)
time_heatmaply <- (system.time(heatmaply::heatmaply(df, hclust_method = 'ave')))[3]
htmp_maply<- append(htmp_maply, values = time_heatmaply)
rm(df)
}
score <- data.frame(htmp_gg, htmp_maply)%>% gather(key = 'method', value = 'time')
p <- ggplot(score, aes(x = method, y = time, fill = method))+geom_violin()+ stat_summary(fun.y=median, geom="point", size=2, color="black")
print(p)