Как предотвратить неточную сегментацию замкнутых фоновых областей в алгоритме водораздела?
Я использую алгоритм водораздела, чтобы сегментировать яркие пятна на темном фоне. Код приведен ниже вместе с некоторыми изображениями, которые он генерирует.
На втором изображении я пометил красными крестиками области закрытого фона, которые сегментированы как "клетки" (это не биологические клетки, просто слово) - это неправильно, они являются частью фона, просто заключенный в "клетки". Я вижу, что это создает ложный минимум, любая помощь о том, как предотвратить это?
% Improve contrast, binarize
RFP_adjust = imadjust(RFP_blur, stretchlim(RFP_blur, 0.001));
figure, imshow(RFP_adjust), title('Contrast adjust');
RFP_binarized = imbinarize(RFP_adjust);
RFP_perimeters = bwperim(RFP_binarized);
% figure, imshow(RFP_binarized), title('Otsu thresholding');
%2B - SEGMENTATION BY WATERSHED METHOD
% Discover putative cell centroids and process
RFP_maxs = imextendedmax(RFP_adjust, 3000);
RFP_maxs = imclose(RFP_maxs, strel('disk',5));
RFP_maxs = imfill(RFP_maxs, 'holes');
RFP_maxs = bwareaopen(RFP_maxs, 5);
RFP_max_overlay = imoverlay(RFP_adjust, RFP_perimeters | RFP_maxs, [1 .3 .3]);
figure, imshow(RFP_max_overlay), title('Maxima');
% Obtain complement - maxima become low-points (required for watershed)
RFP_comp = imcomplement(RFP_adjust);
RFP_imposemin = imimposemin(RFP_comp, ~RFP_binarized | RFP_maxs);
figure, imshow(RFP_imposemin), title('Inverted Maxima');
% Apply watershed
RFP_watershed = watershed(RFP_imposemin);
mask = im2bw(RFP_watershed, 1);
overlay3 = imoverlay(RFP_adjust, mask, [1 .3 .3]);
figure, imshow(overlay3), title('Segmented cells');
% Segment
RFP_cc = bwconncomp(RFP_watershed);
RFP_label_matrix = labelmatrix(RFP_cc);
whos labeled;
RFP_label = label2rgb(RFP_label_matrix, @spring, 'c', 'shuffle');
figure, imshow(RFP_label), title('Cells segmented');
Изображение 0 - результат для изображения с названием "Максима" (т.е. откорректированное исходное изображение с наложенными максимумами и контурами). 
Изображение 1 - результат для изображения с названием 'inverted maxima' 
Изображение 2 - результат для изображения с названием 'Cells segmented' 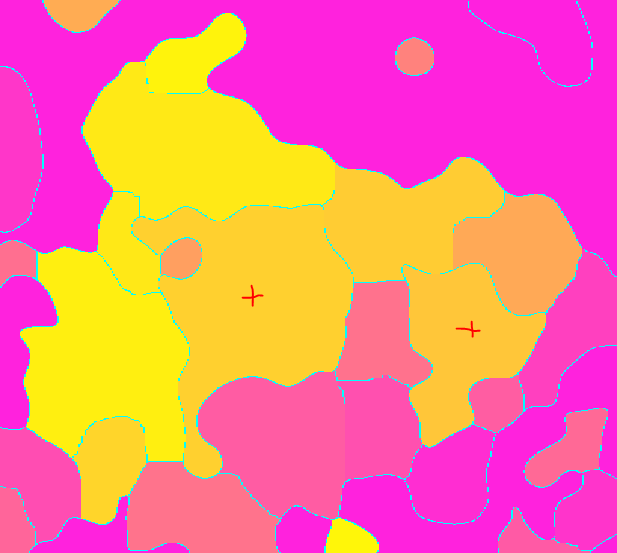
2 ответа
Я хотел бы предложить что-то вроде того, что сделано в примере, включенном для watershed Функция: используйте маску фона, чтобы установить эти пиксели Inf, выполните операцию водораздела, затем установите фоновые пиксели в результате равными 0. Я полагаю, что вы могли бы изменить секцию водораздела своего кода следующим образом:
% Apply watershed
RFP_watershed = RFP_imposemin; % Added
RFP_watershed(~RFP_binarized) = Inf; % Added
RFP_watershed = watershed(RFP_watershed); % Modified
RFP_watershed(~RFP_binarized) = 0; % Added
mask = im2bw(RFP_watershed, 1);
overlay3 = imoverlay(RFP_adjust, mask, [1 .3 .3]);
figure, imshow(overlay3), title('Segmented cells');
Там нет волшебной пули, но есть несколько вещей, которые вы можете попробовать.
Одним из них является фильтрация изображения с очень большим круглым диском, создавая размытое изображение, похожее на фон. Затем вычтите его из исходного изображения. Это будет стремиться заставить фактический фон к нулю.
Другим является отсечение порога для отделения переднего плана от фона. Это создает двоичное изображение. Затем выполните морфологическую операцию открытия, используя маску, разработанную так, чтобы она выглядела как настоящие клетки.