Распечатать "красивые" таблицы для моделей H2O в R
Есть несколько пакетов для R которые помогают печатать "красивые" таблицы (LaTeX/HTML/TEXT) из статистических моделей и легко сравнивать результаты спецификаций альтернативных моделей.
Некоторые из этих пакетов apsrtable, xtable, memisc, texreg, outreg, а также stargazer (примеры см. здесь: https://www.r-statistics.com/2013/01/stargazer-package-for-beautiful-latex-tables-from-r-statistical-models-output/).
Есть ли сопоставимые R пакет, который поддерживает модели из h2o пакет?
Вот пример двух простых моделей GLM с h2o которые я люблю печатать рядом друг с другом как "красивые" столы.
# Load package and setup h2o
library(h2o)
localH2O <- h2o.init(ip = 'localhost', port = 54321, max_mem_size = '4g')
# Load data
prostatePath <- system.file("extdata", "prostate.csv", package = "h2o")
prostate.hex <- h2o.importFile(path = prostatePath, destination_frame = "prostate.hex")
# Run GLMs
model.output.1 <- h2o.glm(y = "CAPSULE", x = c("RACE","PSA","DCAPS"),
training_frame = prostate.hex,family = "binomial", nfolds = 0,
alpha = 0.5, lambda_search = FALSE)
model.output.2 <- h2o.glm(y = "CAPSULE", x = c("AGE","RACE","PSA","DCAPS"),
training_frame = prostate.hex, family = "binomial", nfolds = 0,
alpha = 0.5, lambda_search = FALSE)
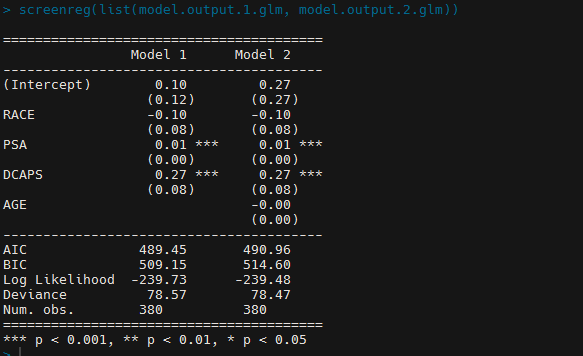
Вот как это будет выглядеть с обычным объектом GLM, использующим screenreg() от texreg пакет:
library(data.table)
library(texreg)
d <- fread(prostatePath)
model.output.1.glm <- glm(CAPSULE ~ RACE + PSA + DCAPS, data=d)
model.output.2.glm <- glm(CAPSULE ~ AGE + RACE + PSA + DCAPS, data=d)
screenreg(list(model.output.1.glm, model.output.2.glm))
3 ответа
texreg Автор пакета здесь. texreg основан на общих функциях, что означает, что любой пользователь может добавить пользовательский extract методы для произвольных моделей и заставить их работать с texreg, Я проведу вас через это ниже. Я надеюсь, что эта подробная экспозиция поможет другим людям, которые задавали подобные вопросы в прошлом, разработать свои собственные texreg расширения.
См. Также раздел 6 в статье 2013 года в журнале статистического программного обеспечения для другого примера. Однако сначала я опишу, как texreg архитектура работает более широко, чтобы дать вам представление о том, что возможно.
texreg и общий extract функция
Есть три функции: texreg (для вывода LaTeX), htmlreg (для вывода HTML, который также может интерпретироваться Word или Markdown в большинстве сценариев использования), и screenreg (для вывода текста ASCII в консоли). Эти три функции служат для преобразования некоторой очищенной информации (коэффициенты, стандартные ошибки, доверительные интервалы, p-значения, статистика соответствия), метки модели и т. Д.) В соответствующие выходные форматы. Это не идеально и может быть даже более гибким, но я думаю, что на данный момент у него довольно много аргументов для настройки, включая такие вещи, как booktabs а также dcolumn служба поддержки. Таким образом, большая проблема заключается в том, чтобы сначала получить очищенную информацию о модели.
Это делается путем предоставления texreg возражать против любой из этих трех функций. texreg Объект является просто контейнером для коэффициентов и т. д. и формально определяется с использованием класса S4. Чтобы создать texreg объект, вы можете использовать функцию конструктора createTexreg (как задокументировано на страницах справки), которая принимает все различные части информации в качестве аргументов, такие как стандартные ошибки и т. д. Или (лучше) вы можете использовать extract функция для извлечения этих частей информации из некоторой оценочной модели и возврата texreg объект для использования с любой из трех функций. Обычно вы делаете это, просто передавая список нескольких моделей texreg или же screenreg и т. д., и эта функция будет вызывать внутренне extract создавать texreg объекты, а затем обрабатывать информацию из этих объектов.
Тем не менее, одинаково справедливо просто сохранить результат вызова extract функция к объекту, возможно, манипулировать этим texreg объект, а затем вызвать texreg функция на управляемом объекте для отображения его в виде таблицы. Это дает некоторую гибкость в настройке результатов.
Ранее я упоминал, что пакет использует универсальные функции. Это означает, что extract Функция является общей в том смысле, что для нее можно зарегистрировать методы, которые работают с произвольными классами моделей. Например, если extract функция не умеет обращаться h2o объекты и как извлечь соответствующую информацию из такого объекта, вы можете просто написать метод для этого и зарегистрировать его с extract функция. Ниже я проведу вас через этот шаг за шагом в надежде, что люди извлекут уроки из этого подробного изложения и начнут писать свои собственные расширения. (Примечание: если кто-то разработает полезный метод, пожалуйста, напишите мне, и я могу включить его в следующий texreg выпуск.) Также стоит отметить, что исходные файлы пакета содержат более 70 примеров extract методы, которые вы можете использовать в качестве шаблонов. Эти примеры хранятся в файле R/extract.R,
Идентификация метки класса и настройка extract метод
Первым шагом является определение имени класса объекта. В вашем примере class(model.output.1) возвращает следующие метки классов: "H2OBinomialModel" и "h2o". Первый ярлык является более конкретным, а второй - более общим. Я упал h2o объекты модели структурированы подобным образом, имеет смысл написать расширение для h2o объекты, а затем решить в рамках метода, как поступить с конкретной моделью. Как я практически ничего не знаю о h2o пакет, я предпочитаю начать с более конкретной extract метод для H2OBinomialModel объекты в этом случае. Это может быть скорректировано позже, если мы захотим это сделать.
extract методы структурированы следующим образом: вы пишете функцию с именем extract.xyz (замените "xyz" на метку класса), у вас должен быть хотя бы один аргумент model, который принимает объект модели (например, model.output.1 в вашем примере), поместите некоторый код в тело, которое извлекает соответствующую информацию из model объект, создать texreg объект с помощью createTexreg конструктор, и вернуть этот объект. Вот пустой контейнер:
extract.H2OBinomialModel <- function(model, ...) {
s <- summary(model)
# extract information from model and summary object here
# then create and return a texreg object (replace NULL with actual values):
tr <- createTexreg(
coef.names = NULL, # character vector of coefficient labels
coef = NULL, # numeric vector with coefficients
se = NULL, # numeric vector with standard error values
pvalues = NULL, # numeric vector with p-values
gof.names = NULL, # character vector with goodness-of-fit labels
gof = NULL, # numeric vector of goodness-of-fit statistics
gof.decimal = NULL # logical vector: GOF statistic has decimal points?
)
return(tr)
}
Обратите внимание, что определение функции также содержит ... аргумент, который можно использовать для пользовательских аргументов, которые должны быть переданы вызовам функций внутри extract метод.
Также обратите внимание, что первая строка в теле определения функции сохраняет сводку модели в объекте с именем s, Это часто полезно, потому что многие разработчики пакетов решили хранить некоторую информацию в более простой версии в сводке, поэтому обычно следует рассматривать и модель, и ее сводку как полезные источники информации. В некоторых случаях, возможно, придется взглянуть на фактическое определение метода сводки в соответствующем пакете, чтобы узнать, как вычисляются фрагменты информации, отображаемые на странице сводки, когда summary Команда называется потому что не все summary методы хранят различные элементы, отображаемые в summary объект.
Нахождение правильной информации в H2OBinomialModel объект
Следующим шагом является проверка объекта и поиск всех деталей, которые должны отображаться в итоговой таблице. Глядя на вывод model.output.1 Я бы предположил, что следующая часть должна составлять блок GOF внизу таблицы:
MSE: 0.202947
R^2: 0.1562137
LogLoss: 0.5920097
Mean Per-Class Error: 0.3612191
AUC: 0.7185655
Gini: 0.4371311
Null Deviance: 512.2888
Residual Deviance: 449.9274
AIC: 457.9274
И следующая часть, вероятно, должна составлять блок коэффициентов в середине таблицы:
Coefficients: glm coefficients
names coefficients standardized_coefficients
1 Intercept -1.835223 -0.336428
2 RACE -0.625222 -0.193052
3 DCAPS 1.314428 0.408336
4 PSA 0.046861 0.937107
Во многих случаях резюме содержит соответствующую информацию, но здесь печать модели дает то, что нам нужно. Нам нужно будет найти все это в model.output.1 объект (или его краткое изложение, если применимо). Для этого есть несколько полезных команд. Среди них есть str(model.output.1), names(summary(model.output.1)) и аналогичные команды.
Начнем с блока коэффициентов. призвание str(model) показывает, что есть слот под названием model в объекте S4. Мы можем посмотреть на его содержание, позвонив model.output.1@model, В результате получается список с несколькими именованными элементами, среди которых coefficients_table, Таким образом, мы можем получить доступ к таблице коэффициентов, вызвав model.output.1@model$coefficients_table, Результатом является фрейм данных, к столбцам которого мы можем получить доступ, используя $ оператор. В частности, нам нужны имена и коэффициенты. Здесь есть два типа коэффициентов, стандартизированные и нестандартные, и мы можем добавить аргумент к нашему методу извлечения позже, чтобы позволить пользователю решить, что он или она хочет. Вот как мы извлекаем коэффициенты и их метки:
coefnames <- model.output.1@model$coefficients_table$names
coefs <- model.output.1@model$coefficients_table$coefficients
coefs.std <- model.output.1@model$coefficients_table$standardized_coefficients
Насколько я вижу, в объекте не хранятся стандартные ошибки или p-значения. Мы могли бы написать дополнительный код для их вычисления, если захотим, но здесь мы сосредоточимся на вещах, которые легко предоставляются как часть вывода модели.
Важно, чтобы мы не перезаписывали имена существующих функций в R, такие как names или же coef, Хотя это технически должно работать, потому что код выполняется внутри функции позже, это может легко привести к путанице при испытании, поэтому вам лучше этого избежать.
Далее нам нужно найти статистику соответствия. Изучая результаты str(model.output.1) тщательно, мы видим, что статистика соответствия пригодности содержится в нескольких слотах под model@model$training_metrics@metrics, Давайте сохраним их в некоторых объектах, к которым проще получить доступ:
mse <- model.output.1@model$training_metrics@metrics$MSE
r2 <- model.output.1@model$training_metrics@metrics$r2
logloss <- model.output.1@model$training_metrics@metrics$logloss
mpce <- model.output.1@model$training_metrics@metrics$mean_per_class_error
auc <- model.output.1@model$training_metrics@metrics$AUC
gini <- model.output.1@model$training_metrics@metrics$Gini
nulldev <- model.output.1@model$training_metrics@metrics$null_deviance
resdev <- model.output.1@model$training_metrics@metrics$residual_deviance
aic <- model.output.1@model$training_metrics@metrics$AIC
В некоторых случаях, но не здесь, автор пакета пишет методы для универсальных функций, которые можно использовать для извлечения некоторой общей информации, например количества наблюдений (nobs(model)), АПК (AIC(model)), БИК (BIC(model)), отклонение (deviance(model)) или журнал вероятности (logLik(model)[[1]]). Так что это то, что вы можете попробовать в первую очередь; но h2o пакет, кажется, не предлагает такие удобные методы.
Добавление информации в extract.H2OBinomialModel функция
Теперь, когда мы нашли всю необходимую нам информацию, мы можем добавить их в extract.H2OBinomialModel Функция, которую мы определили выше.
Тем не менее, мы хотели бы позволить пользователю решать, предпочитает ли он или она необработанные или стандартизированные коэффициенты, и мы хотим, чтобы пользователь решал, какую статистику о соответствии качества следует сообщать, поэтому мы добавляем различные логические аргументы в заголовок функции. а затем использовать if-условия внутри функции, чтобы проверить, следует ли встраивать соответствующую статистику в texreg объект.
Мы также удаляем строку s <- summary(model) в этом случае, потому что нам на самом деле не нужна какая-либо информация из сводки, поскольку мы нашли все, что нам нужно, в объекте модели.
Полная функция может выглядеть так:
# extension for H2OBinomialModel objects (h2o package)
extract.H2OBinomialModel <- function(model, standardized = FALSE,
include.mse = TRUE, include.rsquared = TRUE, include.logloss = TRUE,
include.meanerror = TRUE, include.auc = TRUE, include.gini = TRUE,
include.deviance = TRUE, include.aic = TRUE, ...) {
# extract coefficient table from model:
coefnames <- model@model$coefficients_table$names
if (standardized == TRUE) {
coefs <- model@model$coefficients_table$standardized_coefficients
} else {
coefs <- model@model$coefficients_table$coefficients
}
# create empty GOF vectors and subsequently add GOF statistics from model:
gof <- numeric()
gof.names <- character()
gof.decimal <- logical()
if (include.mse == TRUE) {
mse <- model@model$training_metrics@metrics$MSE
gof <- c(gof, mse)
gof.names <- c(gof.names, "MSE")
gof.decimal <- c(gof.decimal, TRUE)
}
if (include.rsquared == TRUE) {
r2 <- model@model$training_metrics@metrics$r2
gof <- c(gof, r2)
gof.names <- c(gof.names, "R^2")
gof.decimal <- c(gof.decimal, TRUE)
}
if (include.logloss == TRUE) {
logloss <- model@model$training_metrics@metrics$logloss
gof <- c(gof, logloss)
gof.names <- c(gof.names, "LogLoss")
gof.decimal <- c(gof.decimal, TRUE)
}
if (include.meanerror == TRUE) {
mpce <- model@model$training_metrics@metrics$mean_per_class_error
gof <- c(gof, mpce)
gof.names <- c(gof.names, "Mean Per-Class Error")
gof.decimal <- c(gof.decimal, TRUE)
}
if (include.auc == TRUE) {
auc <- model@model$training_metrics@metrics$AUC
gof <- c(gof, auc)
gof.names <- c(gof.names, "AUC")
gof.decimal <- c(gof.decimal, TRUE)
}
if (include.gini == TRUE) {
gini <- model@model$training_metrics@metrics$Gini
gof <- c(gof, gini)
gof.names <- c(gof.names, "Gini")
gof.decimal <- c(gof.decimal, TRUE)
}
if (include.deviance == TRUE) {
nulldev <- model@model$training_metrics@metrics$null_deviance
resdev <- model@model$training_metrics@metrics$residual_deviance
gof <- c(gof, nulldev, resdev)
gof.names <- c(gof.names, "Null Deviance", "Residual Deviance")
gof.decimal <- c(gof.decimal, TRUE, TRUE)
}
if (include.aic == TRUE) {
aic <- model@model$training_metrics@metrics$AIC
gof <- c(gof, aic)
gof.names <- c(gof.names, "AIC")
gof.decimal <- c(gof.decimal, TRUE)
}
# create texreg object:
tr <- createTexreg(
coef.names = coefnames,
coef = coefs,
gof.names = gof.names,
gof = gof,
gof.decimal = gof.decimal
)
return(tr)
}
Вы можете видеть, что для блока добротности соответствия я сначала создал пустые векторы, а затем заполнил их дополнительной статистикой при условии, что соответствующая статистика была включена пользователем с использованием соответствующего аргумента.
gof.decimal логический вектор указывает для каждой статистики GOF, имеет ли она десятичные разряды (TRUE) или нет (FALSE например, по количеству наблюдений).
Наконец, новая функция должна быть зарегистрирована как метод для общего extract функция. Это делается с помощью простой команды:
setMethod("extract", signature = className("H2OBinomialModel", "h2o"),
definition = extract.H2OBinomialModel)
Здесь первый аргумент className это метка класса, а вторая - это пакет, в котором определен класс.
Подводя итог, единственные две вещи, которые необходимо сделать, чтобы написать собственное расширение, - это 1) написание метода извлечения и 2) регистрация метода. Таким образом, этот код может быть выполнен во время выполнения и не должен быть вставлен ни в какой пакет.
Однако для вашего удобства я добавил H2OBinomialModel метод для texreg версия 1.36.13, которая доступна на CRAN.
Обратите внимание, что представленное здесь решение не работает ни с какой предыдущей версией texreg потому что предыдущие версии не могли иметь дело с моделями, которые не имели ни стандартных ошибок, ни доверительных интервалов. По моему мнению, это довольно специализированная установка, и я не сталкивался с пакетом, который просто предоставляет оценки без каких-либо мер неопределенности. Я сейчас исправил это в texreg,
Опробовать новый extract метод
После определения функции и setMethod Команда была выполнена во время выполнения, для создания таблицы можно использовать следующую команду:
screenreg(list(model.output.1, model.output.2), custom.note = "")
Это вывод:
======================================
Model 1 Model 2
--------------------------------------
Intercept -1.84 -1.11
RACE -0.63 -0.62
DCAPS 1.31 1.31
PSA 0.05 0.05
AGE -0.01
--------------------------------------
MSE 0.20 0.20
R^2 0.16 0.16
LogLoss 0.59 0.59
Mean Per-Class Error 0.36 0.38
AUC 0.72 0.72
Gini 0.44 0.44
Null Deviance 512.29 512.29
Residual Deviance 449.93 449.51
AIC 457.93 459.51
======================================
custom.note = "" Аргумент здесь имеет смысл, потому что мы не хотим легенду о значимости, поскольку модели не сообщают о каких-либо показателях неопределенности
Также возможно исключить некоторые из мер GOF и / или использовать стандартизированные коэффициенты:
screenreg(list(model.output.1, model.output.2), custom.note = "",
include.deviance = FALSE, include.auc = FALSE, standardized = TRUE)
Результат:
======================================
Model 1 Model 2
--------------------------------------
Intercept -0.34 -0.34
RACE -0.19 -0.19
DCAPS 0.41 0.41
PSA 0.94 0.94
AGE -0.07
--------------------------------------
MSE 0.20 0.20
R^2 0.16 0.16
LogLoss 0.59 0.59
Mean Per-Class Error 0.36 0.38
Gini 0.44 0.44
AIC 457.93 459.51
======================================
Другие слоты, которые можно использовать с createTexreg
createTexreg Функция конструктора вызывается в любом extract метод. В приведенном выше примере показаны некоторые простые фрагменты информации. Помимо деталей, содержащихся в примере, в слоте доступны следующие слоты. texreg объекты:
- se: стандартные ошибки
- pvalues: р-значения
- ci.low: нижняя граница доверительного интервала
- ci.up: верхняя граница доверительного интервала
- model.name: название текущей модели
Обратите внимание, что должны использоваться либо доверительные интервалы, либо стандартные ошибки и значения p, а не оба.
Манипулирование texreg объекты
Помимо передачи моделей в screenreg, texreg, или же htmlreg функции, можно сохранить извлеченную информацию в texreg Объект сначала и манипулируйте им, прежде чем таблица будет отображена или сохранена. Дополнительным преимуществом является то, что даже сложные изменения в таблице легко применять таким способом. Например, можно переименовать коэффициенты или статистику GOF, добавить новые строки, переименовать модель, изменить значения или изменить порядок коэффициентов или статистики GOF. Вот как вы можете сделать это: во-первых, вы называете extract функция для сохранения информации в texreg объект:
tr <- extract(model.output.1)
Вы можете отобразить содержимое texreg возразить, просто позвонив tr, который показывает следующий вывод:
No standard errors and p-values were defined for this texreg object.
coef.
Intercept -1.83522343
RACE -0.62522179
DCAPS 1.31442834
PSA 0.04686106
GOF dec. places
MSE 0.2029470 TRUE
R^2 0.1562137 TRUE
LogLoss 0.5920097 TRUE
Mean Per-Class Error 0.3612191 TRUE
AUC 0.7185655 TRUE
Gini 0.4371311 TRUE
Null Deviance 512.2888402 TRUE
Residual Deviance 449.9273825 TRUE
AIC 457.9273825 TRUE
В качестве альтернативы, это структура объекта, как показано str(tr):
Formal class 'texreg' [package "texreg"] with 10 slots
..@ coef.names : chr [1:4] "Intercept" "RACE" "DCAPS" "PSA"
..@ coef : num [1:4] -1.8352 -0.6252 1.3144 0.0469
..@ se : num(0)
..@ pvalues : num(0)
..@ ci.low : num(0)
..@ ci.up : num(0)
..@ gof.names : chr [1:9] "MSE" "R^2" "LogLoss" "Mean Per-Class Error" ...
..@ gof : num [1:9] 0.203 0.156 0.592 0.361 0.719 ...
..@ gof.decimal: logi [1:9] TRUE TRUE TRUE TRUE TRUE TRUE ...
..@ model.name : chr(0)
Теперь вы можете просто манипулировать этим объектом произвольным образом, например, добавить статистику GOF:
tr@gof.names <- c(tr@gof.names, "new statistic")
tr@gof <- c(tr@gof, 12)
tr@gof.decimal <- c(tr@gof.decimal, FALSE)
Или вы можете изменить порядок коэффициентов:
tr@coef.names <- tr@coef.names[c(4, 1, 2, 3)]
tr@coef <- tr@coef[c(4, 1, 2, 3)]
Когда вы закончите с манипуляциями, вы можете передать texreg объект вместо исходной модели при вызове, например, screenreg:
screenreg(list(tr, model.output.2), custom.note = "")
Новый результат будет выглядеть так:
======================================
Model 1 Model 2
--------------------------------------
PSA 0.05 0.05
Intercept -1.84 -1.11
RACE -0.63 -0.62
DCAPS 1.31 1.31
AGE -0.01
--------------------------------------
MSE 0.20 0.20
R^2 0.16 0.16
LogLoss 0.59 0.59
Mean Per-Class Error 0.36 0.38
AUC 0.72 0.72
Gini 0.44 0.44
Null Deviance 512.29 512.29
Residual Deviance 449.93 449.51
AIC 457.93 459.51
new statistic 12
======================================
TL; DR
texreg могут быть настроены пользователями. Просто напишите метод извлечения, подобный показанному выше, и зарегистрируйте его, используя setMethods вызов. Я включил H2OBinomialModel метод в последнем texreg версия 1.36.13, а также исправление ошибок при использовании моделей без стандартных ошибок (например, этой).
Вы можете использовать пакет R xtable с H2OTable h2o (или knitr, если вы преобразуете H2OTable в H2OFrame, используя as.h2o(your_H2OTable)), если вы извлекаете их из выходных данных модели.
например, чтобы создать красивую таблицу из коэффициентов модели, вам необходимо сначала извлечь таблицу коэффициентов с помощью model.output.1@model$coefficients_tableтогда вы можете использовать xtable: xtable(prostate.glm@model$coefficients_table) распечатать латексный код.
для просмотра рядом есть несколько сообщений о том, как это сделать в knitr или xtable, или в xtable и sweave
Нет, нет пакета, который делает это в настоящее время. Пакет с метлой пока не поддерживает модели H2O - это было бы круто! Может быть, это может произойти в будущем. Если есть способ "привести в порядок" вывод модели в R data.frame, используя метлу или аналогичную функциональность, тогда xtable и т. Д. Будут работать хорошо.