Как эффективно создать черно-белую маску для этого микроскопического изображения?
Итак, немного предыстории. Мне было поручено написать программу Matlab для подсчета количества дрожжевых клеток в микроскопических изображениях в видимом свете. Для этого я думаю, что первым шагом будет сегментация клеток. Прежде чем я получил реальный экспериментальный набор изображений, я разработал алгоритм, использующий тестовый набор изображений с использованием водораздела. Который выглядит так:
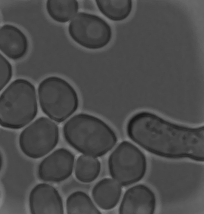
Первым шагом водораздела является создание маски BW для клеток. Затем я генерировал бы изображение bwdist с наложенными локальными минимумами, сгенерированными из маски BW. С этим я могу легко создать водораздел.
Как видите, мой алгоритм опирается на успешную генерацию маски BW. Потому что мне нужно сгенерировать изображение bwdist и маркеры из него. Первоначально я генерирую маску BW, выполнив следующие действия:
- генерировать локальное стандартное отклонение изображения sdImage = stdfilt(grayImage, ones(9))

- Используйте пороговое значение BW для создания начальной маски BW binaryImage = sdImage < 8;
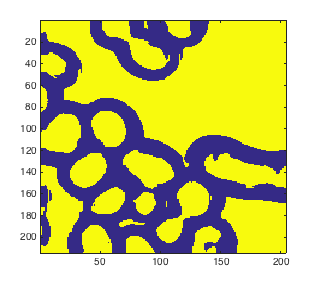
- используйте imclearborder, чтобы очистить фон. Используйте другой код, чтобы добавить ячейки на границе.
Фон закончен. Вот моя проблема
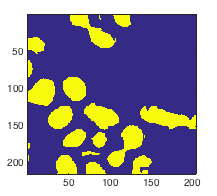
Но сегодня я получил новые реальные наборы данных. Разрешение изображения намного меньше, а условия освещения отличаются от набора тестового изображения. Глубина цвета также намного меньше. Это делает мой алгоритм бесполезным. Вот:
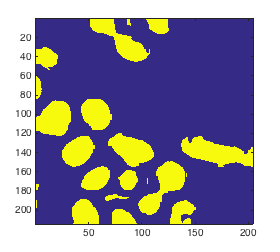
Использование stdfilt не смогло генерировать хорошие чистые изображения. Вместо этого он генерирует что-то вроде этого (Примечание: я настроил параметры для функции stdfilt и пороговое значение BW, следующее - лучший результат, который я могу получить):

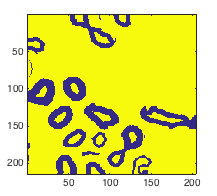
Как вы можете видеть, в центре клеток есть светлые пиксели, которые не обязательно темнее мембраны. Что приводит к порождению bw порождает такие вещи:

Новое изображение bw после bresh thresholding имеет либо неполную мембрану, либо сегментированные клеточные центры и делает их непригодными для других этапов.
Я только недавно начал обработку изображений и не знаю, как мне поступить. Если у вас есть идея, пожалуйста, помогите мне! Спасибо!
Для вашего удобства я прикрепил ссылку из выпадающего списка для подмножества изображений.
1 ответ
Я думаю, что в вашем подходе есть фундаментальная проблема. Ваш алгоритм использует stdfilt для того, чтобы оцифровывать изображение. Но это, по сути, означает, что вы предполагаете, что на заднем плане и внутри ячейки есть низкая "текстура". Это работает для вашего первого изображения. Однако на вашем втором изображении внутри ячейки есть "текстура", поэтому это предположение нарушается.
Я думаю, что более сильное предположение состоит в том, что вокруг каждой ячейки есть "кольцо" (действительно для обоих изображений, которые вы разместили). Поэтому я выбрал способ обнаружения этого кольца.
Так что мой подход по сути:
- Обнаружение этих колец (я использую фильтр 'log', а затем преобразовываю их в двоичную форму на основе положительных значений. Однако, это приводит к большому количеству "болтовни")
- Попробуйте сначала убрать часть "болтовни", отфильтровывая очень маленькие и очень большие регионы.
- Теперь заполните эти кольца. Тем не менее, между ячейками остается некоторая "болтовня" и заполненные области.
- Опять же, удалите маленькие и большие области, но, так как клетки заполнены, увеличьте границы для того, что является приемлемым.
- Есть все еще некоторые плохие области, большинство плохих областей будет областями между ячейками. Области между ячейками обнаруживаются путем наблюдения кривизны вокруг границы области. Они сильно "изгибаются внутрь", что математически выражается как большая часть границы, имеющая отрицательную кривизну. Кроме того, чтобы удалить остальную часть "болтовни", эти области будут иметь большое стандартное отклонение кривизны их границы, поэтому удаляйте также границы с большим стандартным отклонением.
В целом, самой сложной частью будет удаление областей между ячейками и "болтовня" без удаления самих ячеек.
В любом случае, вот код (обратите внимание, что существует много эвристик, а также он очень грубый и основан на коде из более старых проектов, домашних заданий и ответов stackru, так что он определенно далек от завершения):
cell = im2double(imread('cell1.png'));
if (size(cell,3) == 3)
cell = rgb2gray(cell);
end
figure(1), subplot(3,2,1)
imshow(cell,[]);
% Detect edges
hw = 5;
cell_filt = imfilter(cell, fspecial('log',2*hw+1,1));
subplot(3,2,2)
imshow(cell_filt,[]);
% First remove hw and filter out noncell hws
mask = cell_filt > 0;
hw = 5;
mask = mask(hw:end-hw-1,hw:end-hw-1);
subplot(3,2,3)
imshow(mask,[]);
rp = regionprops(mask, 'PixelIdxList', 'Area');
rp = rp(vertcat(rp.Area) > 50 & vertcat(rp.Area) < 2000);
mask(:) = false;
mask(vertcat(rp.PixelIdxList)) = true;
subplot(3,2,4)
imshow(mask,[]);
% Now fill objects
mask1 = true(size(mask) + hw);
mask1(hw+1:end, hw+1:end) = mask;
mask1 = imfill(mask1,'holes');
mask1 = mask1(hw+1:end, hw+1:end);
mask2 = true(size(mask) + hw);
mask2(hw+1:end, 1:end-hw) = mask;
mask2 = imfill(mask2,'holes');
mask2 = mask2(hw+1:end, 1:end-hw);
mask3 = true(size(mask) + hw);
mask3(1:end-hw, 1:end-hw) = mask;
mask3 = imfill(mask3,'holes');
mask3 = mask3(1:end-hw, 1:end-hw);
mask4 = true(size(mask) + hw);
mask4(1:end-hw, hw+1:end) = mask;
mask4 = imfill(mask4,'holes');
mask4 = mask4(1:end-hw, hw+1:end);
mask = mask1 | mask2 | mask3 | mask4;
% Filter out large and small regions again
rp = regionprops(mask, 'PixelIdxList', 'Area');
rp = rp(vertcat(rp.Area) > 100 & vertcat(rp.Area) < 5000);
mask(:) = false;
mask(vertcat(rp.PixelIdxList)) = true;
subplot(3,2,5)
imshow(mask);
% Filter out regions with lots of positive concavity
% Get boundaries
[B,L] = bwboundaries(mask);
% Cycle over boundarys
for i = 1:length(B)
b = B{i};
% Filter boundary - use circular convolution
b(:,1) = cconv(b(:,1),fspecial('gaussian',[1 7],1)',size(b,1));
b(:,2) = cconv(b(:,2),fspecial('gaussian',[1 7],1)',size(b,1));
% Find curvature
curv_vec = zeros(size(b,1),1);
for j = 1:size(b,1)
p_b = b(mod(j-2,size(b,1))+1,:); % p_b = point before
p_m = b(mod(j,size(b,1))+1,:); % p_m = point middle
p_a = b(mod(j+2,size(b,1))+1,:); % p_a = point after
dx_ds = p_a(1)-p_m(1); % First derivative
dy_ds = p_a(2)-p_m(2); % First derivative
ddx_ds = p_a(1)-2*p_m(1)+p_b(1); % Second derivative
ddy_ds = p_a(2)-2*p_m(2)+p_b(2); % Second derivative
curv_vec(j+1) = dx_ds*ddy_ds-dy_ds*ddx_ds;
end
if (sum(curv_vec > 0)/length(curv_vec) > 0.4 || std(curv_vec) > 2.0)
L(L == i) = 0;
end
end
mask = L ~= 0;
subplot(3,2,6)
imshow(mask,[])
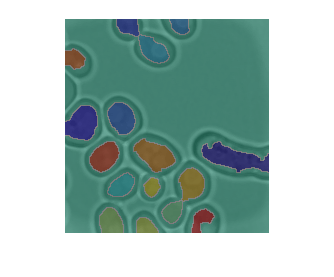
Output1:

Выход2:
