Как пометить барплот в ggplot с метками в другом результате теста?
Я хотел бы пометить свой график выводом метки теста, например, выводом теста LSD (a, b, ab и т. Д.), Используя LSD.test в библиотеке Agricolae. Вот бегущий пример.
library(ggplot2)
library(agricolae)
wt<-gl(3,4,108,labels=c("W30","W60","W90"))
pl<-gl(3,12,108,labels=c("P0","P1","P2"))
gp<-gl(3,36,108,labels=c("A","B","C"))
dat<-cbind(
A=runif(108),
B=runif(108,min=1,max=10),
C=runif(108,min=100,max=200),
D=runif(108,min=1000,max=1500)
)
dat.df<-data.frame(wt,pl,gp,dat)
dat.m<-melt(dat.df)
ggplot(dat.m,aes(x=wt,y=value,group=pl,facet=gp,fill=pl))+
stat_summary(fun.y=mean,geom="bar",size=2,position="dodge")+
stat_summary(fun.ymin=function(x)(mean(x)-sd(x)/sqrt(length(x))),geom="errorbar",
fun.ymax=function(x)(mean(x)+sd(x)/sqrt(length(x))),position="dodge")+
facet_grid(variable~facet,scale="free_y")+
opts(legend.position="top")+
scale_colour_manual(values = c("red", "blue", "green"))
Обычно в другой библиотеке я проверял данные и передавал метку текстовому графику, но возможно ли это сделать в ggplot? например, в stat_summary(), которые используют LSD.test в fun.y?
2 ответа
Решение
Чтобы достичь этого, вы должны создать еще один слой меток, используя geom_text и укажите свой собственный набор данных.
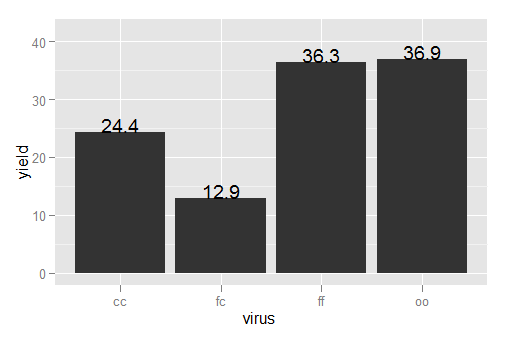
Расширение на примере в lsd.test в упаковке agricolae:
library(agricolae)
library(ggplot2)
data(sweetpotato)
model <- aov(yield~virus, data=sweetpotato)
lsd <- LSD.test(model,"virus",p.adj="bon")
ggplot() +
stat_summary(data=sweetpotato, aes(x=virus, y=yield), fun.y=mean, geom="bar") +
geom_text(data=lsd, aes(x=trt, y=means, label=round(means, 1)), vjust=0)
Ты можешь попробовать это
во-первых, определяется вектор на основе значения pvalue, например:
padj=ifelse(pval<0.001,'*','')
затем добавьте столбец padj к вашему data.fram,
head(df)
name type number padj
1 metabolic process biological_process 968 *
2 catalytic activity molecular_function 801 *
3 cellular metabolic process biological_process 617 *
4 biosynthetic process biological_process 357 *
5 cellular protein metabolic process biological_process 279
6 cytoplasm cellular_component 202 *
и, наконец, добавьте geom_text к вашему графику
p <- ggplot(data=df, aes(x=name,y=number,fill=type))+
geom_bar(position=position_dodge())+
scale_fill_brewer(palette="Set2")+
geom_text(aes(label=padj), vjust=0.25,hjust=0.25)
p