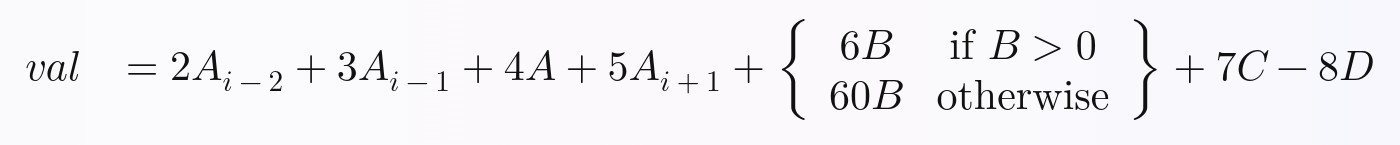Каков наиболее эффективный способ циклически перебирать кадры данных с пандами?
Я хочу последовательно выполнять свои собственные сложные операции с финансовыми данными в рамках данных.
Например, я использую следующий файл MSFT CSV, полученный от Yahoo Finance:
Date,Open,High,Low,Close,Volume,Adj Close
2011-10-19,27.37,27.47,27.01,27.13,42880000,27.13
2011-10-18,26.94,27.40,26.80,27.31,52487900,27.31
2011-10-17,27.11,27.42,26.85,26.98,39433400,26.98
2011-10-14,27.31,27.50,27.02,27.27,50947700,27.27
....
Затем я делаю следующее:
#!/usr/bin/env python
from pandas import *
df = read_csv('table.csv')
for i, row in enumerate(df.values):
date = df.index[i]
open, high, low, close, adjclose = row
#now perform analysis on open/close based on date, etc..
Это самый эффективный способ? Учитывая фокус на скорости в пандах, я бы предположил, что должна быть какая-то специальная функция для итерации значений таким образом, чтобы каждый также извлекал индекс (возможно, через генератор, чтобы эффективно использовать память)? df.iteritems к сожалению, перебирает только столбец за столбцом.
13 ответов
В новейшие версии панд теперь включена встроенная функция для перебора строк.
for index, row in df.iterrows():
# do some logic here
Или, если вы хотите быстрее использовать itertuples()
Но предложение unutbu использовать функции numpy, чтобы избежать итерации по строкам, даст самый быстрый код.
Панды основаны на массивах NumPy. Ключом к скорости работы с массивами NumPy является выполнение операций со всем массивом одновременно, а не строка за строкой или пункт за элементом.
Например, если close массив 1-й, и вы хотите, чтобы процентное изменение за день,
pct_change = close[1:]/close[:-1]
Это вычисляет весь массив процентных изменений как один оператор, а не
pct_change = []
for row in close:
pct_change.append(...)
Поэтому постарайтесь избежать цикла Python for i, row in enumerate(...) целиком и подумайте о том, как выполнять свои вычисления с операциями над всем массивом (или фреймом данных) в целом, а не построчно.
Как и ранее, объект pandas наиболее эффективен при одновременной обработке всего массива. Однако для тех, кому действительно нужно выполнить цикл DataFrame для панд, чтобы выполнить что-то, как я, я нашел по крайней мере три способа сделать это. Я сделал короткий тест, чтобы увидеть, какой из трех занимает меньше всего времени.
t = pd.DataFrame({'a': range(0, 10000), 'b': range(10000, 20000)})
B = []
C = []
A = time.time()
for i,r in t.iterrows():
C.append((r['a'], r['b']))
B.append(time.time()-A)
C = []
A = time.time()
for ir in t.itertuples():
C.append((ir[1], ir[2]))
B.append(time.time()-A)
C = []
A = time.time()
for r in zip(t['a'], t['b']):
C.append((r[0], r[1]))
B.append(time.time()-A)
print B
Результат:
[0.5639059543609619, 0.017839908599853516, 0.005645036697387695]
Это, вероятно, не лучший способ измерить потребление времени, но для меня это быстро.
Вот некоторые плюсы и минусы ИМХО:
- .iterrows (): возвращает элементы index и row в отдельных переменных, но значительно медленнее
- .itertuples(): быстрее, чем.iterrows(), но возвращает индекс вместе с элементами строки, ir[0] - индекс
- zip: самый быстрый, но нет доступа к индексу строки
Вы можете циклически проходить по строкам, транспонируя и вызывая iteritems:
for date, row in df.T.iteritems():
# do some logic here
Я не уверен в эффективности в этом случае. Чтобы получить максимально возможную производительность в итеративном алгоритме, вы, возможно, захотите изучить написание его на Cython, чтобы вы могли сделать что-то вроде:
def my_algo(ndarray[object] dates, ndarray[float64_t] open,
ndarray[float64_t] low, ndarray[float64_t] high,
ndarray[float64_t] close, ndarray[float64_t] volume):
cdef:
Py_ssize_t i, n
float64_t foo
n = len(dates)
for i from 0 <= i < n:
foo = close[i] - open[i] # will be extremely fast
Я бы порекомендовал сначала написать алгоритм на чистом Python, убедиться, что он работает, и посмотреть, насколько он быстр - если он недостаточно быстр, конвертируйте вещи в Cython, как это, с минимальными затратами, чтобы получить что-то такое же быстрое, как C-код, написанный вручную. /C++.
У вас есть три варианта:
По индексу (самое простое):
>>> for index in df.index:
... print ("df[" + str(index) + "]['B']=" + str(df['B'][index]))
С помощью iterrows (наиболее часто используемых):
>>> for index, row in df.iterrows():
... print ("df[" + str(index) + "]['B']=" + str(row['B']))
С itertuples (самый быстрый):
>>> for row in df.itertuples():
... print ("df[" + str(row.Index) + "]['B']=" + str(row.B))
Три варианта отображают что-то вроде:
df[0]['B']=125
df[1]['B']=415
df[2]['B']=23
df[3]['B']=456
df[4]['B']=189
df[5]['B']=456
df[6]['B']=12
Источник: neural-networks.io
Я проверил iterrows заметив ответ Nick Crawford, но обнаружив, что он дает (индекс, серия) кортежи. Не уверен, что будет работать лучше для вас, но в итоге я использовал itertuples метод для моей задачи, который дает (index, row_value1...) кортежи.
Есть также iterkv, который перебирает (столбец, серия) кортежи.
Как небольшое дополнение, вы также можете подать заявку, если у вас есть сложная функция, которую вы применяете к одному столбцу:
http://pandas.pydata.org/pandas-docs/dev/generated/pandas.DataFrame.apply.html
df[b] = df[a].apply(lambda col: do stuff with col here)
Как отметил joris, iterrows намного медленнее, чем itertuples а также itertuples примерно в 100 раз больше, чем iterrowsи я проверил скорость обоих методов в DataFrame с 5027505 записями, результат для iterrows, это 1200it/s, и itertuples 120000it/ с.
Если вы используете itertuplesОбратите внимание, что каждый элемент цикла for является именованным кортежем, поэтому для получения значения в каждом столбце вы можете обратиться к следующему примеру кода
>>> df = pd.DataFrame({'col1': [1, 2], 'col2': [0.1, 0.2]},
index=['a', 'b'])
>>> df
col1 col2
a 1 0.1
b 2 0.2
>>> for row in df.itertuples():
... print(row.col1, row.col2)
...
1, 0.1
2, 0.2
Конечно, самый быстрый способ перебрать данные - это получить доступ к лежащему в основе nardy ndarray либо через df.values (как вы делаете) или путем доступа к каждому столбцу отдельно df.column_name.values, Поскольку вы также хотите иметь доступ к индексу, вы можете использовать df.index.values для этого.
index = df.index.values
column_of_interest1 = df.column_name1.values
...
column_of_interestk = df.column_namek.values
for i in range(df.shape[0]):
index_value = index[i]
...
column_value_k = column_of_interest_k[i]
Не питон? Конечно. Но быстро.
Если вы хотите выжать из сока больше сока, загляните в cython. Cython позволит вам получить огромные ускорения (думаю, 10x-100x). Для максимальной производительности проверьте память представлений для Cython.
Другим предложением было бы объединить групповые и векторизованные вычисления, если подмножества строк имеют общие характеристики, что позволяет вам это делать.
Я считаю, что самый простой и эффективный способ перебора DataFrames — это использование numpy и numba. В этом случае зацикливание во многих случаях может быть примерно таким же быстрым, как векторизованные операции. Если numba не вариант, следующий лучший вариант, вероятно, будет простой numpy. Как уже неоднократно отмечалось, по умолчанию вам следует использовать векторизацию, но этот ответ просто рассматривает эффективное зацикливание, учитывая решение зацикливаться по какой-либо причине.
В качестве тестового случая давайте воспользуемся примером из ответа @DSM для расчета процентного изменения. Это очень простая ситуация, и с практической точки зрения вы не стали бы писать цикл для ее вычисления, но как таковой он обеспечивает разумную основу для определения времени векторизованных подходов по сравнению с циклами.
Давайте настроим 4 подхода с небольшим фреймом данных, а ниже мы проверим их время на большом наборе данных.
import pandas as pd
import numpy as np
import numba as nb
df = pd.DataFrame( { 'close':[100,105,95,105] } )
pandas_vectorized = df.close.pct_change()[1:]
x = df.close.to_numpy()
numpy_vectorized = ( x[1:] - x[:-1] ) / x[:-1]
def test_numpy(x):
pct_chng = np.zeros(len(x))
for i in range(1,len(x)):
pct_chng[i] = ( x[i] - x[i-1] ) / x[i-1]
return pct_chng
numpy_loop = test_numpy(df.close.to_numpy())[1:]
@nb.jit(nopython=True)
def test_numba(x):
pct_chng = np.zeros(len(x))
for i in range(1,len(x)):
pct_chng[i] = ( x[i] - x[i-1] ) / x[i-1]
return pct_chng
numba_loop = test_numba(df.close.to_numpy())[1:]
А вот тайминги для DataFrame со 100 000 строк (тайминги, выполненные с помощью Jupyter
%timeitфункция, свернутая в сводную таблицу для удобочитаемости):
pandas/vectorized 1,130 micro-seconds
numpy/vectorized 382 micro-seconds
numpy/looped 72,800 micro-seconds
numba/looped 455 micro-seconds
Резюме: для простых случаев, таких как этот, вы бы использовали (векторизованные) панды для простоты и удобочитаемости и (векторизованные) numpy для скорости. Если вам действительно нужно использовать цикл, сделайте это в numpy. Если numba доступен, объедините его с numpy для дополнительной скорости. В этом случае numpy + numba почти так же быстр, как векторизованный код numpy.
Другие детали:
- Не показаны различные параметры, такие как iterrows, itertuples и т. д., которые на несколько порядков медленнее и никогда не должны использоваться.
- Тайминги здесь довольно типичны: numpy быстрее, чем pandas, а vectorized быстрее, чем циклы, но добавление numba к numpy часто значительно ускоряет numpy.
- Все, кроме опции pandas, требует преобразования столбца DataFrame в массив numpy. Это преобразование включено в тайминги.
- Время для определения/компиляции функций numpy/numba не было включено в тайминги, но, как правило, было бы незначительным компонентом тайминга для любого большого фрейма данных.
посмотри на последний
t = pd.DataFrame({'a': range(0, 10000), 'b': range(10000, 20000)})
B = []
C = []
A = time.time()
for i,r in t.iterrows():
C.append((r['a'], r['b']))
B.append(round(time.time()-A,5))
C = []
A = time.time()
for ir in t.itertuples():
C.append((ir[1], ir[2]))
B.append(round(time.time()-A,5))
C = []
A = time.time()
for r in zip(t['a'], t['b']):
C.append((r[0], r[1]))
B.append(round(time.time()-A,5))
C = []
A = time.time()
for r in range(len(t)):
C.append((t.loc[r, 'a'], t.loc[r, 'b']))
B.append(round(time.time()-A,5))
C = []
A = time.time()
[C.append((x,y)) for x,y in zip(t['a'], t['b'])]
B.append(round(time.time()-A,5))
B
0.46424
0.00505
0.00245
0.09879
0.00209
Это самый эффективный способ? Учитывая акцент на скорости в пандах, я бы предположил, что должна быть какая-то специальная функция для перебора значений...
Безусловно, есть самый эффективный способ: векторизация . После этого следует понимание списка , а затем
itertuples(). Держись подальше от . Это довольно ужасно, поскольку выполняется намного медленнее, чем необработанный цикл for с обычнымиdf["A"][i]-типовая индексация, даже.
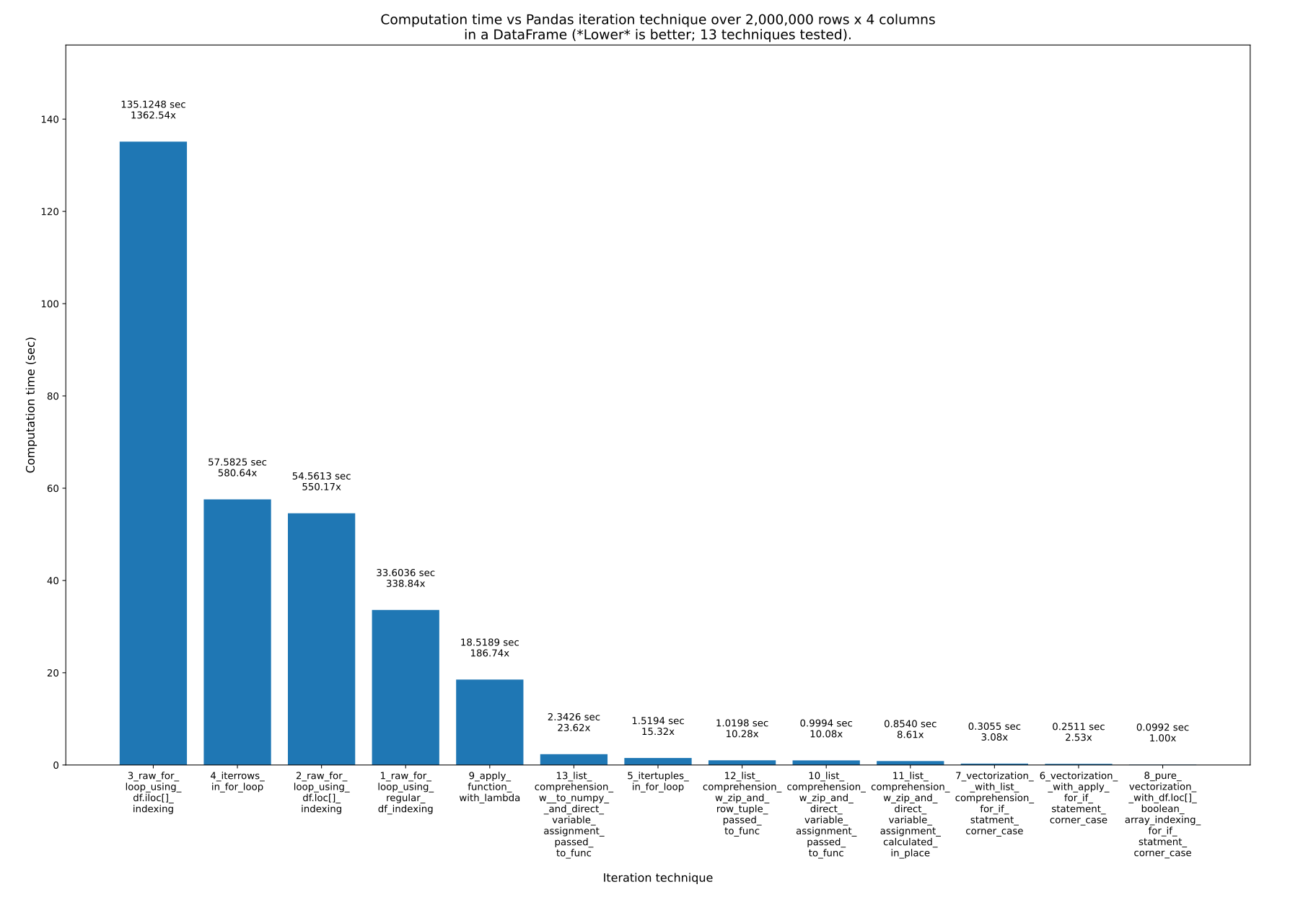
Здесь я очень подробно представляю 13 способов, проверяю их скорость и показываю весь код: .
Я трачу несколько недель на написание этого ответа. Вот результаты:
Ключевые выводы::
- Если ваша цель — код, который легко читать, писать и поддерживать, но при этом оставаться очень быстрым, используйте понимание списка . Это всего лишь в 10 раз медленнее , чем чистая векторизация.
- Если ваша цель — максимально быстрый код , используйте чистую векторизацию . Труднее читать и писать, если у вас есть сложные уравнения, такие как
ifОднако утверждения в вашей формуле вы рассчитываете для каждой строки.
Такие функции, какiterrows()работают ужасно медленно , примерно в 600 раз медленнее , чем чистая векторизация.
Чтобы доказать, что все 13 методов, которые я тестировал на скорости, возможны даже в сложных формулах, я выбрал эту нетривиальную формулу для вычислений с использованием всех методов:
Более подробную информацию и код для всех 13 методов можно найти в моем основном ответе: Как перебирать Pandas [с итерацией и без]Как перебирать Pandas.
DataFrames без итерации .