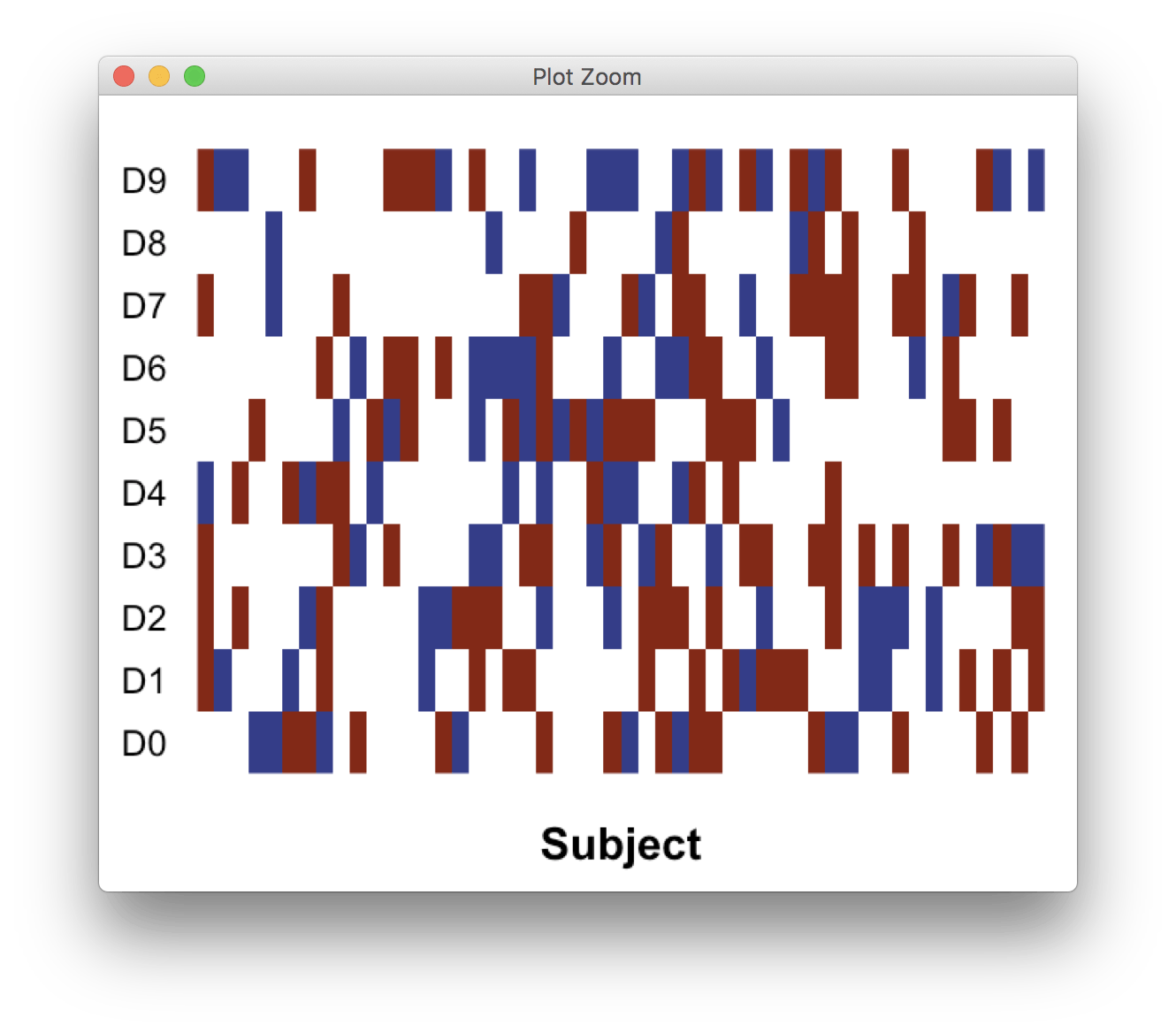
Двойные цветные прямоугольники из ggplot2 geom_raster()
Мой вопрос похож на эту ссылку. В этом примере 1 кодируется для мутации, 0 для дикого типа и NA для недоступной. Мой фрейм данных настроен идентично, однако он может содержать два или более типов мутаций на ген. Я хотел бы создать аналогичную фигуру, за исключением случаев, когда ген имеет два типа мутаций, я хотел бы, чтобы квадрат был разрезан пополам, и оба типа мутаций были закрашены, как в этом примере. В настоящее время, если ген у субъекта имеет две мутации, вторая мутация заполняет запись первой. Заранее спасибо, что нашли время помочь.
dat <- expand.grid(gene=1:10, subj=1:50)
dat$mut <- as.factor(sample(c(rep(0,300),rep(1,200)),500))
dat$mut[sample(500,300)] <- NA
dat[501,] = c(10,50,1) #included from comment below
ggplot(dat, aes(x=subj, y=gene, fill=mut)) +
geom_raster() +
scale_fill_manual(values = c("#8D1E0B","#323D8D"), na.value="#FFFFFF") +
scale_x_discrete("Subject") +
scale_y_continuous(breaks=1:10,
labels=c("D0","D1","D2","D3","D4","D5","D6","D7","D8","D9")) +
guides(fill=FALSE) +
theme(
axis.ticks.x=element_blank(), axis.ticks.y=element_blank(),
axis.text.x=element_blank(), axis.text.y=element_text(colour="#000000"),
axis.title.x=element_text(face="bold"), axis.title.y=element_blank(),
panel.grid.major.x=element_blank(), panel.grid.major.y=element_blank(),
panel.grid.minor.x=element_blank(), panel.grid.minor.y=element_blank(),
panel.background=element_rect(fill="#ffffff")
)
1 ответ
Я не вижу, что ваши данные когда-либо имеют несколько записей для субъекта и гена? Ничего не перезаписывается, потому что нечего перезаписывать.
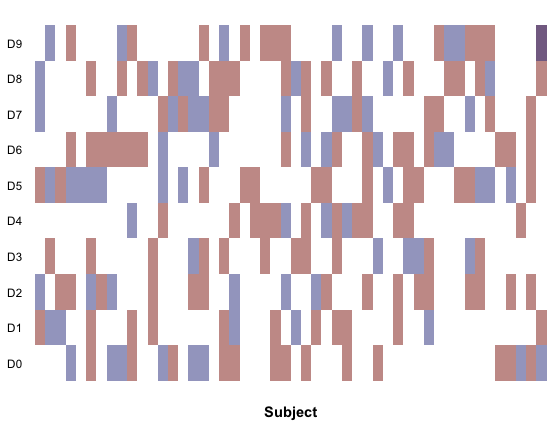
Я добавил повтор последней строки, но изменил mut на 1, чтобы показать. Я также изменил растр на плитку и изменил непрозрачность, чтобы плитки с несколькими значениями имели другой цвет.
Если вы хотите что-то вроде графика, на который вы ссылаетесь, вам нужно также создать вектор смещения и высоты, как показано в этом сообщении, чтобы каждая плитка снова была подразделена.
dat[501,] = c(10,50,1)
ggplot(dat, aes(x=subj, y=gene)) +
geom_tile(alpha=.5,aes(fill=mut), show.legend = F) +
scale_fill_manual(values = c("#8D1E0B","#323D8D"), na.value="transparent") +
scale_x_discrete("Subject") +
scale_y_continuous(breaks=1:10,
labels=c("D0","D1","D2","D3","D4","D5","D6","D7","D8","D9")) +
theme(
axis.ticks.x=element_blank(), axis.ticks.y=element_blank(),
axis.text.x=element_blank(), axis.text.y=element_text(colour="#000000"),
axis.title.x=element_text(face="bold"), axis.title.y=element_blank(),
panel.grid.major.x=element_blank(), panel.grid.major.y=element_blank(),
panel.grid.minor.x=element_blank(), panel.grid.minor.y=element_blank(),
panel.background=element_rect(fill="#ffffff")
)