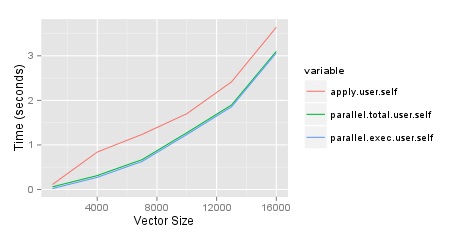
Почему параллельный пакет медленнее, чем просто применение apply?
Я пытаюсь определить, когда использовать parallel Пакет для ускорения времени, необходимого для запуска некоторого анализа. Одна из вещей, которые мне нужно сделать, это создать матрицы, сравнивающие переменные в двух фреймах данных с разным количеством строк. Я задал вопрос об эффективном способе работы в Stackru и написал о тестах в своем блоге. Поскольку я доволен лучшим подходом, я хотел ускорить процесс, запустив его параллельно. Приведенные ниже результаты основаны на 2 ГГц i7 Mac с 8 ГБ оперативной памяти. Я удивлен, что parallel пакет, parSapply в частности, функция хуже, чем просто использование apply функция. Код для воспроизведения этого ниже. Обратите внимание, что в настоящее время я использую только один из двух созданных мной столбцов, но в конечном итоге хочу использовать оба.
http://jason.bryer.org/images/ParalleVsApplyTiming.png
require(parallel)
require(ggplot2)
require(reshape2)
set.seed(2112)
results <- list()
sizes <- seq(1000, 30000, by=5000)
pb <- txtProgressBar(min=0, max=length(sizes), style=3)
for(cnt in 1:length(sizes)) {
i <- sizes[cnt]
df1 <- data.frame(row.names=1:i,
var1=sample(c(TRUE,FALSE), i, replace=TRUE),
var2=sample(1:10, i, replace=TRUE) )
df2 <- data.frame(row.names=(i + 1):(i + i),
var1=sample(c(TRUE,FALSE), i, replace=TRUE),
var2=sample(1:10, i, replace=TRUE))
tm1 <- system.time({
df6 <- sapply(df2$var1, FUN=function(x) { x == df1$var1 })
dimnames(df6) <- list(row.names(df1), row.names(df2))
})
rm(df6)
tm2 <- system.time({
cl <- makeCluster(getOption('cl.cores', detectCores()))
tm3 <- system.time({
df7 <- parSapply(cl, df1$var1, FUN=function(x, df2) { x == df2$var1 }, df2=df2)
dimnames(df7) <- list(row.names(df1), row.names(df2))
})
stopCluster(cl)
})
rm(df7)
results[[cnt]] <- c(apply=tm1, parallel.total=tm2, parallel.exec=tm3)
setTxtProgressBar(pb, cnt)
}
toplot <- as.data.frame(results)[,c('apply.user.self','parallel.total.user.self',
'parallel.exec.user.self')]
toplot$size <- sizes
toplot <- melt(toplot, id='size')
ggplot(toplot, aes(x=size, y=value, colour=variable)) + geom_line() +
xlab('Vector Size') + ylab('Time (seconds)')
3 ответа
Параллельное выполнение заданий связано с накладными расходами. Только если задания, выполняемые на рабочих узлах, занимают значительное время, параллелизация повышает общую производительность. Когда отдельные задания занимают только миллисекунды, накладные расходы на постоянное увольнение ухудшают общую производительность. Хитрость заключается в том, чтобы разделить работу по узлам таким образом, чтобы задания были достаточно длинными, скажем, по крайней мере, несколько секунд. Я использовал это с большим эффектом, запустив шесть моделей Фортрана одновременно, но эти отдельные прогоны модели заняли часы, почти сводя на нет эффект накладных расходов.
Обратите внимание, что я не запускал ваш пример, но ситуация, которую я описал выше, часто является проблемой, когда парализация занимает больше времени, чем последовательная работа.
Эти различия могут быть отнесены к 1) накладным расходам связи (особенно если вы работаете на узлах) и 2) накладным расходам производительности (если ваша работа не такая интенсивная по сравнению с инициацией распараллеливания, например). Обычно, если задача, которую вы распараллеливаете, не так трудоемка, вы обнаружите, что распараллеливание НЕ дает большого эффекта (что очень заметно на огромных наборах данных.
Даже если это не может напрямую повлиять на ваш сравнительный анализ, я надеюсь, что это будет довольно просто и может быть связано с. В качестве примера здесь я строю data.frame с 1e6 строки с 1e4 уникальный столбец group записи и некоторые значения в столбце val, И тогда я бегу, используя plyr в parallel с помощью doMC и без распараллеливания.
df <- data.frame(group = as.factor(sample(1:1e4, 1e6, replace = T)),
val = sample(1:10, 1e6, replace = T))
> head(df)
group val
# 1 8498 8
# 2 5253 6
# 3 1495 1
# 4 7362 9
# 5 2344 6
# 6 5602 9
> dim(df)
# [1] 1000000 2
require(plyr)
require(doMC)
registerDoMC(20) # 20 processors
# parallelisation using doMC + plyr
P.PLYR <- function() {
o1 <- ddply(df, .(group), function(x) sum(x$val), .parallel = TRUE)
}
# no parallelisation
PLYR <- function() {
o2 <- ddply(df, .(group), function(x) sum(x$val), .parallel = FALSE)
}
require(rbenchmark)
benchmark(P.PLYR(), PLYR(), replications = 2, order = "elapsed")
test replications elapsed relative user.self sys.self user.child sys.child
2 PLYR() 2 8.925 1.000 8.865 0.068 0.000 0.000
1 P.PLYR() 2 30.637 3.433 15.841 13.945 8.944 38.858
Как видите, параллельная версия plyr работает в 3,5 раза медленнее
Теперь позвольте мне использовать то же самое data.frame, но вместо вычислений sumПозвольте мне построить немного более сложную функцию, скажем, median(.) * median(rnorm(1e4) ((бессмысленно, да):
Вы увидите, что приливы начинают меняться:
# parallelisation using doMC + plyr
P.PLYR <- function() {
o1 <- ddply(df, .(group), function(x)
median(x$val) * median(rnorm(1e4)), .parallel = TRUE)
}
# no parallelisation
PLYR <- function() {
o2 <- ddply(df, .(group), function(x)
median(x$val) * median(rnorm(1e4)), .parallel = FALSE)
}
> benchmark(P.PLYR(), PLYR(), replications = 2, order = "elapsed")
test replications elapsed relative user.self sys.self user.child sys.child
1 P.PLYR() 2 41.911 1.000 15.265 15.369 141.585 34.254
2 PLYR() 2 73.417 1.752 73.372 0.052 0.000 0.000
Здесь параллельная версия 1.752 times быстрее, чем непараллельная версия.
Изменить: После комментария @ Пола, я только что реализовал небольшую задержку, используя Sys.sleep(), Конечно результаты очевидны. Но только для полноты приведем результат для 20*2 data.frame:
df <- data.frame(group=sample(letters[1:5], 20, replace=T), val=sample(20))
# parallelisation using doMC + plyr
P.PLYR <- function() {
o1 <- ddply(df, .(group), function(x) {
Sys.sleep(2)
median(x$val)
}, .parallel = TRUE)
}
# no parallelisation
PLYR <- function() {
o2 <- ddply(df, .(group), function(x) {
Sys.sleep(2)
median(x$val)
}, .parallel = FALSE)
}
> benchmark(P.PLYR(), PLYR(), replications = 2, order = "elapsed")
# test replications elapsed relative user.self sys.self user.child sys.child
# 1 P.PLYR() 2 4.116 1.000 0.056 0.056 0.024 0.04
# 2 PLYR() 2 20.050 4.871 0.028 0.000 0.000 0.00
Разница здесь не удивительна.
Полностью согласен с аргументами @Arun и @PaulHiemestra относительно почему...? часть вашего вопроса.
Тем не менее, кажется, что вы можете воспользоваться parallel пакет в вашей ситуации (по крайней мере, если вы не застряли с Windows). Возможное решение использует mclapply вместо parSapply, который опирается на быструю разветвленность и разделяемую память.
tm2 <- system.time({
tm3 <- system.time({
df7 <- matrix(unlist(mclapply(df2$var1, FUN=function(x) {x==df1$var1}, mc.cores=8)), nrow=i)
dimnames(df7) <- list(row.names(df1), row.names(df2))
})
})
Конечно, вложенные system.time здесь не нужно С моими 2 ядрами я получил: