ggplot2 горизонтальная полоса с градиентной заливкой
Я пытаюсь создать горизонтальную линейчатую диаграмму и хотел бы заполнить отдельную полосу в соответствии с их значениями логарифмического сгиба: белый для самого низкого значения и самый темный для самого высокого значения. Однако я не в состоянии это сделать. Вот моя работа, может кто-нибудь помочь мне это исправить?
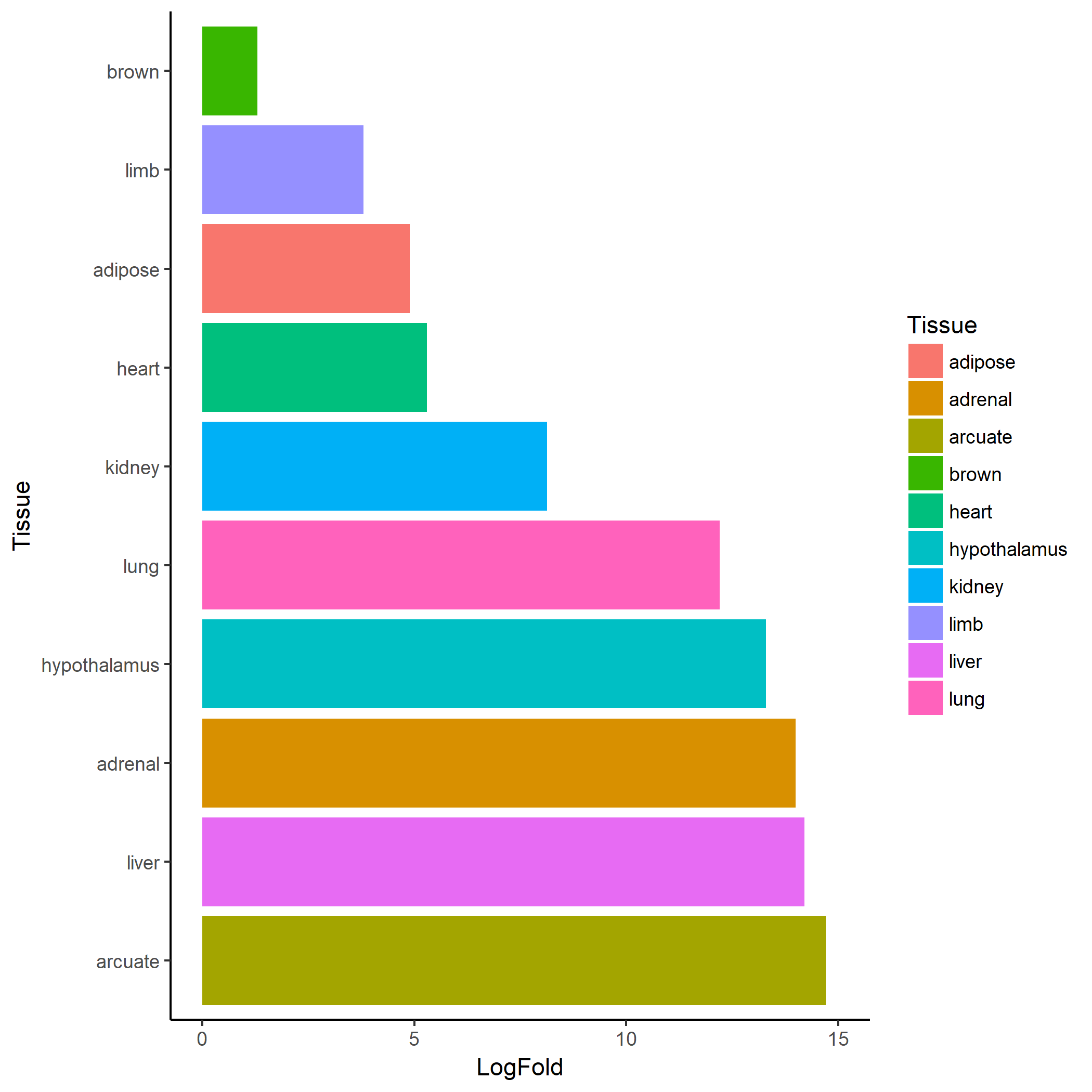
df <- data.frame(LogFold = c(14.20, 14.00, 8.13, 5.3, 3.8, 4.9, 1.3, 13.3, 14.7, 12.2),
Tissue = c("liver", "adrenal", "kidney", "heart", "limb", "adipose", "brown", "hypothalamus", "arcuate", "lung"))
df1<-df%>%
arrange(desc(LogFold))
ggplot(data=df1, aes(x=Tissue, y=LogFold, fill = Tissue)) +
geom_bar(stat="identity")+
scale_colour_gradient2()+
coord_flip()+
ylim(0, 15)+
scale_x_discrete(limits = df1$Tissue)+
theme_classic()
Заранее спасибо!
2 ответа
Вот что вам нужно подумать:
- сортировка значений фрейма данных не имеет значения для ggplot
- в вашем коде вы отображаете цвет заливки в Tissue, а не в LogFold
- новее
geom_colменьше печатать, чемgeom_bar(stat = ...) - пределы вашей шкалы не нужны, так как вы указываете все значения для ткани
- если вы хотите заполнить с использованием градиента
scale_fill_gradient2() - заливка = белый не будет виден на белом фоне
theme_classic
Таким образом, вы можете попробовать что-то вроде этого:
library(tidyverse)
df1 %>%
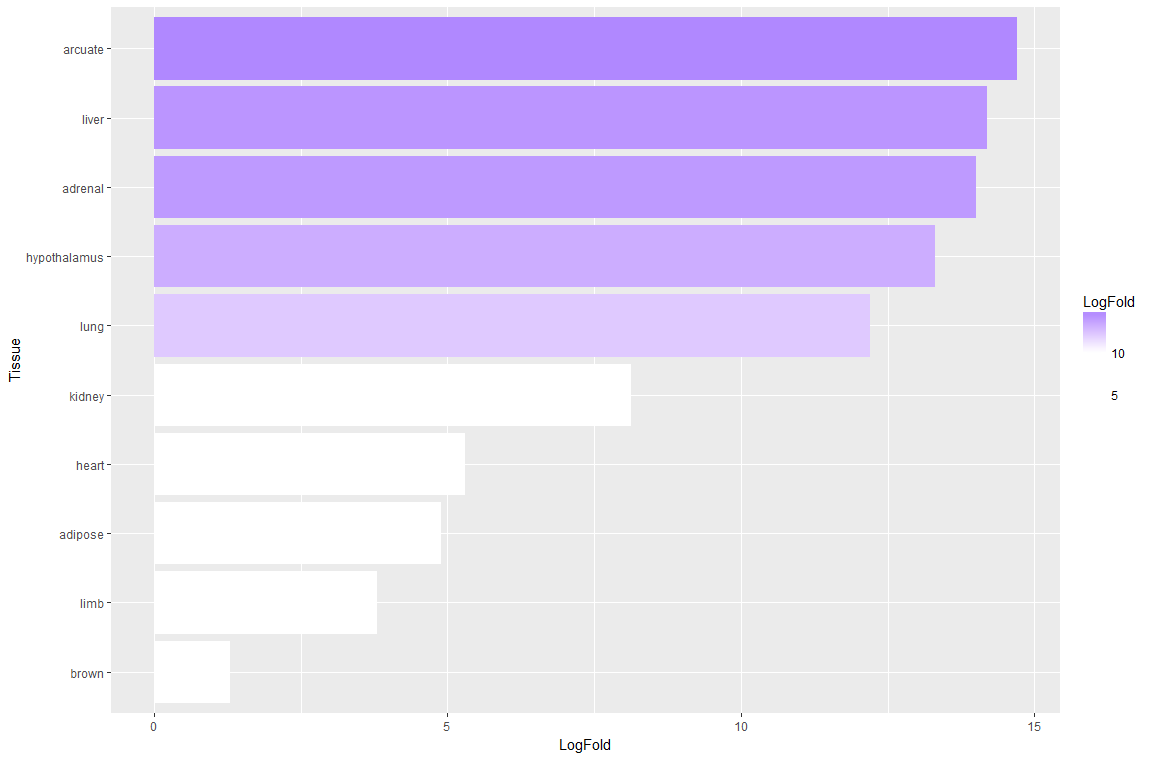
ggplot(aes(reorder(Tissue, LogFold), LogFold)) +
geom_col(aes(fill = LogFold)) +
scale_fill_gradient2(low = "white",
high = "blue",
midpoint = median(df1$LogFold)) +
coord_flip() +
labs(x = "Tissue")
Но я не знаю, что цветовой градиент действительно добавляет что-то в плане интерпретации информации. Итак, вот результат без него, вы будете судьей:
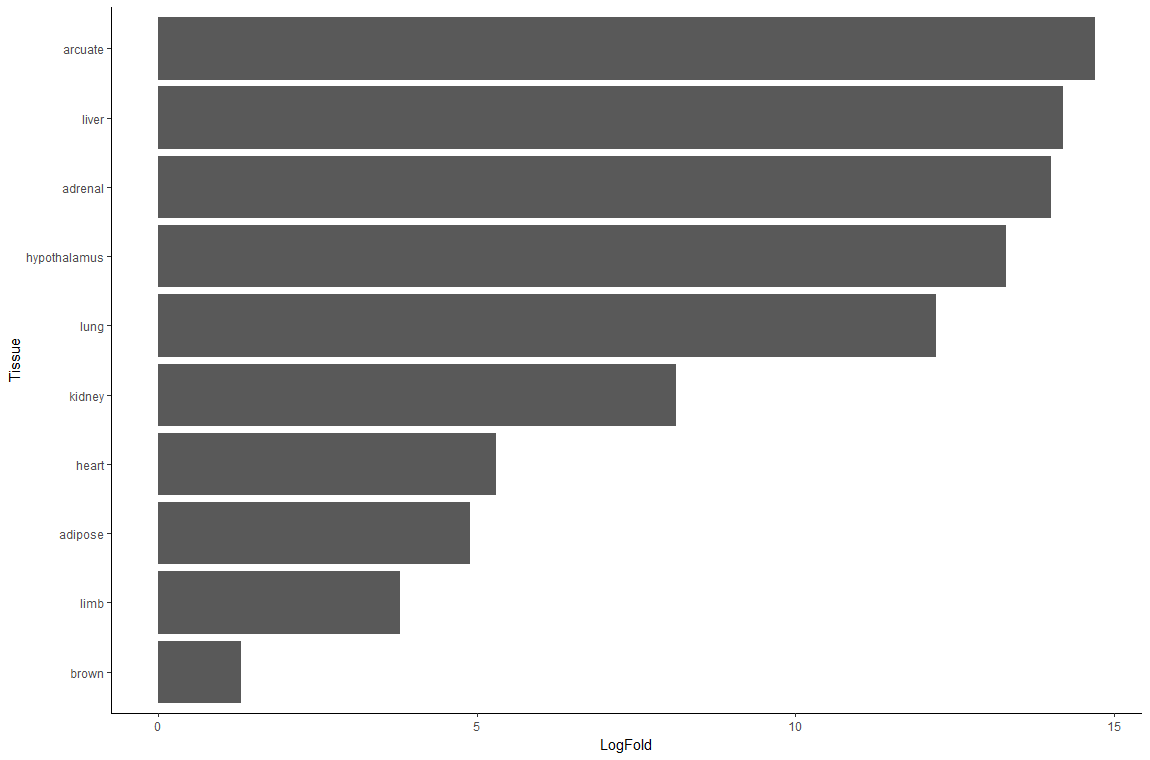
df1 %>%
ggplot(aes(reorder(Tissue, LogFold), LogFold)) +
geom_col() +
coord_flip() +
labs(x = "Tissue") +
theme_classic()
Надеюсь, что это работает.
ggplot(data=df1, aes(x=Tissue, y=LogFold, fill = LogFold)) +
geom_bar(stat="identity",color="black")+
scale_fill_gradient(low="white",high="darkred")+
coord_flip()+
ylim(0, 15)+
scale_x_discrete(limits = df1$Tissue)+
theme_classic()