Выбираете eps и minpts для DBSCAN (R)?
Я долго искал ответ на этот вопрос, поэтому надеюсь, что кто-нибудь сможет мне помочь. Я использую dbscan из библиотеки fpc в R. Например, я смотрю на набор данных USArrests и использую для него dbscan следующим образом:
library(fpc)
ds <- dbscan(USArrests,eps=20)
Выбор eps был просто методом проб и ошибок в этом случае. Однако мне интересно, есть ли доступная функция или код для автоматизации выбора лучших eps / minpts. Я знаю, что в некоторых книгах рекомендовано составить график сортировки k-го расстояния до ближайшего соседа. То есть ось x представляет "точки, отсортированные по расстоянию до k-го ближайшего соседа", а ось y представляет "k-е расстояние ближайшего соседа".
Этот тип графика полезен для выбора подходящего значения для eps и minpts. Я надеюсь, что предоставил достаточно информации, чтобы кто-нибудь помог мне. Я хотел опубликовать фотографию того, что имел в виду, однако я все еще новичок, поэтому пока не могу опубликовать изображение.
4 ответа
Нет общего способа выбора minPts. Это зависит от того, что вы хотите найти. Низкое значение minPts означает, что оно будет создавать больше кластеров из шума, поэтому не выбирайте его слишком маленьким.
Для эпсилона существуют различные аспекты. Это снова сводится к выбору того, что работает с этим набором данных и этими minPts и этой функцией расстояния и этой нормализацией. Вы можете попытаться сделать гистограмму с дистанционным управлением и выбрать там "колено", но там может не быть ни видимого, ни множественного числа.
OPTICS является преемником DBSCAN, которому не нужен параметр epsilon (за исключением соображений производительности с поддержкой индексов, см. Википедию). Это гораздо приятнее, но я считаю, что это сложно реализовать в R, потому что для этого нужны усовершенствованные структуры данных (в идеале, дерево индексов данных для ускорения и обновляемая куча для очереди приоритетов), а R - это все о матричных операциях.
Наивно можно представить, что OPTICS выполняет все значения Epsilon одновременно и помещает результаты в кластерную иерархию.
Однако первое, что вам нужно проверить - в значительной степени независимо от того, какой алгоритм кластеризации вы собираетесь использовать - это убедиться, что у вас есть полезная функция расстояния и соответствующая нормализация данных. Если ваше расстояние вырождается, алгоритм кластеризации работать не будет.
MinPts
Как объяснил Anony-Mousse: "Низкие значения minPts означают, что он будет создавать больше кластеров из шума, поэтому не выбирайте его слишком маленьким".,
minPts лучше всего устанавливать экспертом в области, который хорошо понимает данные. К сожалению, во многих случаях мы не знаем предметной области, особенно после нормализации данных. Одним из эвристических подходов является использование ln (n), где n - общее количество точек, которые будут кластеризованы.
эпсилон
Есть несколько способов определить это:
1) k-расстояние участка
В кластеризации с minPts = k мы ожидаем, что точки ядра и k-границы пограничных точек находятся в определенном диапазоне, в то время как точки шума могут иметь гораздо большее k-расстояние, поэтому мы можем наблюдать точку перегиба на графике k-distance, Однако иногда может не быть очевидного колена или может быть несколько колен, что затрудняет решение 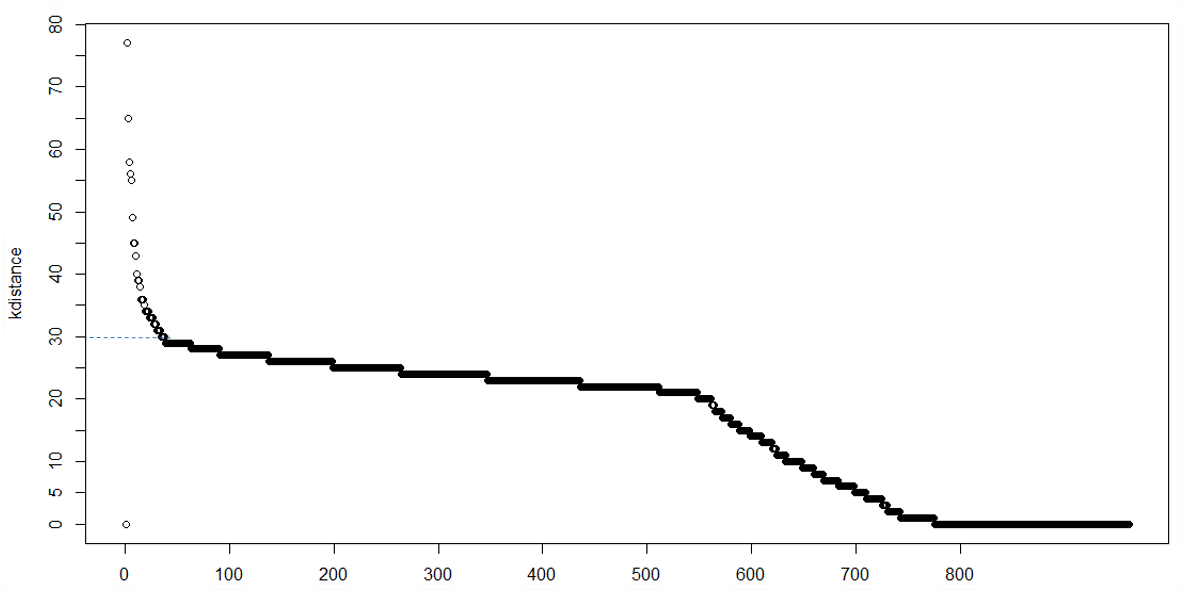
2) Расширения DBSCAN, такие как OPTICS
OPTICS создает иерархические кластеры, мы можем извлечь значимые плоские кластеры из иерархических кластеров визуальным осмотром, реализация OPTICS доступна в Pyclustering модуля Python. Один из первоначальных авторов DBSCAN и OPTICS также предложил автоматический способ извлечения плоских кластеров, где не требуется вмешательство человека, для получения дополнительной информации вы можете прочитать эту статью.
3) анализ чувствительности
По сути, мы хотим выбрать радиус, способный кластеризовать более истинно правильные точки (точки, похожие на другие точки), в то же время обнаруживая больше шума (точки выброса). Мы можем нарисовать процент регулярных точек (точки принадлежат кластеру) VS. Анализ эпсилон, где мы устанавливаем различные значения эпсилон в качестве оси X, и их соответствующий процент регулярных точек в качестве оси Y, и, мы надеемся, мы можем определить сегмент, где процентное значение регулярных точек является более чувствительным к значению эпсилон, и мы выбираем значение верхней границы эпсилона в качестве нашего оптимального параметра.
Один из распространенных и популярных способов управления параметром epsilon в DBSCAN - это вычисление графика k-расстояния вашего набора данных. По сути, вы вычисляете k-ближайших соседей (k-NN) для каждой точки данных, чтобы понять, каково распределение плотности ваших данных для разных k. KNN удобен, потому что это непараметрический метод. Выбрав minPTS (который сильно зависит от ваших данных), вы фиксируете k на это значение. Затем вы используете в качестве эпсилона k-расстояние, соответствующее области графика k-расстояния (для вашего фиксированного k) с низким наклоном.
Подробнее о выборе параметров см. Статью ниже на с. 11:
Schubert, E., Sander, J., Ester, M., Kriegel, HP, & Xu, X. (2017). DBSCAN вновь, снова и снова: почему и как вы должны (все еще) использовать DBSCAN. Транзакции ACM в системах баз данных (TODS), 42(3), 19.
- Для двумерных данных: используйте значение по умолчанию minPts=4 (Ester et al., 1996)
- Для более чем двух измерений: minPts=2*dim (Sander et al., 1998)
Как только вы знаете, какой MinPts выбрать, вы можете определить Epsilon:
- Постройте k-расстояния с k=minPts (Ester et al., 1996)
- Найдите "колено" на графике -> значение k-расстояния - это ваше значение Epsilon.
Если у вас есть ресурсы, вы также можете протестировать кучу epsilon а также minPtsценности и посмотрите, что работает. Я делаю это используяexpand.grid а также mapply.
# Establish search parameters.
k <- c(25, 50, 100, 200, 500, 1000)
eps <- c(0.001, 0.01, 0.02, 0.05, 0.1, 0.2)
# Perform grid search.
grid <- expand.grid(k = k, eps = eps)
results <- mapply(grid$k, grid$eps, FUN = function(k, eps) {
cluster <- dbscan(data, minPts = k, eps = eps)$cluster
sum <- table(cluster)
cat(c("k =", k, "; eps =", eps, ";", sum, "\n"))
})
См. Эту веб-страницу, раздел 5: http://www.sthda.com/english/wiki/dbscan-density-based-clustering-for-discovering-clusters-in-large-datasets-with-noise-unsupervised-machine-learning
Он дает подробные инструкции о том, как найти эпсилон. MinPts ... не так много.