Как сделать сгруппированный макет в igraph?
В igraphПосле применения алгоритма модульности для нахождения сообществ графов я хотел бы нарисовать схему сети, которая четко делает видимыми отдельные сообщества и их связи. Что-то вроде "макета атрибутов группы" в Cytoscape: я хочу показать членов каждой группы / сообщества близко друг к другу, и держать некоторое расстояние между группами / сообществами. Я не мог найти какую-либо функцию в igraph предоставляя эту функцию из коробки. Размещая этот вопрос, я уже нашел простое решение для поделки, я собираюсь опубликовать его в качестве ответа. Но мне интересно, есть ли лучшая возможность или более продуманное решение?
4 ответа
Чтобы расширить предложение Габора, я создал эту функцию:
weight.community=function(row,membership,weigth.within,weight.between){
if(as.numeric(membership[which(names(membership)==row[1])])==as.numeric(membership[which(names(membership)==row[2])])){
weight=weigth.within
}else{
weight=weight.between
}
return(weight)
}
Просто примените его к строкам матрицы ребер вашего графа (get.edgelist(your_graph)) чтобы установить новые веса ребер (членство является вектором членства из результата любого алгоритма обнаружения сообщества):
E(g)$weight=apply(get.edgelist(g),1,weight.community,membership,10,1)
Затем просто используйте алгоритм компоновки, который принимает веса ребер, такие как fruchterman.reingold, как это было предложено Габором. Вы можете настроить весовые аргументы, чтобы получить график, который вы хотите. Например:
E(g)$weight=apply(get.edgelist(g),1,weight.community,membership,10,1)
g$layout=layout.fruchterman.reingold(g,weights=E(g)$weight)
plot(g)

E(g)$weight=apply(get.edgelist(g),1,weight.community,membership,1000,1)
g$layout=layout.fruchterman.reingold(g,weights=E(g)$weight)
plot(g)

Примечание 1: прозрачность / цвета краев - это другие параметры моих графиков. Я покрасил узлы сообществом, чтобы показать, что это действительно работает.
Примечание 2: обязательно используйте membership(comm) и не comm$membership, где comm является результатом алгоритма обнаружения сообщества (например, comm=leading.eigenvector.community(g)). Причина в том, что в первом случае вы получаете числовой вектор с именами (что мы хотим), а во втором - тот же вектор без имен.
Чтобы получить консенсус в отношении нескольких алгоритмов обнаружения сообщества, см. Эту функцию.
Вдохновленный предложением Антуана, я создал эту функцию:
edge.weights <- function(community, network, weight.within = 100, weight.between = 1) {
bridges <- crossing(communities = community, graph = network)
weights <- ifelse(test = bridges, yes = weight.between, no = weight.within)
return(weights)
}
Функция делает то же самое; просто поместите свой объект сообщества в слот сообщества, а свой график - в сетевой. Я бы оставил weight.between = 1 и настроить weight.within значение.
Затем перенесите вес в weight слот в узлах:
E(graph)$weight <- edge.weights(community, graph)
Наконец, используйте алгоритм макета, который использует веса, такие как layout_with_fr (новое имя fruchterman.reingold в igraph 1.0.1).
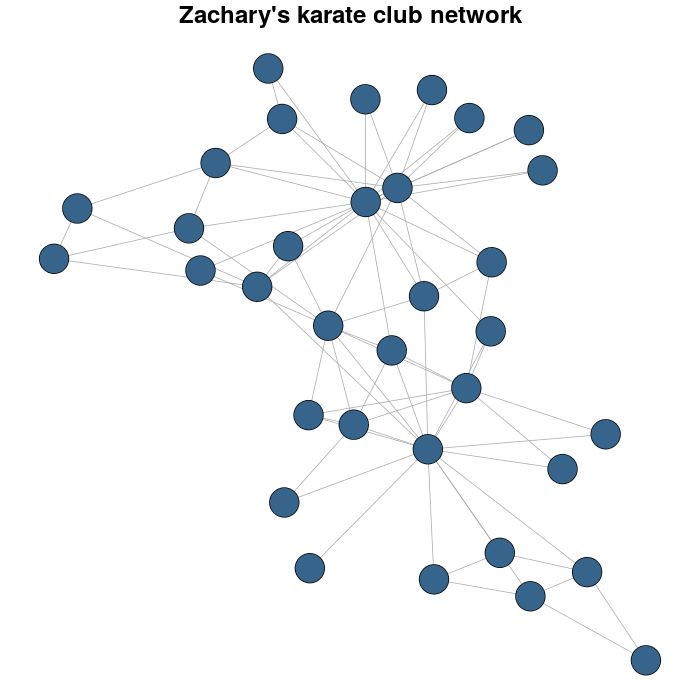
В качестве примера я использую сеть каратэ-клубов Захари.
library(igraph)
library(igraphdata)
#I load the network
data(karate)
#for reproducible purposes
set.seed(23548723)
karateLayout <- layout_with_fr(karate)
par(mar = c(0,0,2,0))
plot(karate, vertex.size = 10, vertex.color = "steelblue4", edge.width = 1,
vertex.label = NA, edge.color = "darkgrey", layout = karateLayout,
main = "Zachary's karate club network" )
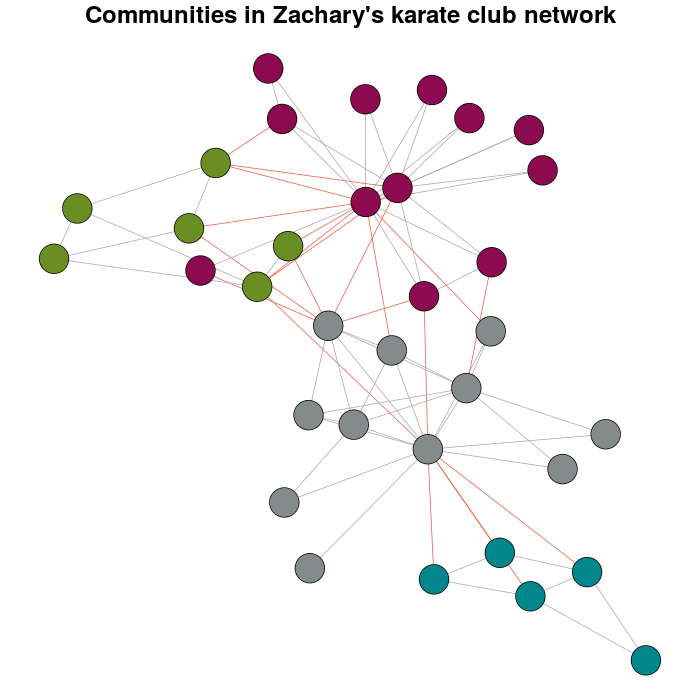
Я обнаруживаю сообщества путем многоуровневой оптимизации модульности с cluster_louvain функция:
Communitykarate <- cluster_louvain(karate)
Следующее это личное предпочтение по умолчанию:
prettyColors <- c("turquoise4", "azure4", "olivedrab","deeppink4")
communityColors <- prettyColors[membership(Communitykarate)]
График с выделенными сообществами с использованием цветов:
plot(x = Communitykarate, y = karate, edge.width = 1, vertex.size = 10,
vertex.label = NA, mark.groups = NULL, layout = karateLayout, col = communityColors,
main = "Communities in Zachary's karate club network",
edge.color = c("darkgrey","tomato2")crossing(Communitykarate, karate) + 1])
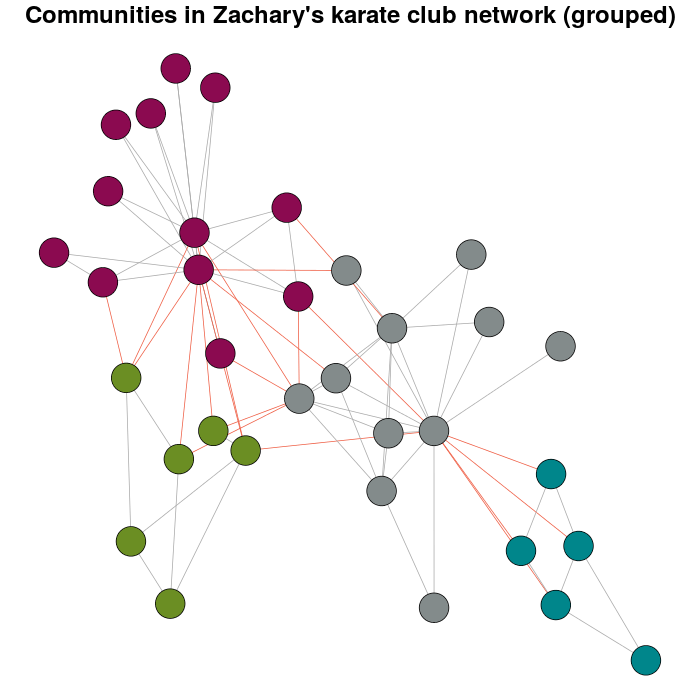
Теперь смысл того, почему этот вопрос существует.
E(karate)$weight <- edge.weights(Communitykarate, karate)
# I use the original layout as a base for the new one
karateLayoutA <- layout_with_fr(karate, karateLayout)
# the graph with the nodes grouped
plot(x = Communitykarate, y = karate, edge.width = 1, vertex.size = 10,
mark.groups = NULL, layout = karateLayoutA, vertex.label = NA, col = communityColors,
c("darkgrey","tomato2")[crossing(Communitykarate, karate) + 1],
main = "Communities in Zachary's karate club network (grouped)")
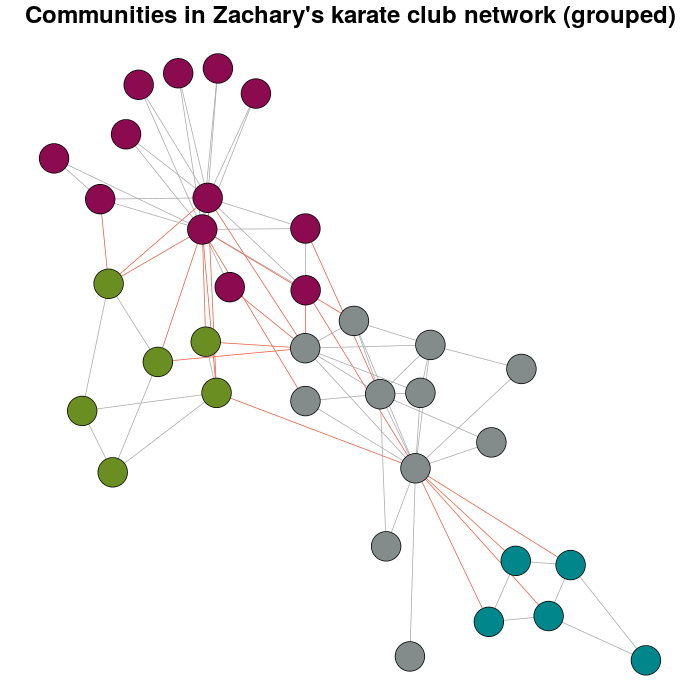
Если вы попробуете с большим весом, у вас будет:
E(karate)$weight <- edge.weights(Communitykarate, karate, weight.within = 1000)
karateLayoutB <- layout_with_fr(karate, karateLayout)
plot(x = Communitykarate, y = karate, edge.width = 1, vertex.size = 10,
mark.groups = NULL, layout = karateLayoutB, vertex.label = NA, col = communityColors,
c("darkgrey","tomato2")[crossing(Communitykarate, karate) + 1],
main = "Communities in Zachary's karate club network (grouped)")
Одним из решений было бы установить вес ребер графа на основе модульности. Установите для ребер внутри модуля некоторый большой вес, а для ребер модуля - небольшой вес. Тогда позвони layout.fruchterman.reingold()или любой алгоритм, который поддерживает веса ребер.
Возможно, вам придется немного поиграть с фактическими значениями веса, потому что это зависит от вашего графика.
Функция layout.modular предоставляет сгруппированный макет для графика, полученный в результате любого метода обнаружения сообщества igraph:
c <- fastgreedy.community(G)
layout.modular <- function(G,c){
nm <- length(levels(as.factor(c$membership)))
gr <- 2
while(gr^2<nm){
gr <- gr+1
}
i <- j <- 0
for(cc in levels(as.factor(c$membership))){
F <- delete.vertices(G,c$membership!=cc)
F$layout <- layout.kamada.kawai(F)
F$layout <- layout.norm(F$layout, i,i+0.5,j,j+0.5)
G$layout[c$membership==cc,] <- F$layout
if(i==gr){
i <- 0
if(j==gr){
j <- 0
}else{
j <- j+1
}
}else{
i <- i+1
}
}
return(G$layout)
}
G$layout <- layout.modular(G,c)
V(G)$color <- rainbow(length(levels(as.factor(c$membership))))[c$membership]
plot(G)