Обработка изображений (сегментация) в Matlab
Как я могу определить оптическую чашку и диск по сетчатке с помощью Matlab? Я хочу узнать размеры оптического обода (расстояние между оптической чашкой и оптическим диском)
Я пробовал следующий код
RGB = imread('img/A(4).jpg');
G = DialateBloodVessel(RGB);
[BW,H] = RGBThresh(G,220,60);
H = H(:,:,3);
I = edge(H,'Roberts',0.1);
imshowpair(I,G);
%%%%%%%%%% DialateBloodVessel( RGB ) %%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%
function [ RemovedBV ] = DialateBloodVessel( RGB )
%UNTITLED3 Summary of this function goes here
% Detailed explanation goes here
IM = RGB;
SE = strel('disk',10);
IM2 = imdilate(IM,SE);
%SE2 = strel('disk',10);
RemovedBV = imerode(IM2,SE);
end
%%%%%%%%%% RGBThresh(RGB,Ch1,Ch3) %%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%
function [BW,maskedRGBImage] = RGBThresh(RGB,Ch1,Ch3)
I = RGB;
% Define thresholds for channel 1 based on histogram settings
channel1Min = Ch1;
channel1Max = 255.000;
% Define thresholds for channel 2 based on histogram settings
channel2Min = 0.000;
channel2Max = 185.000;
% Define thresholds for channel 3 based on histogram settings
channel3Min = Ch3;
channel3Max = 255.000;
% Create mask based on chosen histogram thresholds
sliderBW = (I(:,:,1) >= channel1Min ) & (I(:,:,1) <= channel1Max) & ...
(I(:,:,2) >= channel2Min ) & (I(:,:,2) <= channel2Max) & ...
(I(:,:,3) >= channel3Min ) & (I(:,:,3) <= channel3Max);
BW = sliderBW;
% Initialize output masked image based on input image.
maskedRGBImage = RGB;
% Set background pixels where BW is false to zero.
maskedRGBImage(repmat(~BW,[1 1 3])) = 0;
end
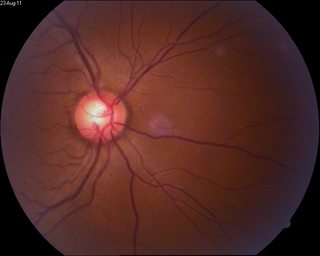
Я получаю следующий вывод, но мне нужны идеальные круги на любом изображении:
1 ответ
Когда я смотрю на ваше изображение, я замечаю две важные вещи:
Цвет не так полезен (что часто верно), потому что все довольно красное. Итак, преобразование в оттенки серого - хорошая идея.
Круг, который вы хотите выбрать, характеризуется большим изменением интенсивности, а не высокой интенсивностью. Поэтому расчет градиентов может быть полезным.
Маленькие кровеносные сосуды также имеют высокие градиенты. Так что ваши
DialateBloodVesselможет быть полезным
RGB = imread('0PBEL.jpg'); % load the image
% I crop the image to remove the black background (which gives high gradients too)
RGB = imcrop(RGB, floor([.2*size(RGB, 2) .2*size(RGB, 1) .6*size(RGB, 2) .6*size(RGB, 1)]));
G = rgb2gray(RGB); % convert to grayscale
G = DialateBloodVessel(G); % remove blood vessels
grad = imgradient(G); % calculate the gradient magnitude (direction is not important)
%display the (transformed) images: useful to validate method and tune parameters
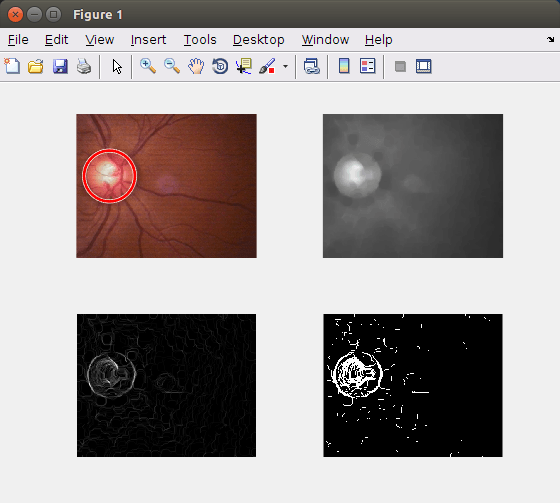
figure
subplot(2, 2, 1);
imshow(RGB)
subplot(2, 2, 2);
imshow(G)
subplot(2, 2, 3);
imshow(grad, [])
subplot(2, 2, 4);
imshow(grad >= 20, [])
% calculate the centroid and radius of all the regions
stats = regionprops('table',grad >= 20,'Centroid', 'MajorAxisLength','MinorAxisLength');
centers = stats.Centroid;
diameters = mean([stats.MajorAxisLength stats.MinorAxisLength],2);
radii = diameters/2;
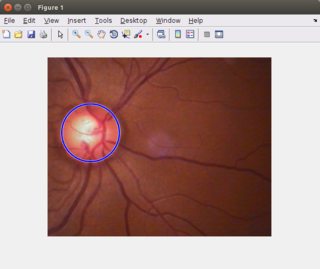
[maxRadii, iMax] = max(radii); % select the largest circle
subplot(2, 2, 1);
viscircles(centers(iMax, :),maxRadii); % visualise the selected circle
В качестве альтернативы вы можете использовать встроенный imfindcircles функционирует следующим образом:
[centers, radii, metric] = imfindcircles(G,[50 100]);
figure
imshow(RGB)
hold on
viscircles(centers, radii,'EdgeColor','b');
Обратите внимание, что этот метод может работать, но его недостатком является черный ящик.