Учебник для scipy.cluster.hierarchy
Я пытаюсь понять, как управлять иерархическим кластером, но документация слишком... техническая?... и я не могу понять, как это работает.
Есть ли какой-нибудь учебник, с которого можно начать, объясняя шаг за шагом некоторые простые задачи?
Допустим, у меня есть следующий набор данных:
a = np.array([[0, 0 ],
[1, 0 ],
[0, 1 ],
[1, 1 ],
[0.5, 0 ],
[0, 0.5],
[0.5, 0.5],
[2, 2 ],
[2, 3 ],
[3, 2 ],
[3, 3 ]])
Я легко могу сделать иерархию кластеров и построить дендрограмму:
z = linkage(a)
d = dendrogram(z)
- Теперь, как я могу восстановить конкретный кластер? Допустим, тот, с элементами
[0,1,2,4,5,6]в дендрограмме? - Как я могу вернуть значения этих элементов?
1 ответ
Существует три этапа в иерархической агломерационной кластеризации (HAC):
- Количественные данные (
metricаргумент) - Данные кластера (
methodаргумент) - Выберите количество кластеров
дела
z = linkage(a)
выполнит первые два шага. Поскольку вы не указали никаких параметров, он использует стандартные значения
metric = 'euclidean'method = 'single'
Так z = linkage(a) даст вам единую связанную иерархическую агломерацию a, Эта кластеризация является своего рода иерархией решений. Из этой иерархии вы получаете некоторую информацию о структуре ваших данных. Что вы можете сделать сейчас:
- Проверьте, какие
metricуместно, напримерcityblockили жеchebychevбудет количественно определять ваши данные (cityblock,euclideanа такжеchebychevсоответствоватьL1,L2, а такжеL_infнорма) - Проверьте различные свойства / поведение
methdos(напримерsingle,completeа такжеaverage) - Проверьте, как определить количество кластеров, например, читая вики об этом
- Вычислить индексы для найденных решений (кластеров), таких как коэффициент силуэта (с этим коэффициентом вы получаете обратную связь о качестве того, насколько хорошо точка / наблюдение соответствует кластеру, которому она назначена кластеризацией). Различные индексы используют разные критерии для определения кластеризации.
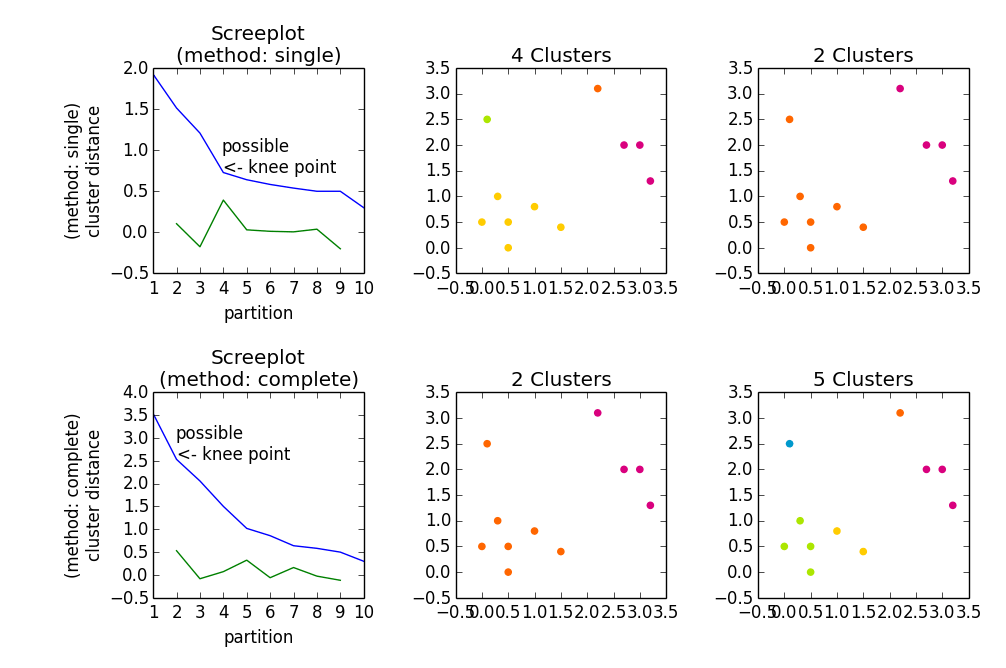
Вот с чего начать
import numpy as np
import scipy.cluster.hierarchy as hac
import matplotlib.pyplot as plt
a = np.array([[0.1, 2.5],
[1.5, .4 ],
[0.3, 1 ],
[1 , .8 ],
[0.5, 0 ],
[0 , 0.5],
[0.5, 0.5],
[2.7, 2 ],
[2.2, 3.1],
[3 , 2 ],
[3.2, 1.3]])
fig, axes23 = plt.subplots(2, 3)
for method, axes in zip(['single', 'complete'], axes23):
z = hac.linkage(a, method=method)
# Plotting
axes[0].plot(range(1, len(z)+1), z[::-1, 2])
knee = np.diff(z[::-1, 2], 2)
axes[0].plot(range(2, len(z)), knee)
num_clust1 = knee.argmax() + 2
knee[knee.argmax()] = 0
num_clust2 = knee.argmax() + 2
axes[0].text(num_clust1, z[::-1, 2][num_clust1-1], 'possible\n<- knee point')
part1 = hac.fcluster(z, num_clust1, 'maxclust')
part2 = hac.fcluster(z, num_clust2, 'maxclust')
clr = ['#2200CC' ,'#D9007E' ,'#FF6600' ,'#FFCC00' ,'#ACE600' ,'#0099CC' ,
'#8900CC' ,'#FF0000' ,'#FF9900' ,'#FFFF00' ,'#00CC01' ,'#0055CC']
for part, ax in zip([part1, part2], axes[1:]):
for cluster in set(part):
ax.scatter(a[part == cluster, 0], a[part == cluster, 1],
color=clr[cluster])
m = '\n(method: {})'.format(method)
plt.setp(axes[0], title='Screeplot{}'.format(m), xlabel='partition',
ylabel='{}\ncluster distance'.format(m))
plt.setp(axes[1], title='{} Clusters'.format(num_clust1))
plt.setp(axes[2], title='{} Clusters'.format(num_clust2))
plt.tight_layout()
plt.show()
дает