Отличаются ли значения, возвращаемые rgeos::gCentroid() и sf::st_centroid()?
Вопрос
Есть ли значения, возвращаемые rgeos::gCentroid() а также sf::st_centroid() отличаются? Если так, то как?
контекст
После прочтения соответствующих команд, экспортированных rgeos раздел в пределах r-spatial/sf Вики, я был рад видеть, что мне нужно было только sf пакет - и больше не нужно импортировать rgeos пакет - для расчета центроида заданной геометрии.
Тем не менее, использование sf::st_centroid() дал мне это предупреждение, которое адресовано здесь:
Warning message:
In st_centroid.sfc(x = comarea606$geometry) :
st_centroid does not give correct centroids for longitude/latitude data
Это предупреждение заставило меня проверить на равенство между двумя методами извлечения центроида, просто чтобы убедиться, что координаты одинаковы независимо от метода.
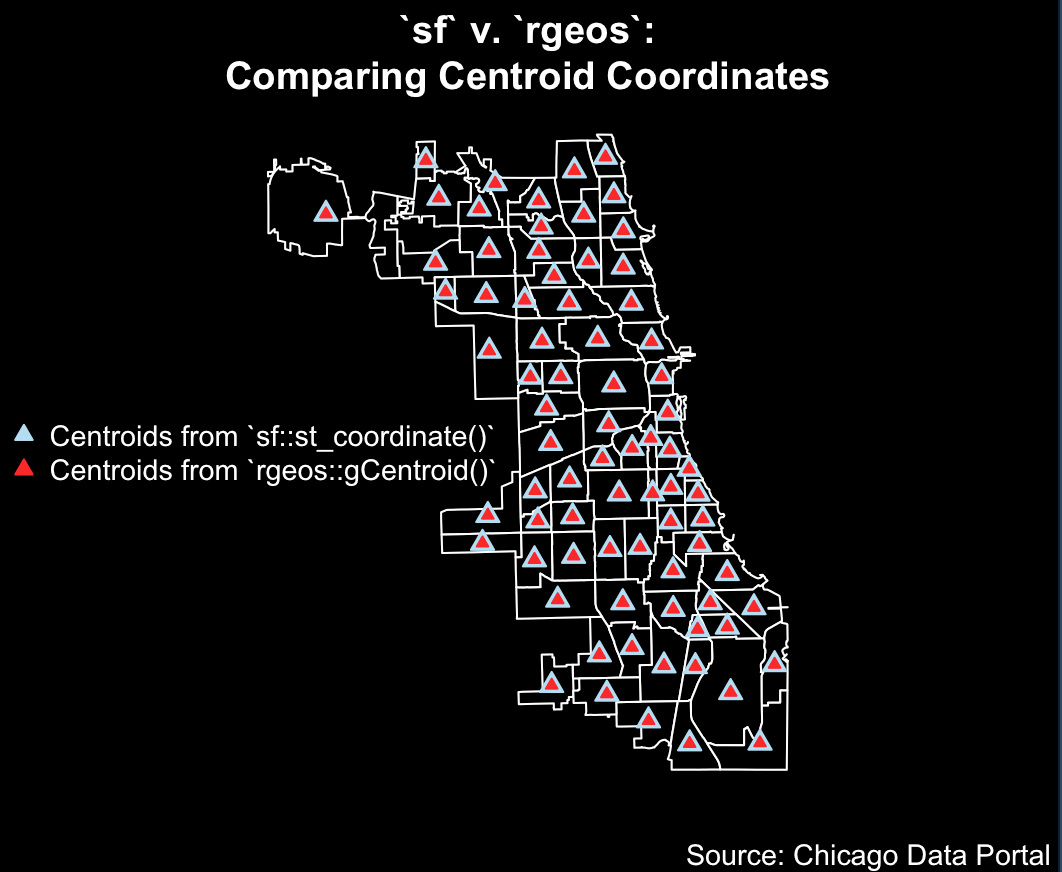
Хотя мое использование identical() а также %in% привело к неидентичным совпадениям, all.equal() и карта, на которой изображены центроиды каждого метода, говорит, что эти два метода почти идентичны.
Есть ли какая-либо причина, по которой один метод будет возвращать другой набор значений, чем другой?
Воспроизводимый пример
# load neccessary packages
library( sf )
library( rgeos )
# load data
comarea606 <-
read_sf(
dsn = "https://data.cityofchicago.org/api/geospatial/cauq-8yn6?method=export&format=GeoJSON"
, layer = "OGRGeoJSON"
)
# find the centroid of each polygon
comarea606$centroids <-
st_centroid( x = comarea606$geometry ) %>%
st_geometry()
# Warning message:
# In st_centroid.sfc(x = comarea606$geometry) :
# st_centroid does not give correct centroids for longitude/latitude data
# Ensure the st_centroid() method
# contains identical values to gCentroid()
sf.centroids <-
st_coordinates( x = comarea606$centroids )
rgeos.centroids <-
gCentroid(
spgeom = methods::as(
object = comarea606
, Class = "Spatial"
)
, byid = TRUE
)@coords
# ensure the colnames are the same
colnames( rgeos.centroids ) <-
colnames( sf.centroids )
# Test for equality
identical(
x = sf.centroids
, y = rgeos.centroids
) # [1] FALSE
all.equal(
target = sf.centroids
, current = rgeos.centroids
) # [1] TRUE
# View the first six results
head( sf.centroids )
# X Y
# 1 -87.61868 41.83512
# 2 -87.60322 41.82375
# 3 -87.63242 41.80909
# 4 -87.61786 41.81295
# 5 -87.59618 41.80892
# 6 -87.68752 41.97517
head( rgeos.centroids )
# X Y
# 1 -87.61868 41.83512
# 2 -87.60322 41.82375
# 3 -87.63242 41.80909
# 4 -87.61786 41.81295
# 5 -87.59618 41.80892
# 6 -87.68752 41.97517
# Identify the numbers which aren't identical
sf.centroids[ , "X" ] %in% rgeos.centroids[ , "X" ]
# [1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [11] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [21] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [31] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [41] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [51] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [71] FALSE FALSE FALSE FALSE FALSE FALSE FALSE
sf.centroids[ , "Y" ] %in% rgeos.centroids[ , "Y" ]
# [1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [11] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [21] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [31] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [41] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [51] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# [71] FALSE FALSE FALSE FALSE FALSE FALSE FALSE
# view results
par(
mar = c( 2, 0, 3, 0 )
, bg = "black"
)
plot(
x = comarea606$geometry
, main = "`sf` v. `rgeos`:\nComparing Centroid Coordinates"
, col.main = "white"
, col = "black"
, border = "white"
)
plot(
x = comarea606$centroids
, add = TRUE
, pch = 24
, col = "#B4DDF2"
, bg = "#B4DDF2"
, cex = 1.2
)
points(
x = rgeos.centroids[ , "X" ]
, y = rgeos.centroids[ , "Y" ]
, pch = 24
, col = "#FB0D1B"
, bg = "#FB0D1B"
, cex = 0.6
)
legend(
x = "left"
, legend = c(
"Centroids from `sf::st_coordinate()`"
, "Centroids from `rgeos::gCentroid()`"
)
, col = c( "#B4DDF2", "#FB0D1B" )
, pt.bg = c( "#B4DDF2", "#FB0D1B" )
, pch = 24
, bty = "n"
, cex = 0.9
, text.col = "white"
)
mtext(
adj = 0.99
, line = 1
, side = 1
, cex = 0.9
, text = "Source: Chicago Data Portal"
, col = "white"
)
# end of script #
Информация о сессии
R version 3.4.4 (2018-03-15)
Platform: x86_64-apple-darwin15.6.0 (64-bit)
Running under: macOS High Sierra 10.13.2
Matrix products: default
BLAS: /System/Library/Frameworks/Accelerate.framework/Versions/A/Frameworks/vecLib.framework/Versions/A/libBLAS.dylib
LAPACK: /Library/Frameworks/R.framework/Versions/3.4/Resources/lib/libRlapack.dylib
locale:
[1] en_US.UTF-8/en_US.UTF-8/en_US.UTF-8/C/en_US.UTF-8/en_US.UTF-8
attached base packages:
[1] stats graphics grDevices utils datasets methods
[7] base
other attached packages:
[1] rgeos_0.3-26 sf_0.6-0
loaded via a namespace (and not attached):
[1] modeltools_0.2-21 kernlab_0.9-25 reshape2_1.4.3
[4] lattice_0.20-35 colorspace_1.3-2 htmltools_0.3.6
[7] stats4_3.4.4 viridisLite_0.3.0 yaml_2.1.18
[10] utf8_1.1.3 rlang_0.2.0 R.oo_1.21.0
[13] e1071_1.6-8 pillar_1.2.1 withr_2.1.2
[16] DBI_0.8 prabclus_2.2-6 R.utils_2.6.0
[19] sp_1.2-7 fpc_2.1-11 plyr_1.8.4
[22] robustbase_0.92-8 stringr_1.3.0 munsell_0.4.3
[25] gtable_0.2.0 raster_2.6-7 R.methodsS3_1.7.1
[28] devtools_1.13.5 mvtnorm_1.0-7 memoise_1.1.0
[31] evaluate_0.10.1 knitr_1.20 flexmix_2.3-14
[34] class_7.3-14 DEoptimR_1.0-8 trimcluster_0.1-2
[37] Rcpp_0.12.16 udunits2_0.13 scales_0.5.0
[40] backports_1.1.2 diptest_0.75-7 classInt_0.1-24
[43] squash_1.0.8 gridExtra_2.3 ggplot2_2.2.1
[46] digest_0.6.15 stringi_1.1.6 grid_3.4.4
[49] rprojroot_1.3-2 rgdal_1.2-18 cli_1.0.0
[52] tools_3.4.4 magrittr_1.5 lazyeval_0.2.1
[55] tibble_1.4.2 cluster_2.0.6 crayon_1.3.4
[58] whisker_0.3-2 dendextend_1.7.0 MASS_7.3-49
[61] assertthat_0.2.0 rmarkdown_1.9 rstudioapi_0.7
[64] viridis_0.5.0 mclust_5.4 units_0.5-1
[67] nnet_7.3-12 compiler_3.4.4
1 ответ
Этот вопрос упрощается до: как это all.equal() отличный от identical()Переходим к документации для этих функций:
all.equal (x, y) - это утилита для сравнения R объектов x и y, проверяющих "почти равенство".
и для identical()
Безопасный и надежный способ проверить два объекта на совпадение. В этом случае он возвращает TRUE, FALSE в любом другом случае.
Давайте посмотрим поближе all.equal.numeric(), который вызывается на этих двух объектах, так как оба возвращают "double" с typeof(), Мы видим, что есть tolerance аргумент в all.equal.numeric(), который установлен в sqrt(.Machine$double.eps), по умолчанию. .Machine$double.eps это наименьшее число, которое ваша машина может добавить к 1 и быть в состоянии отличить его от 1, Это не точно, но это на порядок. all.equal.numeric() по сути, проверяет, все ли значения в векторе near() все значения в другом векторе. Вы можете посмотреть на исходный код (который в основном проверяется на наличие ошибок), чтобы точно узнать, как он это делает.
Убедить себя, что они на самом деле не identical()попробуйте посмотреть на вывод sf.centroids - rgeos.centroids,
head(sf.centroids - rgeos.centroids)
# X Y
# 1 -5.056506e-10 2.623175e-09
# 2 -2.961613e-09 -4.269602e-09
# 3 4.235119e-10 4.358100e-09
# 4 -7.688925e-10 -1.051717e-09
# 5 1.226582e-09 1.668568e-10
# 6 -2.957009e-09 4.875247e-10
Эти две матрицы, наиболее определенно, почти одинаковы (но ни одно из значений не является абсолютно одинаковым).