Спектральная кластеризация графа в питоне
Я хотел бы кластеризовать граф в Python, используя спектральную кластеризацию.
Спектральная кластеризация - это более общий метод, который можно применять не только к графам, но также к изображениям или любым другим данным, однако он считается исключительным методом кластеризации графов. К сожалению, я не могу найти примеры спектральных графов кластеризации в Python онлайн.
В Scikit Learn задокументированы два метода спектральной кластеризации: SpectralClustering и spectral_clustering, которые кажутся не псевдонимами.
Оба этих метода упоминают, что они могут использоваться на графиках, но не предлагают конкретных инструкций. Ни один не делает руководство пользователя. Я просил такой пример у разработчиков, но они перегружены работой и не дошли до этого.
Хорошая сеть, чтобы документировать это против сети Каратэ Клуба. Это включено как метод в networkx.
Я хотел бы получить какое-то направление, как это сделать. Если кто-то может помочь мне разобраться, я могу добавить документацию в scikit learn.
Заметки:
3 ответа
Без большого опыта в области спектральной кластеризации и просто прохождения документации (пропустите до конца для результатов!):
Код:
import numpy as np
import networkx as nx
from sklearn.cluster import SpectralClustering
from sklearn import metrics
np.random.seed(1)
# Get your mentioned graph
G = nx.karate_club_graph()
# Get ground-truth: club-labels -> transform to 0/1 np-array
# (possible overcomplicated networkx usage here)
gt_dict = nx.get_node_attributes(G, 'club')
gt = [gt_dict[i] for i in G.nodes()]
gt = np.array([0 if i == 'Mr. Hi' else 1 for i in gt])
# Get adjacency-matrix as numpy-array
adj_mat = nx.to_numpy_matrix(G)
print('ground truth')
print(gt)
# Cluster
sc = SpectralClustering(2, affinity='precomputed', n_init=100)
sc.fit(adj_mat)
# Compare ground-truth and clustering-results
print('spectral clustering')
print(sc.labels_)
print('just for better-visualization: invert clusters (permutation)')
print(np.abs(sc.labels_ - 1))
# Calculate some clustering metrics
print(metrics.adjusted_rand_score(gt, sc.labels_))
print(metrics.adjusted_mutual_info_score(gt, sc.labels_))
Выход:
ground truth
[0 0 0 0 0 0 0 0 0 1 0 0 0 0 1 1 0 0 1 0 1 0 1 1 1 1 1 1 1 1 1 1 1 1]
spectral clustering
[1 1 0 1 1 1 1 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]
just for better-visualization: invert clusters (permutation)
[0 0 1 0 0 0 0 1 1 1 0 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1]
0.204094758281
0.271689477828
Общая идея:
Введение в данные и задачи отсюда:
Узлы на графике представляют 34 члена клуба каратэ колледжа. (Захари - социолог, и он был одним из членов.) Разница между двумя узлами указывает на то, что два члена провели значительное время вместе вне обычных клубных встреч. Набор данных интересен тем, что, пока Захари собирал свои данные, в клубе каратэ возник спор, и он разделился на две фракции: одна во главе с "Мистером Мистером". Привет ", и один во главе с" Джоном А ". Оказывается, используя только информацию о связности (ребра), можно восстановить две фракции.
Использование sklearn и спектральной кластеризации для решения этой проблемы:
Если сродство является матрицей смежности графа, этот метод можно использовать для поиска нормализованных разрезов графа.
Это описывает нормализованные разрезы графа как:
Найдите два непересекающихся разбиения A и B вершин V графа так, чтобы A ∪ B = V и A ∩ B = ∅
Учитывая меру сходства w(i,j) между двумя вершинами (например, тождество, когда они связаны), значение среза (и его нормализованная версия) определяется как: cut(A, B) = SUM u в A, v в B: w (u, v)
...
мы стремимся минимизировать диссоциацию между группами A и B и максимизировать ассоциацию в каждой группе
Звучит хорошо. Итак, мы создаем матрицу смежности (nx.to_numpy_matrix(G)) и установить параметр affinity к предварительно вычисленному (поскольку наша матрица смежности является нашей предварительно вычисленной мерой подобия).
В качестве альтернативы, с использованием предварительно вычисленного, может использоваться предоставленная пользователем матрица сродства.
Редактировать: пока незнакомый с этим, я искал параметры для настройки и нашел assign_labels:
Стратегия, используемая для назначения меток в области встраивания. Есть два способа назначить метки после вложения Лапласа. K-средства могут быть применены и является популярным выбором. Но он также может быть чувствительным к инициализации. Дискретизация - это другой подход, который менее чувствителен к случайной инициализации.
Итак, попробуем менее чувствительный подход:
sc = SpectralClustering(2, affinity='precomputed', n_init=100, assign_labels='discretize')
Выход:
ground truth
[0 0 0 0 0 0 0 0 0 1 0 0 0 0 1 1 0 0 1 0 1 0 1 1 1 1 1 1 1 1 1 1 1 1]
spectral clustering
[0 0 1 0 0 0 0 0 1 1 0 0 0 0 1 1 0 0 1 0 1 0 1 1 1 1 1 1 1 1 1 1 1 1]
just for better-visualization: invert clusters (permutation)
[1 1 0 1 1 1 1 1 0 0 1 1 1 1 0 0 1 1 0 1 0 1 0 0 0 0 0 0 0 0 0 0 0 0]
0.771725032425
0.722546051351
Это в значительной степени идеально подходит для истины!
Вот фиктивный пример, чтобы увидеть, что он делает с простой матрицей подобия - вдохновленный ответом sascha.
Код
import numpy as np
from sklearn.cluster import SpectralClustering
from sklearn import metrics
np.random.seed(0)
adj_mat = [[3,2,2,0,0,0,0,0,0],
[2,3,2,0,0,0,0,0,0],
[2,2,3,1,0,0,0,0,0],
[0,0,1,3,3,3,0,0,0],
[0,0,0,3,3,3,0,0,0],
[0,0,0,3,3,3,1,0,0],
[0,0,0,0,0,1,3,1,1],
[0,0,0,0,0,0,1,3,1],
[0,0,0,0,0,0,1,1,3]]
adj_mat = np.array(adj_mat)
sc = SpectralClustering(3, affinity='precomputed', n_init=100)
sc.fit(adj_mat)
print('spectral clustering')
print(sc.labels_)
Выход
spectral clustering
[0 0 0 1 1 1 2 2 2]
Если мы хотим разделить график на 2 кластера, мы могли бы использовать следующую функцию
linalg.algebraicconnectivity.fiedler_vector()изnetworkxтакже, чтобы вычислить вектор Фидлера (собственный вектор, соответствующий второму наименьшему собственному значению матрицы Лапласа графа) графа, с предположением, что граф является связным неориентированным графом.Мы можем использовать указанную выше функцию, чтобы получить вектор Фидлера, а затем установить пороговые значения вектора для вычисления индексов кластера, которым соответствует каждый узел.
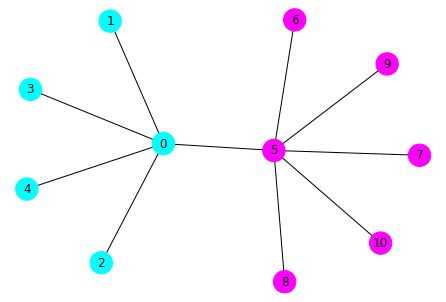
import networkx as nx import numpy as np A = np.zeros((11,11)) A[0,1] = A[0,2] = A[0,3] = A[0,4] = 1 A[5,6] = A[5,7] = A[5,8] = A[5,9] = A[5,10] = 1 A[0,5] = 5 G = nx.from_numpy_matrix(A) ev = nx.linalg.algebraicconnectivity.fiedler_vector(G) labels = [0 if v < 0 else 1 for v in ev] # using threshold 0 labels # [0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1] nx.draw(G, pos=nx.drawing.layout.spring_layout(G), with_labels=True, node_color=labels)
Мы можем итеративно / рекурсивно использовать функцию для дальнейшего разбиения каждого из кластеров.
С участием
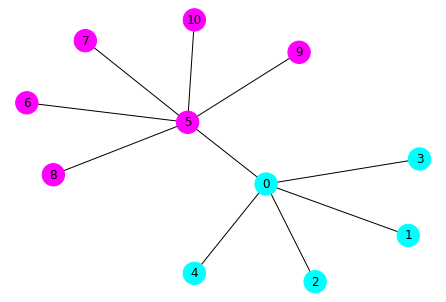
SpectralClusteringизsklearn.clusterмы можем получить точно такой же результат:sc = SpectralClustering(2, affinity='precomputed', n_init=100) sc.fit(A) sc.labels_ # [0 0 0 0 0 1 1 1 1 1 1] nx.draw(G, pos=nx.drawing.layout.spring_layout(G), with_labels=True, node_color=sc.labels_)