Как преобразовать распределение частот в распределение вероятностей в R
У меня есть матрица с n рядами наблюдений. Наблюдения - это частотные распределения характеристик. Я хотел бы преобразовать частотные распределения в вероятностные распределения, где сумма каждой строки равна 1. Поэтому каждый элемент в матрице должен быть разделен на сумму строки элемента.
Я написал следующую функцию R, которая работает, но она работает очень медленно с большими матрицами:
prob_dist <- function(x) {
row_prob_dist <- function(row) {
return (t(lapply(row, function(x,y=sum(row)) x/y)))
}
for (i in 1:nrow(x)) {
if (i==1) p_dist <- row_prob_dist(x[i,])
else p_dist <- rbind(p_dist, row_prob_dist(x[i,]))
}
return(p_dist)
}
B = matrix(c(2, 4, 3, 1, 5, 7), nrow=3, ncol=2)
B
[,1] [,2]
[1,] 2 1
[2,] 4 5
[3,] 3 7
prob_dist(B)
[,1] [,2]
[1,] 0.6666667 0.3333333
[2,] 0.4444444 0.5555556
[3,] 0.3 0.7
Не могли бы вы предложить функцию R, которая выполняет эту работу, и / или скажите мне, как я могу оптимизировать свою функцию для более быстрого выполнения?
4 ответа
Вот попытка, но на матрице данных вместо матрицы:
df <- data.frame(replicate(100,sample(1:10, 10e4, rep=TRUE)))
Я попробовал dplyr подход:
library(dplyr)
df %>% mutate(rs = rowSums(.)) %>% mutate_each(funs(. / rs), -rs) %>% select(-rs)
Вот результаты:
library(microbenchmark)
mbm = microbenchmark(
dplyr = df %>% mutate(rs = rowSums(.)) %>% mutate_each(funs(. / rs), -rs) %>% select(-rs),
t = t(t(df) / rep(rowSums(df), each=ncol(df))),
apply = t(apply(df, 1, prop.table)),
times = 100
)
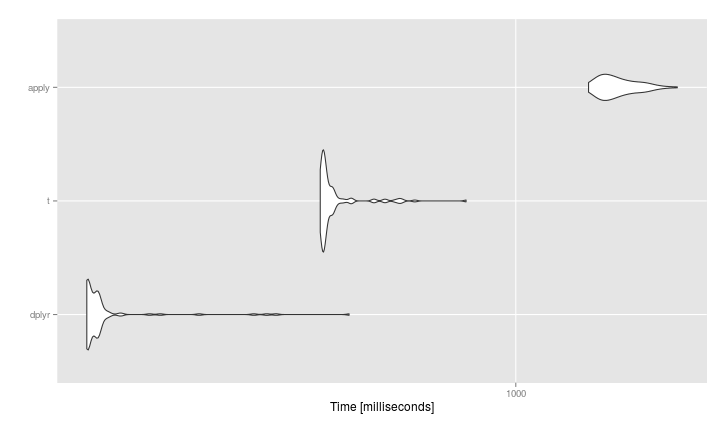
#> mbm
#Unit: milliseconds
# expr min lq mean median uq max neval
# dplyr 123.1894 124.1664 137.7076 127.3376 131.1523 445.8857 100
# t 384.6002 390.2353 415.6141 394.8121 408.6669 787.2694 100
# apply 1425.0576 1520.7925 1646.0082 1599.1109 1734.3689 2196.5003 100
Изменить: бенчмарк @David больше соответствует OP, поэтому я предлагаю вам рассмотреть его подход, если вы хотите работать с матрицами.
Без применения векторизованное решение в одну строку:
t(t(B) / rep(rowSums(B), each=ncol(B)))
[,1] [,2]
[1,] 0.6666667 0.3333333
[2,] 0.4444444 0.5555556
[3,] 0.3000000 0.7000000
Или же:
diag(1/rowSums(B)) %*% B
На самом деле, я быстро обдумал это, и лучшая заветность была бы просто
B/rowSums(B)
# [,1] [,2]
# [1,] 0.6666667 0.3333333
# [2,] 0.4444444 0.5555556
# [3,] 0.3000000 0.7000000
На самом деле, тест @Stevens вводил в заблуждение, потому что у OP есть матрица, а у Стивена - на фрейме данных.
Вот эталонный тест с матрицей. Так что для матриц оба векторизованных решения будут лучше, чем dplyr который не работает с матрицами
set.seed(123)
m <- matrix(sample(1e6), ncol = 100)
library(dplyr)
library(microbenchmark)
Res <- microbenchmark(
dplyr = as.data.frame(m) %>% mutate(rs = rowSums(.)) %>% mutate_each(funs(. / rs), -rs) %>% select(-rs),
t = t(t(m) / rep(rowSums(m), each=ncol(m))),
apply = t(apply(m, 1, prop.table)),
DA = m/rowSums(m),
times = 100
)
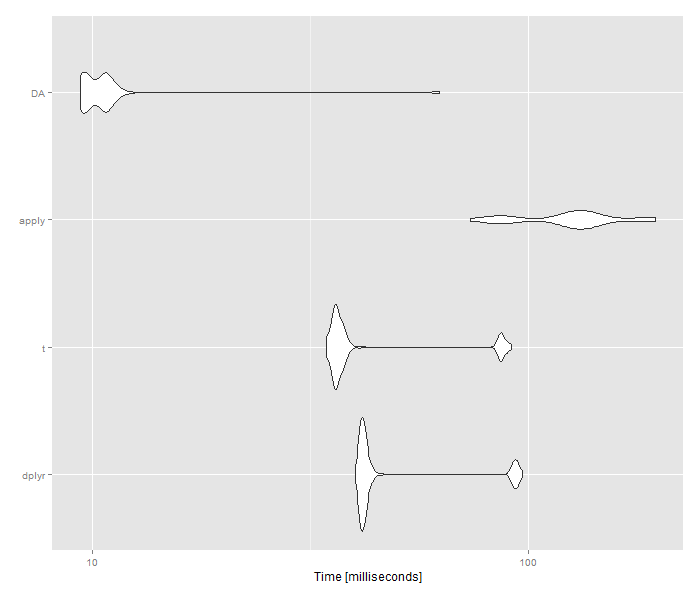
Я не уверен, что ваша функция имеет какое-либо значение, так как вы могли бы просто использовать hist или же density функции для достижения того же результата. Кроме того, использование apply будет работать, как упоминалось. Но это служит разумным примером программирования.
В вашем коде есть несколько недостатков.
- Вы используете цикл for вместо векторизации вашего кода. Это очень дорого. Вы должны использовать применить, как указано в комментариях выше.
Ты используешь
rbindвместо предварительного выделения места для вашего вывода. Это также очень дорого.out <- matrix(NA, nrow= n, ncol= ncol(B)) for (i in 1:nrow(B)) { out[i,] <- row_prob_dist(B[i,]) }