Выпуклая оболочка для троичного заговора с использованием ggtern в R
Площадь полинаторов выпуклой оболочки
Рассмотрим следующую data.frame
DGChi <- structure(list(Sucrose = c(42, 40, 15, 19, 33, 49, 35, 31, 22,
25, 37, 28, 31, 41, 27, 28, 33, 43, 21, 37, 14, 41, 30, 34, 38,
40, 40, 33, 33), Fructose = c(27, 29, 41, 35, 29, 23, 27, 33,
38, 38, 28, 31, 29, 26, 32, 34, 31, 28, 40, 30, 39, 27, 32, 31,
29, 28, 28, 32, 29), Glucose = c(31, 31, 44, 46, 38, 28, 38,
36, 40, 37, 35, 41, 40, 33, 41, 38, 36, 30, 39, 33, 47, 32, 38,
35, 33, 32, 32, 35, 38), Sindrome = c("Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily")), .Names = c("Sucrose",
"Fructose", "Glucose", "Sindrome"), row.names = c(NA, -29L), class = c("tbl_df",
"tbl", "data.frame"))
Первая попытка
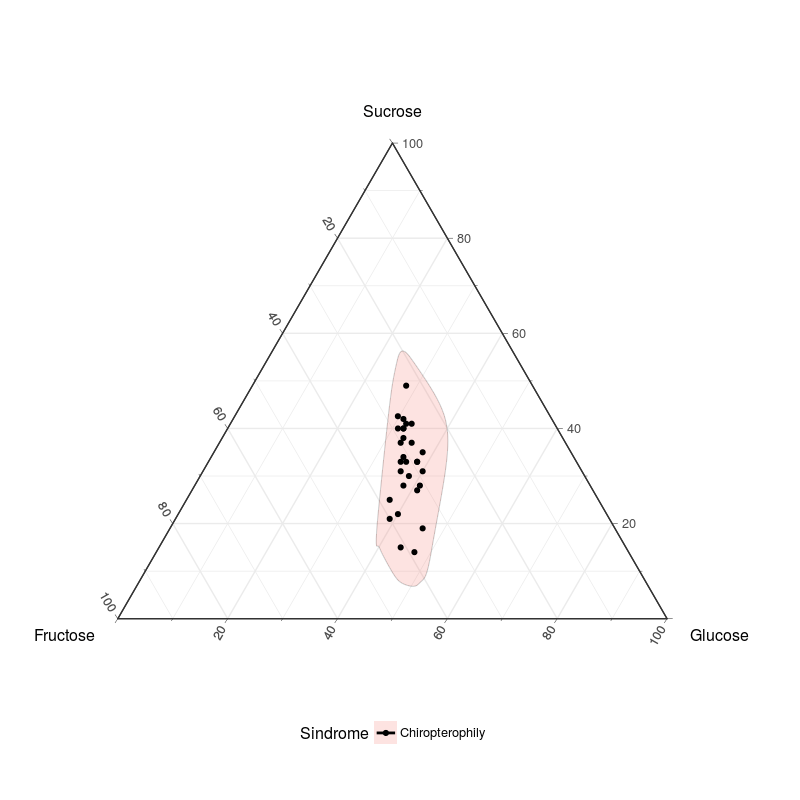
Я пытаюсь создать тройной график и добавить выпуклую оболочку вокруг точек, моей первой попыткой было использование geom_encircle из пакета ggalt:
library(ggtern)
library(ggalt)
ggtern(data = DGChi, aes(x = Fructose, y = Sucrose, z = Glucose, fill = Sindrome)) +
theme_bw() +
geom_encircle(alpha=0.2,size=1, spread = 0.5) +
geom_point() +
theme(legend.position="bottom")
С этим результатом
Который окружает точки, но не является выпуклой оболочкой
Вторая попытка с использованием пакета геометрии
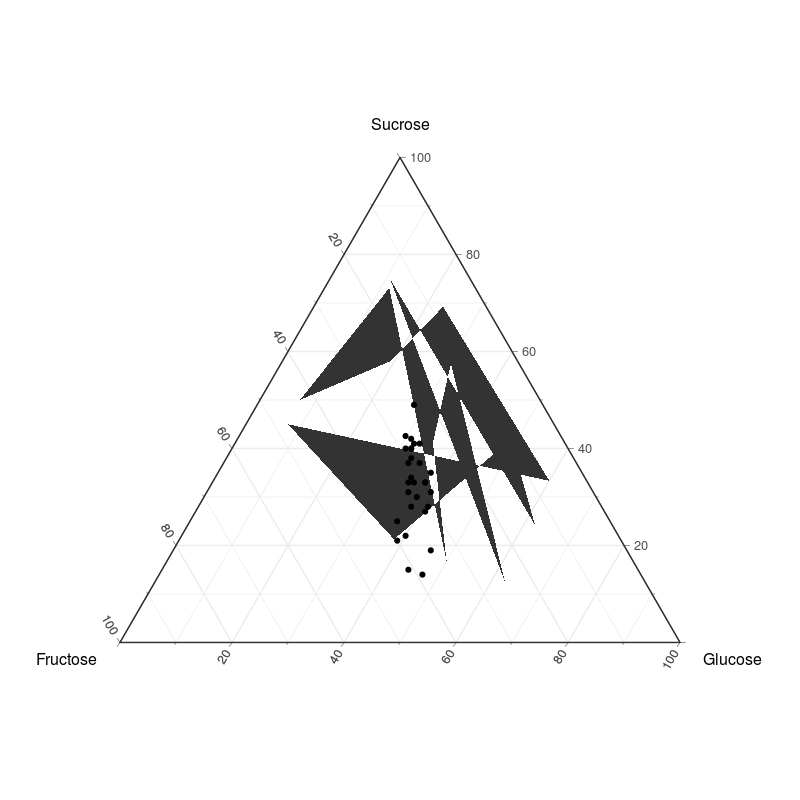
Пытаясь следовать моему собственному ответу для rgl, я попробовал это:
library(geometry)
DGChiMin <- as.data.frame(convhulln(matrix(c(DGChi$Fructose, DGChi$Sucrose, DGChi$Glucose), ncol = 3)))
colnames(DGChiMin) <- c("Fructose", "Sucrose", "Glucose")
и тогда это для сюжета:
ggtern(data = DGChi, aes(x = Fructose, y = Sucrose, z = Glucose)) +
theme_bw() +
geom_polygon(data = DGChiMin,aes(x = Fructose, y = Sucrose, z = Glucose)) +
geom_point() +
theme(legend.position="bottom")
Но получил этот супер странный многоугольник:
Может ли кто-нибудь помочь мне получить выпуклый корпус?
2 ответа
Решение
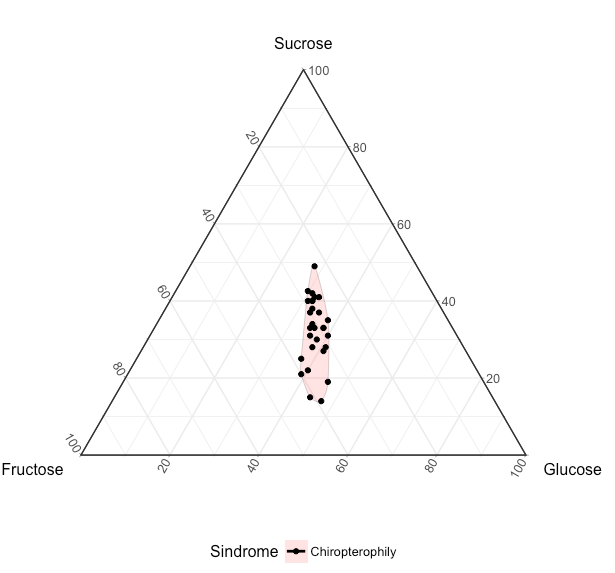
Думаю, вы обнаружите, что geom_encircle использует chull внутри. Установите параметр расширения на 0.
library(ggalt)
library(ggtern)
ggtern(data = DGChi, aes(x = Fructose, y = Sucrose, z = Glucose, fill = Sindrome)) +
theme_bw() +
geom_encircle(alpha=0.2,size=1, expand=0) + ##<<<<<< expand = 0
geom_point() +
theme(legend.position="bottom")
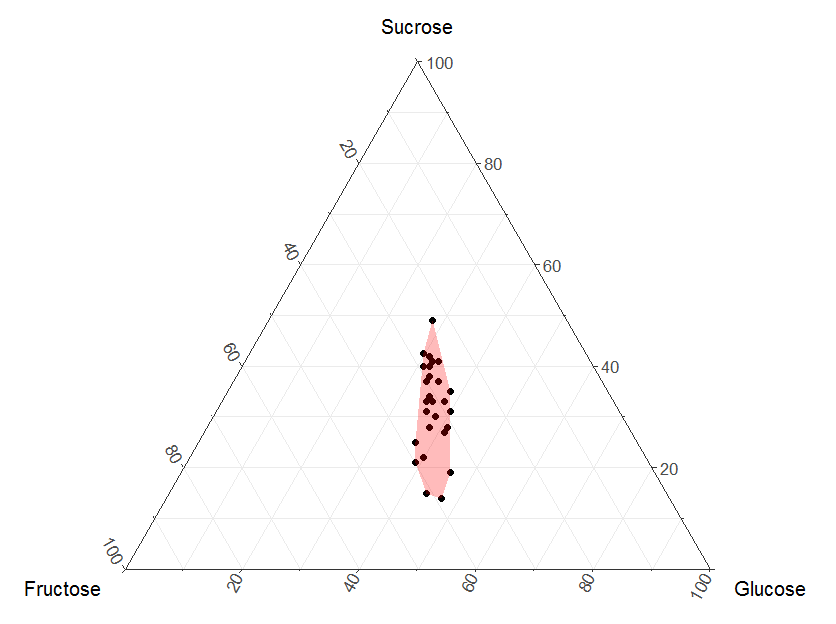
Вот простое решение, основанное на chull функция.
DGChi <- structure(list(Sucrose = c(42, 40, 15, 19, 33, 49, 35, 31, 22,
25, 37, 28, 31, 41, 27, 28, 33, 43, 21, 37, 14, 41, 30, 34, 38,
40, 40, 33, 33), Fructose = c(27, 29, 41, 35, 29, 23, 27, 33,
38, 38, 28, 31, 29, 26, 32, 34, 31, 28, 40, 30, 39, 27, 32, 31,
29, 28, 28, 32, 29), Glucose = c(31, 31, 44, 46, 38, 28, 38,
36, 40, 37, 35, 41, 40, 33, 41, 38, 36, 30, 39, 33, 47, 32, 38,
35, 33, 32, 32, 35, 38), Sindrome = c("Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily", "Chiropterophily",
"Chiropterophily", "Chiropterophily", "Chiropterophily")), .Names = c("Sucrose",
"Fructose", "Glucose", "Sindrome"), row.names = c(NA, -29L), class = c("tbl_df",
"tbl", "data.frame"))
# Convex hull
ch <- chull(DGChi[,1:2])
DGChiMin <- DGChi[ch, 1:3]
library(ggtern)
ggtern(data = DGChi, aes(x = Fructose, y = Sucrose, z = Glucose)) + geom_point() +
theme_bw() +
geom_polygon(data = DGChiMin, aes(x = Fructose, y = Sucrose, z = Glucose), fill="#FF000044") +
theme(legend.position="bottom")