Представление на одном графике двух графиков в разных осях с использованием ggplot
Давайте воспроизведем пример, с которым мы работаем:
chol <- read.table(url("http://assets.datacamp.com/blog_assets/chol.txt"), header = TRUE)
Теперь мы представляем две гистограммы ggplot:
library(ggplot2)
plot1 <- ggplot(data=chol, aes(chol$AGE)) +
geom_histogram(breaks=seq(20, 50, by = 2),
col="red",
fill="green",
alpha = .2) +
labs(title="Histogram for Age") +
labs(x="Age", y="Count") +
xlim(c(18,52)) +
ylim(c(0,100))
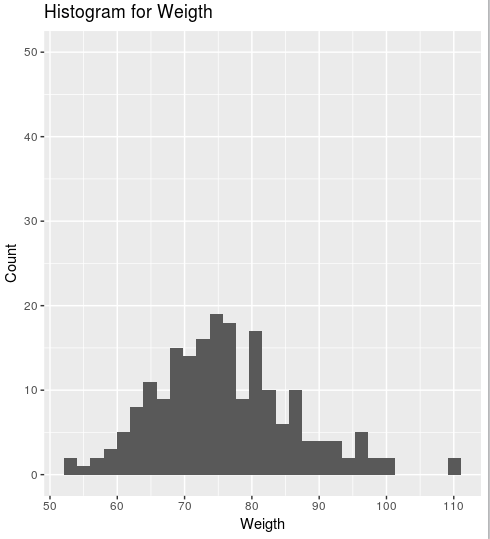
plot2 <- ggplot(data=chol, aes(WEIGHT)) +
geom_histogram() +
labs(title="Histogram for Weigth") +
labs(x="Weigth", y="Count") +
ylim(0,50)
Это две гистограммы, первые plot1 и второй plot2, 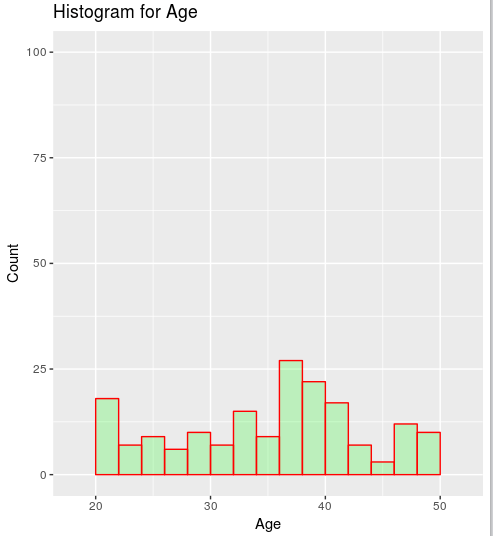
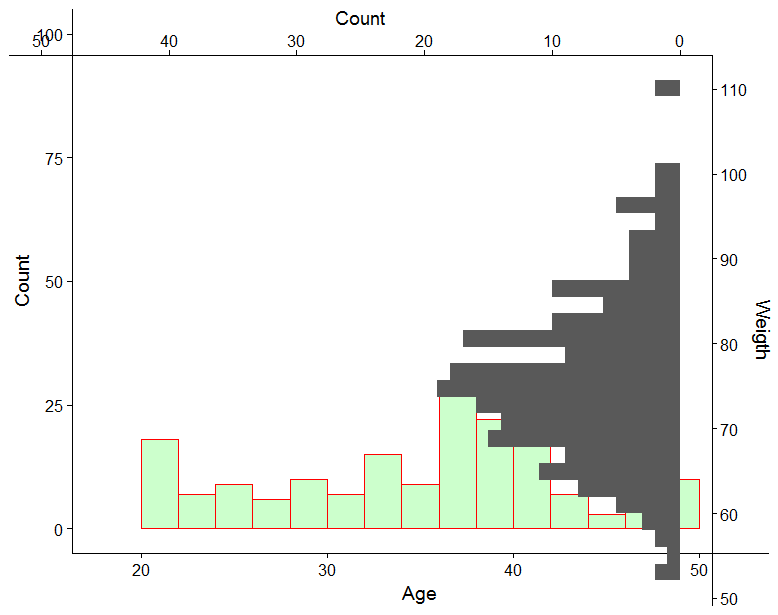
Я хотел бы объединить их обоих, представляя plot1 по оси X и plot2 по оси Y нового сюжета. Результат может быть похож на это: 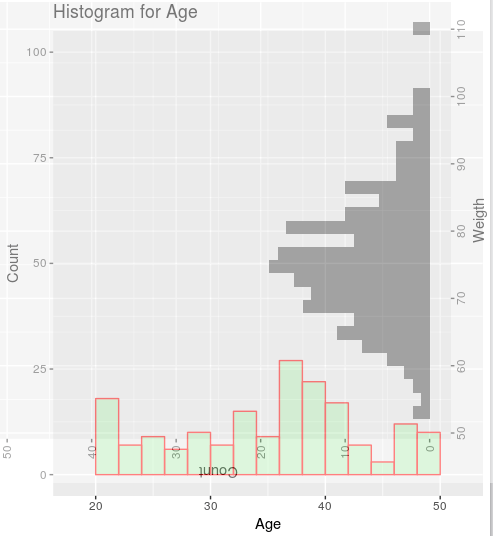
Как я могу достичь этой цели?
2 ответа
Вы можете попробовать следующее. Хотя я не понимаю смысла такого перплотинга.
plot1 <- ggplot(data=chol, aes(AGE)) +
geom_histogram(breaks=seq(20, 50, by = 2),
col="red",
fill="green",
alpha = .2) +
labs(x="Age", y="Count") +
xlim(c(18,52)) +
ylim(c(0,100))
plot2 <- ggplot(data=chol, aes(WEIGHT)) +
geom_histogram() +
labs(x="Weigth", y="Count") +
scale_x_continuous(position = "top")+
scale_y_reverse(position = "right",limits = c(50,0)) +
coord_flip()
library(cowplot)
ggdraw(plot1) +
draw_plot(plot2)
Я использую код следующего блога и адаптировался к вашему примеру. У вас есть обе гистограммы на одном графике, а также график рассеяния. Но вместо этого они находятся в обратном направлении.
http://sas-and-r.blogspot.com/2011/06/example-841-scatterplot-with-marginal.html
scatterhist = function(x, y, xlab="", ylab=""){
zones=matrix(c(2,0,1,3), ncol=2, byrow=TRUE)
layout(zones, widths=c(4/5,1/5), heights=c(1/5,4/5))
xhist = hist(x, plot=FALSE)
yhist = hist(y, plot=FALSE)
top = max(c(xhist$counts, yhist$counts))
par(mar=c(3,3,1,1))
plot(x,y)
par(mar=c(0,3,1,1))
barplot(xhist$counts, axes=FALSE, ylim=c(0, top), space=0)
par(mar=c(3,0,1,1))
barplot(yhist$counts, axes=FALSE, xlim=c(0, top), space=0, horiz=TRUE)
par(oma=c(3,3,0,0))
mtext(xlab, side=1, line=1, outer=TRUE, adj=0,
at=.8 * (mean(x) - min(x))/(max(x)-min(x)))
mtext(ylab, side=2, line=1, outer=TRUE, adj=0,
at=(.8 * (mean(y) - min(y))/(max(y) - min(y))))
}
#with your code#
with(chol, scatterhist(chol$AGE, chol$WEIGHT, xlab="AGE", ylab="WEIHGT"))