График накопленной смертности в R
Мне нужно получить график, который имеет среднюю совокупную смертность для двух популяций устриц при 4 разных соленостях.
Date = c("5/29/2017","5/31/2017","6/2/2017", "5/29/2017","5/31/2017","6/2/2017","5/29/2017","5/31/2017","6/2/2017","5/29/2017","5/31/2017","6/2/2017","5/29/2017","5/31/2017 ","6/2/2017","5/29/2017 ","5/31/2017","6/2/2017", "5/29/2017","5/31/2017","6/2/2017","5/29/2017", "5/31/2017","6/2/2017")
Mortality = c(0, 0.4, 0.5, 0.1, 0.3, 0.4, 0.2, 0.3, 0.5, 0.1, 0.2, 0.6, 0, 0.7, 0.8, 0.1, 0.6, 0.9, 0.1, 0.5, 0.9, 0.1, 0.6, 0.8)
Tank = c("1A", "1A", "1A", "1B", "1B", "1B", "5A", "5A", "5A", "5B", "5B", "5B", "1A", "1A", "1A", "1B", "1B", "1B", "5A", "5A", "5A", "5B", "5B", "5B")
Population = c("SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC") = c (SL SL SL SL SL SL SL SL SL SL SL SL LC LC LC LC LC LC LC LC LC LC LC LC)
Мне нужно усреднить два значения (например, 1A и 1B) и затем построить это значение для даты. Мне бы хотелось, чтобы только даты, когда совокупная смертность была рассчитана, были показаны на оси х.
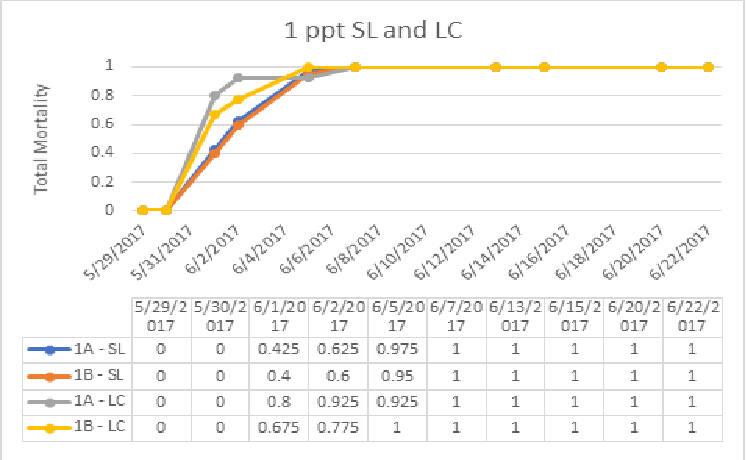
У меня есть следующий график, который я сделал в Excel, но это не совсем то, что мне нужно.
1 ответ
Вот мое решение с использованием ggplot2 а также dplyr
Вы можете использовать другие пакеты для обобщения ваших данных перед построением графика. Здесь я сделал две группы (1 = 1A и 1B, 2 = другие танки).
Просто из любопытства, почему вы не используете графики Капплана-Мейера для этих данных?
Date = c("5/29/2017","5/31/2017","6/2/2017", "5/29/2017","5/31/2017","6/2/2017","5/29/2017","5/31/2017","6/2/2017","5/29/2017","5/31/2017","6/2/2017","5/29/2017","5/31/2017 ","6/2/2017","5/29/2017 ","5/31/2017","6/2/2017", "5/29/2017","5/31/2017","6/2/2017","5/29/2017", "5/31/2017","6/2/2017")
Mortality = c(0, 0.4, 0.5, 0.1, 0.3, 0.4, 0.2, 0.3, 0.5, 0.1, 0.2, 0.6, 0, 0.7, 0.8, 0.1, 0.6, 0.9, 0.1, 0.5, 0.9, 0.1, 0.6, 0.8)
Tank = c("1A", "1A", "1A", "1B", "1B", "1B", "5A", "5A", "5A", "5B", "5B", "5B", "1A", "1A", "1A", "1B", "1B", "1B", "5A", "5A", "5A", "5B", "5B", "5B")
Population = c("SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "SL", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC", "LC")
df <- data.frame(Date, Mortality, Tank, Population)
library(dplyr)
library(ggplot2)
dfs <- df %>% mutate(TankGroup = if_else(Tank %in% c('1A','1B'),1,2)) %>%
group_by(Date, TankGroup) %>%
summarise(Mortality = mean(Mortality))
ggplot(dfs, aes(x = Date, y = Mortality, group = factor(TankGroup), color = factor(TankGroup))) +
geom_point() + geom_line() + theme_bw()